CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
弗尼斯弧菌PCR检测方法的建立及应用
作者:麻丽丹1田卓2齐欣2
麻丽丹1田卓2齐欣2
摘要目的:弗尼斯弧菌(Vibrio furnissii)是引起世界范围内胃肠功能紊乱的致病菌之一,一般通过摄入生的或者未煮熟的水产品进行传播。本研究旨在建立高效、可靠的PCR检测方法应用于食品中弗尼斯弧菌的快速检测。方法和结果:根据弗尼斯弧菌的toxS基因分别设计普通PCR引物及实时PCR引物、探针,建立普通PCR和实时荧光PCR方法,分别对两种方法的特异性和灵敏度进行比较分析,同时应用于384份食品样品检测。结果表明,建立的普通PCR和实时荧光PCR方法特异性良好,检测灵敏度分别为:2.4×103 CFU/ml和24 CFU/ml,并于5 h内完成整个检测过程,同时对384份食品进行检测,共检出6株阳性菌株。结论:本研究建立的PCR检测技术灵敏度高、特异性好,可应用于食品中弗尼斯弧菌的快速检测。
关键词 弗尼斯弧菌;普通PCR;实时荧光PCR;toxS基因
Establishment and Application of a PCR Technique BasedMethod for Detection of Vibrio Furnissii
MA Li-Dan1TIAN Zhuo2QI Xin2
AbstractAims: Vibrio furnissii is a cause of human gastrointestinal disorders worldwide, could transmit by ingestion of raw or undercooked contaminated seafood. The aim of this work was to develop an effective and reliable method for the identification of V. furnissii. Methods and Results: Conventional PCR and real-time fluorescence PCR techniques were applied to the rapid detection of V. furnissii by designing primers and probe target for the toxS gene. The specificity and sensitivity of the method were investigated, and the practical application was analyzed using 384 commercial food products. The results showed that a high detection specificity of the developed method. The detection limit of the conventional PCR and real-time fluorescence PCR was 2.4×103 CFU/ml and 24 CFU/ml, respectively, and the entire identification could be completed within 5 h. In addition, the technique enabled the accurate recognition of V. furnissii from food products (6 positive products). Conclusions: A specific, sensitive and rapid PCR method target for V. furnissii toxS gene was established and could be applied to the bacterial detection in food products.
Keywords Vibrio furnissii; conventional PCR; real-time fluorescence PCR; toxS gene
弗尼斯弧菌原名为产气河弧菌(Vibrio fluvialis),1983年被Brenner重新划分为弧菌属的一个新种。如同副溶血弧菌一样,弗尼斯弧菌也可引起人类腹泻、腹痛、恶心、呕吐等急性胃肠炎的发生[1-2]。弗尼斯弧菌分布广泛,不仅可以从腹泻病人粪便中分离得到,也可从水产环境中分离得到[3-5]。研究表明,弗尼斯弧菌可引起欧洲鳗鱼、虾、贝类等水产品的污染,而摄入被污染的生的或未煮熟的水产品会导致人类食源性感染性腹泻[6-11]。另外,弗尼斯弧菌也可引起人类浅表创伤感染、菌血症等[12]。
ToxS基因是首次发现于霍乱弧菌中的毒力基因,位于toxR基因(作为霍乱弧菌毒素操纵子的转率激活子)的下游以增强toxR基因的调节转录活性[13]。toxS基因除了存在于霍乱弧菌中外,还存在于副溶血弧菌、费歇尔弧菌、创伤弧菌和弗尼斯弧菌等弧菌中[14-19]。目前,toxS基因作为一个靶基因进行弧菌属的PCR检测已得到广泛应用,但是针对弗尼斯弧菌的PCR检测尚未进行研究。因此本研究以toxS基因为靶基因,建立高效、特异的PCR检测方法,用于食品中弗尼斯弧菌的快速检测。
1 材料与方法
1.1 菌株
用于本研究的菌株共82株,其中包括弗尼斯弧菌标准菌株(ATCC 35016、IQCC 12301、IQCC 12309)3株及其分离株3株、溶藻弧菌标准菌株(ATCC 17749)1株及其分离株13株、霍乱弧菌分离株9株、副溶血弧菌标准菌株(ATCC 17802)1株及其分离株20株、麦氏弧菌标准菌株(ATCC 700040)1株及其分离株1株、河弧菌标准菌株(CICC 21612)1株及其分离株10株、拟态弧菌标准菌株(CICC 21613、IQCC 12304、IQCC 12318)3株及其分离株5株、创伤弧菌标准菌株(ATCC27652)1株、哈氏弧菌标准菌株(ATCC35084)1株、霍利斯弧菌标准菌株(ATCC33564)1株、坎氏弧菌分离株1株、大肠杆菌标准菌株(ATCC 25922)1株及其分离株3株、嗜水气单孢菌分离株2株、志贺氏菌标准菌株(ATCC 12022)1株等。
本实验采用的标准菌株购自于ATCC、CICC、IQCC,其余菌株来自于本单位在实测样品中分离的菌株和来自北京、上海、广东、辽宁等海关的样品分离株及疾病预防控制中心的临床分离菌株。所有菌株都经生理生化实验和测序进行验证。
1.2 材料
江瑶贝、蚬子肉、冻鸡肉及冻花菜等共384份样品为丹东海关进出口样品及丹东市市场监测样品。
1.3 仪器与试剂
ABI Veriti PCR仪、ABI 7300型实时荧光定量PCR系统、ABI 3500/3500xL基因测序仪 美国应用生物系统公司;3-18K型台式高速冷冻离心机 德国Sigma公司。
Path-IDTM qPCR Master mix Kit(货号4388643) 美国应用生物系统公司;TaKaRa Ex Taq(货号:RR001A)、TaKaRa MiniBEST Bacteria Genomic DNA Extraction Kit Ver.3.0(货号:9763) 宝生物工程(大连)有限公司。
1.4 方法
1.4.1 DNA提取
按照宝生物MiniBEST Bacteria Genomic DNA Extraction Kit Ver.3.0试剂盒操作说明进行DNA提取。
1.4.2 引物及探针
弗尼斯弧菌toxS基因的普通PCR引物及实时荧光PCR引物和探针序列见表1,由Invitrogen公司合成。
1.4.3 普通PCR方法
反应体系:模板液2μL、10×PCR缓冲液2.5 μL、dNTP 0.25μL、Taq酶0.125μL、正反向引物(10μM)各0.5μL、补充灭菌双蒸水至总体积为25 μL;以上各组分加入至0.2mL PCR反应管中,混匀,5000 r/min 离心10s;
反应条件:94℃预变性4min,然后94℃1min、59℃1min、72℃1min,35个循环,最后72℃延伸10min,4℃保存。
1.4.4 实时荧光PCR方法
反应体系:模板液2μL、qPCR Maser mix12.5 μL、正向引物(μM)0.5μL、反向引物(10μM)0.5μL、补充灭菌双蒸水至总体积为25μL;以上各组分加入至0.2 mL PCR反应管中,混匀,5000 r/min 离心10s;
反应条件:94℃预变性10min,然后94℃15 s、60℃1min,35个循环,每次循环的退火时收集荧光。
1.4.5 特异性实验
选取弗尼斯弧菌菌株6株、溶藻弧菌、霍乱弧菌、副溶血弧菌、麦氏弧菌等弧菌属和肠杆菌科的菌株(共76株),分别按照普通PCR方法和实时荧光PCR方法进行扩增,以检验两种方法的特异性。
1.4.6 灵敏度实验
将弗尼斯弧菌标准菌株于3%氯化钠碱性蛋白胨水中培养18 h左右后,测其麦氏浊度,估计其菌数,然后按十倍递增进行梯度稀释到10-7,取最后4个稀释液的菌液进行平板计数,每个稀释度重复3个,按照相应的方法培养后进行菌落计数,取平均值确定其真实的菌密度。同时每个稀释液各取1mL菌液于2 mL离心管中,并重复取3管,用于提取DNA,随后分别采用普通PCR方法和实时荧光PCR方法进行检测,以确定两种方法的检测灵敏度。
1.4.7 应用于实际样品检测
以传统的检测方法(ISO/TS 21872-2[20])为对照方法,对江瑶贝、蚬子肉、冻鸡肉及冻花菜等共384份样品分别进行普通PCR和实时荧光PCR检测,以检验本方法的效果。
2 结果
2.1 特异性试验结果
特异性实验结果显示,普通PCR方法(见图1)中的6株弗尼斯弧菌均可扩增出150 bp大小的特异性片断,实时荧光PCR方法(图2)中的6株弗尼斯弧菌均可扩增出典型的扩增曲线,而采用两种PCR方法对其余弧菌及肠杆菌的扩增呈阴性,表明两种PCR方法的特异性均为100 %。
图2 实时荧光PCR检测方法特异性试验结果
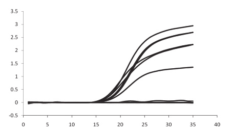
1: 弗尼斯弧菌标准菌株 (ATCC 35016);2:弗尼斯标准菌株(IQCC 12301);3:弗尼斯标准菌株(IQCC 12309):4:弗尼斯弧菌分离株(病人);5:弗尼斯弧菌分离株(蚬子肉);6:弗尼斯弧菌分离株(病人)
2.2 灵敏度试验结果
弗尼斯弧菌RNA梯度稀释检测结果(图3和图4)显示,普通PCR方法和实时荧光PCR检测方法对于弗尼斯弧菌的检测灵敏度分别为2400 CFU/mL和24 CFU/mL,表明实时荧光PCR检测方法的灵敏度是普通PCR检测方法的100倍。
图3 普通PCR灵敏度试验结果

1、17:DNA Marker;2、3:2.4×108 CFU/mL;4、5:2.4×107 CFU/mL;6、7:2.4×106 CFU/mL;8、9:2.4×105 CFU/mL;10、11:2.4×104 CFU/mL;12、13:2.4×103 CFU/mL;14、15:2.4×102 CFU/mL;16:阴性对照
图4 实时荧光PCR灵敏度试验结果

1:2.4×106 CFU/mL;2:2.4×105 CFU/mL;3:2.4×104 CFU/mL ;4:2.4×103 CFU/mL;5: 2.4×102 CFU/mL;6: 2.4×101 CFU/mL
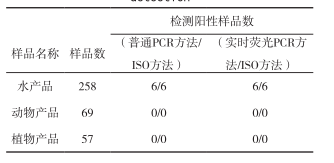
2.3 食品样品检测结果
对384份样品的检测结果如表2:在258份水产品中,检出弗尼斯弧菌阳性6份样品,这6份样品分别来自为:牡蛎(1例)、杂色蛤(2例)、虾(1例)、章鱼(1例)、腌蟹子(1例)样品。其余126份食品中均未检出弗尼斯弧菌,且所有检测结果与传统生化培养方法检测结果一致,6株弧菌测序结果与Genbank中登录号为:CP002377.1的弗尼斯弧菌一致性为100 %,证明本研究所建立两种PCR方法适用于食品中弗尼斯弧菌的检测。
表2 食品样品检测结果
3 讨论
传统的培养法对于致病性弧菌的鉴定不仅费时,而且操作复杂,不适用于样本的高通量检测。近年来,随着分子生物学的不断发展,PCR技术的应用大大改善了致病性弧菌检测的准确性和速度[21-28]。本研究建立了基于PCR技术的弗尼斯弧菌快速检测方法,结果表明,普通PCR检测方法和实时荧光PCR检测方法的引物和探针具有良好的特异性(100 %),两种方法的灵敏度分别为2400CFU/mL和24CFU/mL,实时荧光PCR检测方法的灵敏度是普通PCR检测方法的100倍。检测方法简单快速,可在5h内完成整个检测过程。采用本研究建立的方法对384份水产品样品的检测结果显示,共有6份样品检出弗尼斯弧菌阳性,其检测结果与ISO方法检测结果及测序结果一致,表明该方法适用于食品中弗尼斯弧菌的检测。
目前,已有关于从虾[9]、鱿鱼[28]、牡蛎[24]等水产品中检测出弗尼斯弧菌的报道,与本研究的数据保持一致。研究结果也表明,弗尼斯弧菌引起的污染已成为食品安全的一个重要隐患,因此,建立基于PCR技术的高度特异性方法对于食品中弗尼斯弧菌的快速检测具有重要意义。
表1 弗尼斯弧菌普通PCR引物及实时荧光PCR引物和探针序列

图1 普通PCR检测方法特异性试验结果

1、20:DNA Marker;2:弗尼斯弧菌标准菌株 (ATCC 35016);3:弗尼斯标准菌株(IQCC 12301);4:弗尼斯标准菌株(IQCC 12309);5: 弗尼斯弧菌分离株(病人);6:弗尼斯弧菌分离株(蚬子肉)7:弗尼斯弧菌分离株(病人);8:麦氏弧菌标准菌株(ATCC 700040);9:河弧菌标准菌株(CICC 21612);10:拟态弧菌标准菌株(CICC 21613);11:拟态弧菌标准菌株(:IQCC 12304);12:创伤弧菌标准菌株(ATCC27652);13: 哈氏弧菌标准菌株(ATCC35084);14:霍利斯弧菌标准菌株(ATCC33564);15:坎氏弧菌分离株(毛蚬);16:大肠杆菌标准菌株(ATCC 25922);17:副溶血弧菌标准菌株(ATCC 17802);18:志贺氏菌标准菌株(ATCC 12022);19:阴性对照。
参考文献
[1] DANIELS N A, SHAFAIE A. A review of pathogenic Vibrio infections for clinicians[J]. Infections In Medicine, 2000, 17(10): 665-685.
[2] SCHIRMEISTER F, WIECZOREK A, DIECKMANN R, et al. Evaluation of molecular methods to discriminate the closely related species Vibrio fluvialis and Vibrio furnissii[J]. International Journal of Medical Microbiology, 2014,304(7):851-857.
[3] DALSGAARD A, BERNAL M, SHIMADA T, et al . Vibrio furnissii isolated from humans in Peru: a possible human pathogen?[J]. Epidemiology Infection, 1997,119(2): 143-149.
[4]HERNÁNDEZ-LÓPEZ J, GOLLAS-GALVÁN T, MAGALLÓN-BARAJAS F, et al. Isolation of Vibrio and Pseudomonas from brown shrimp ,Penaeus Californiensis Holmes. Intestine[J]. Revista Latinoamericana Microbiologia, 1997, 39[3-4]: 109-115 .
[5] DERBER C, COUDRON P, TARR C, et al. Vibrio furnissii: an unusual cause of bacteremia and skin lesions after ingestion of seafood[J]. Journal of Clinical Microbiology, 2011. 49(6): 2348-2349.
[6] SUNG H H, HSU S F, CHEN C K, et al. Relationships between disease outbreak in cultured tiger shrimp ,Penaeus monodon. and the composition of Vibrio communities in pond water and shrimp hepatopancreas during cultivation[J]. Aquaculture, 2001,192(2): 101-110.
[7] MIAO L, QIAN P Y. Antagonistic antimicrobial activity of marine fungi and bacteria isolated from marine biofilm and seawaters of Hong Kong[J]. Aquatic Microbial Ecology, 2005, 38(3): 231-238.
[8] ESTEVE C, AMARO C, BIOSCA E G,et al. Biochemical and toxigenic properties of Vibrio furnissii isolated from a European eel farm[J]. Aquaculture, 1995, 132(1-2): 81-90.
[9] JAYASINGHE C, AHMED S, KARIYAWASAM M. The isolation and identification of Vibrio species in marine shrimps of Sri Lanka[J]. Journal of Food Agriculture, 2008, 1(1): 36-44.
[10] MATTE G R, MATTE M H, RIVERA I G, et al. Distribution of potentially vibrios in oysters from a tropical region[J]. Journal of Food Protection ,1994, 57(57): 870-873.
[11] JANDA J M, POWERS C, BRYANT R G, et al. Current perspectives on the epidemiology and pathogenesis of clinically significant Vibrio spp[J]. Clinical Microbiology Reviews, 1988, 1(3): 245-267.
[12] 徐嘉良, 惠书瑶, 卢 昕, 等. 利用比较基因组学研究弗尼斯弧菌和霍乱弧菌在鸟氨酸脱羧酶试验中不同表型的分子遗传基础[J]. 食品科技, 2015,40(8):301-304.
[13] ILLER V L, DIRITA V J, MEKALANOS J J. Identification of toxS, a regulatory gene whose product enhances toxR-mediated activation of the cholera toxin promoter[J]. Journal of Bacteriology, 1989, 171(3): 1288-1293.
[14] LI X J, ZHAO H, SU J, et al. A method for screening and detection of virulence genes of Vibrio isolates from freshwater crayfish[J]. Food Science, 2011, 32(18): 264-267.
[15] REICH K A, SCHOOLNIK G K. The light organ symbiont Vibrio fischeri possesses a homolog of the Vibrio cholerae transmembrane transcriptional activator ToxR[J]. Journal of Bacteriology, 1994, 176(10): 3085-3088.
[16] LEE S E, SHIN S H, KIM S Y, et al. Vibrio vulnificus has the transmembrane transcription activator ToxRS stimulating the expression of the hemolysin gene vvhA[J]. Journal of Bacteriology, 2000, 182(12): 3405-3415.
[17] LUX T M, LEE R, LOVE J. Complete genome sequence of a free-living Vibrio furnissii sp nov strain ,NCTC 11218[J]. Journal of Bacteriology, 2011, 193(6): 1487-1488.
[18] FETHI B A, KAMEL C, HELA K, et al. RT-PCR assays for in vivo expression of Vibrio alginolyticus virulence genes in cultured gilthead Dicentrarchus labrax and Sparus aurata[J]. Annals of Microbiology, 2009, 59(1): 63-67.
[19] KHOUADJA S, SUFFREDINI E, BACCOUCHE B, et al. Occurrence of virulence genes among Vibrio cholerae and Vibrio parahaemolyticus strains from treated wastewaters[J]. Environmental Monitoring Assessment, 2014, 186(10): 6935-6945 .
[20] INTERNATIONAL ORGANIZATION FOR STANDARDS. ISO/TS 21872-1 Microbiology of food and animal feed stuffs&—Horizontal method for the detection of potentionally enteropathogenetic vibrio spp.&—Part 1: Detection of Vibrio parahaemolyticus and Vibrio cholera [S]. Switzerland: International Organization for Standardization, 2007.
[21] PONTES D S, LIMA-BITTENCOURT C I, CHARTONE-SOUZA E, et al. Molecular approaches: advantages and artifacts in assessing bacterial diversity[J]. Journal of Industrial Microbiology Biotechnolgy, 2007, 34(7): 463-473.
[22] CARBONNELLE E, MESQUITA C, BILLE E, et al. MALDI-TOF mass spectrometry tools for bacterial identification in clinical microbiology laboratory[J]. Biotribune magazine, 2011, 44(1): 104-109.
[23] 李丹丹, 徐义刚, 王昱, 等. 副溶血弧菌实时荧光定量PCR快速检测方法的建立[J]. 中国畜牧兽医, 2016, 43(1):58-62..
[24] BAKR W M, HAZZAH W A, ABAZA A F. Detection of Salmonella and Vibrio species in some seafood in Alexandria[J]. Journal of American Science, 2011, 7(9): 663-668.
[25] SENACHAI P, CHOMVARIN C , NAMWAT W, et al. Application of tetraplex PCR for detection of Vibrio cholerae,V.parahaemolyticus,V. vulnificus and V. mimicus in cockle[J]. Southeast Asian Journal of Tropical Medicine puplic Health, 2013, 44(2):249-258.
[26] 胡兴娟, 沈飚, 余辉, 等. 多重荧光定量PCR法检测水产品中副溶血性弧菌、沙门菌和单增李斯特菌[J].中国食品卫生杂志, 2016, 28(4):440-444.
[27] MCCLEARY S, HENSHILWOOD K. Novel quantitative TaqMan MGB real-time PCR for sensitive detection of Vibrio aestuarianus in Crassostrea gigas[J]. Diseases of Aquatic Organisms, 2015, 114(3):239-248.
[28] SMITHA N R, JAMES L, IYER T S G, et al. Quantitative studies on halophilic pathogenic vibrios in fish and fish products[J]. Society of Fisheries Technologists Cochin, 2011. 48(1): 75-80.







