CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
基于叶绿体DNA的籼-粳稻谷(米)SYBR-Green实时荧光PCR鉴定方法的建立
作者:陈文炳1 邵碧英1 林阳武1 缪婷玉1 彭 娟1 郭 菁1 陈 彬1 彭华毅1
陈文炳1 邵碧英1 林阳武1 缪婷玉1 彭 娟1 郭 菁1 陈 彬1 彭华毅1
摘 要 亚洲栽培稻分为籼稻(hsien或indica)和粳稻(keng或japonica)两个亚种。基于籼-粳稻叶绿体DNA(cpDNA)的PstⅠ-12片段ORF100区域的序列差异,即籼稻cpDNA的ORF100区域缺失69 bp的碱基片段,建立籼-粳稻亚种鉴定方法。本文选择典型的籼稻谷(米)样品(台籼11号)和粳稻谷(米)(东北珍珠大米)进行叶绿体DNA(cpDNA) PstⅠ-12片段的ORF100区域DNA的 SYBR-Green实时荧光PCR扩增和测序,在籼-粳稻2个PCR产物序列比对的基础上,设计正向引物4条(F1-F4)、反向引物3条(R1-R3),组合成12对引物进行优化筛选,选出F4R1引物对。在粳稻样品中,检出cpDNA PCR产物片段长度为204 bp(非缺失型),荧光PCR扩增熔解温度Tm值为78.00;在籼稻样品中,检出cpDNA PCR产物片段长度为135 bp(cpDNA 缺失型),荧光PCR扩增熔解温度Tm值为76.50。建立了籼-粳稻亚种SYBR-Green实时荧光PCR鉴定方法。基于上述实验结果,选择国内多个省份的典型籼/粳稻品种与籼/粳型杂交稻组合共538个,对该籼粳稻米亚种鉴定方法进行验证。在177个粳稻及粳型杂交组合样品中,cpDNA荧光PCR熔解温度Tm值为78.00的样品170个,与粳稻符合率为96.05%,Tm值为76.50的7个样品,不符合率为3. 95%;在361个籼稻及籼型杂交稻样品中, Tm值为76.50的样品331个,与籼稻符合率为91.69%,Tm值为78.00的30个样品,不符合率为8.31%。本方法只检测一个遗传稳定的细胞质叶绿体DNA位点,用于籼-粳稻米的亚种鉴定,简便高效,准确率高。
关键词 籼稻;粳稻;叶绿体DNA;SYBR-Green荧光PCR;鉴定
Identification Method of Hsien and Keng Rice by SYBR-Green Fluorescent-PCR Detection for Chloroplast DNA
CHEN Wen-Bing1 SHAO Bi-Ying1 LIN Yang-Wu1 MIAO Ting-Yu1
PENG Juan1 GUO Jing1 CHEN Bin1 PENG Hua-Yi1
Abstract Asia cultivated rice (Oryza sativa L.) is divided into two subspecies, namely hsien rice (also known as Oryza sativa L. subs. indica rice) and keng rice(also known as Oryza sativa L. subs. japonica rice). Based on the sequence difference of ORF100 region within the PstⅠ-12 fragments of chloroplast DNA(cpDNA) of hsien-keng rice, it was discovered that there was a 69bps deletion in the ORF100 region of cpDNA in hsien (indica) rice. Typical hsien rice samples (Taixian 11) and keng rice (Northeast Pearl Rice) were used to amplify and sequence for ORF100 region nucleotides within the cpDNA Pst Ⅰ-12 fragment by SYBR-Green real-time PCR. Based on the comparison of two PCR product sequences of hsien and keng rice, four forward primers (F1-F4) and three reverse primers(R1-R3) were designed, and 12 pairs of primers were synthesized, a SYBR-Green real-time PCR method was developed for the identification of hsien-keng subspecies. In hsien subspecies, the length of cpDNA fragment was 204 bp (non-deletion type) , and the melting temperature Tm value was 78.00, while in keng subspecies, and the length of cpDNA fragment was 135 bp (cpDNA deletion type) , and the melting temperature Tm value was 76.50 in hsien subspecies. Furthermore, 538 typical hsien-keng rice varieties and hsien-keng hybrids combinations from several provinces in China were selected to validate above identification method of hsien-keng subspecies rice based on chloroplast DNA SYBR-Green real-time fluorescent PCR detection. Among 177 samples of keng rices including keng type hybrids combinations, 170 samples with a Tm value of 78.00 were detected by SYBR-Green real-time PCR for cpDNA. The coincidence rate was 96.05% and the non-coincidence rate was 3.95% . Among the 361 samples of hsien rices including hsien type hybrids combinations, 331 samples with a Tm value of 76.50 were detected by SYBR-Green real-time fluorescence PCR for cpDNA. The coincidence rate was 91.69% and the non-coincidence rate was 8.31% . This method is simple, efficient and accurate for the identification of typical hsien-keng subspecies rice by detecting only one deletion site of chloroplast DNA in the cytoplasm.
Keywords hsien(indica) rice; keng(japonica) rice; chloroplast DNA(cpDNA); SYBR-Green fluorescence PCR; identification
基金项目:福建省科技计划农业引导性(重点)项目(2019N0026)
第一作者:陈文炳(1962—),男,汉族,福建福州人,博士,研究员,研究方向为农产品、食品分子生物学检测技术,E-mail:621213wbc@163.com
1.福州海关技术中心,福建省检验检疫技术研究重点实验室 福州 350001
1.Technology Center of Fuzhou Customs District, Fujian Provincial Key Laboratory of Inspection and Quarantine Technology Research, Fuzhou 350001
亚洲栽培水稻分为籼-粳稻亚种(hsien-keng),国际上也称为Oryza sativa L. subs. indica亚种与Oryza sativa L. subs. japonica亚种,其传统的鉴别方法主要是形态学鉴别与生化指标分类方法 [1-7],这些方法存在中间过渡类型而不能清晰分为两类的缺点。随着分子生物学技术的发展,越来越多的分子标记,如同工酶标记[7-8]及RFLP[9-11]、RAPD[12-13]、ISSR/SSR[14-16]、InDel (insertion/deletion, 粳特异插入缺失) [16-20]、细胞质分子标记、叶绿体[7,21-23]和线粒体DNA片段特异性[24]、反转录转座子(retrotransposon)[13]及微型倒转座子(mping) [25]等PCR扩增技术以及基于微流体芯片平台(Fluidigm platform)的SNP特异标记[26]均被应用于亚洲栽培稻的起源与籼-粳亚种的分化研究。日本学者Kanno等(1993)发现与粳稻相比,籼稻(indica)在叶绿体DNA(cpDNA,下同)的PstⅠ-12片段的ORF100区域中存在长度为69 bp的缺失片段[27]。由于长期的自然或人为杂交,遗传信息交流频繁,细胞核的DNA成分复杂,因此,基于核DNA的差异标记进行籼-粳亚种的鉴别难度较大。前面提到了已发表的基于细胞核DNA与cpDNA的PCR扩增多态性的籼-粳稻分化以及鉴别方法,检测的DNA位点均为多个甚至百个以上,作为籼-粳稻亚种的鉴定方法,十分繁琐,存在检测成本高、准确率偏低等问题。本技术仅检测1个遗传成分稳定的cpDNA位点,准确高效,经济简便。经检索,未见应用荧光PCR方法鉴定籼-粳稻亚种的报道。
基于籼-粳稻cpDNA的PstⅠ-12片段ORF100区域的69 bp碱基差异建立的普通PCR鉴别方法[7,20],其PCR产物的序列长度太长,达978 bp~1051 bp,检测成本高,且不适用于荧光PCR检测。因此,在该PCR产物测得的序列中,设计了4对引物(F1-F4,R1-R4),组成12个组合,从中筛选出籼-粳稻谷(米)的SYBR-Green 荧光PCR检测的熔解温度Tm值差异明显的F4R1引物对,探寻籼与粳稻扩增出的DNA片段长短与熔解温度Tm值的差异。基于典型性籼、粳稻品种样品的SYBR-Green 荧光PCR熔解温度Tm值与PCR扩增产物电泳及测序结果的差异比较,建立了籼-粳稻谷(米)的SYBR-Green荧光PCR快速鉴定方法。该方法仅需检测一个cpDNA位点,适用于实验室快速检测。另外,应用177个粳稻品种和粳型杂交组合样品与361个籼稻常规品种及籼型杂交组合样品进行方法准确率的验证试验,对不符合的原因进行了探讨分析。
1 材料与方法
1.1 材料
稻谷(米)主要搜集自委托实验室检验的水稻种子、福州本地超市食用大米以及福建省农业科学院、广西壮族自治区农业科学院、云南农业大学、凯里学院等单位的地方水稻品种和杂交稻组合,共538个样品。典型的粳稻东北珍珠米与典型的籼稻台籼11号用于籼-粳亚种的cpDNA特异性片段的PCR扩增与测序。
1.2 仪器与试剂
1.2.1 仪器
超微量核苷酸定量测定仪(美国Quawell Technology公司);实时荧光定量PCR仪(美国Bio-Rad公司);Power Pac 1000电泳仪(美国Bio-Rad公司);凝胶成像分析系统(美国Protein Simple公司)。
1.2.2 试剂
SYBR Premix Ex Taq(2times;)为宝生物工程(大连)有限公司(大连Takara公司)产品;引物序列参照文献[7,21]:大米-F:5'-AGTCCACTCAGCCATCTCTC-3',大米-R :5'-GGCCATCATTTTCTTCTTTAG-3'。用这对引物对典型粳稻东北珍珠大米和籼稻谷台籼11号进行SYBR Green荧光PCR测试,PCR产物委托铂尚生物技术(上海)有限公司测序。在测得的序列中,设计7条引物,正向(F)4条,反向(R)3条,序列如表1所示。引物由福州尚亚生物技术有限公司合成,用TE溶液(pH8.0)稀释至10 mu;mol/L,-20℃保存备用。
1.3 方法
1.3.1 DNA提取
将水稻种子或大米研磨成粉,称取1.0 g于50 mL离心管中,加5 mL CTAB裂解液,65℃温育过夜;15000 rpm离心10 min,取上清液;加2倍体积的CTAB沉淀液,摇匀,室温放置1 h;15000 rpm离心10 min,取沉淀,加900 mu;L 1.2 mol/L 氯化钠溶液,65℃温育,溶解后移至2.0 mL离心管中;加900 mu;L氯仿:异戊醇(24∶1),涡旋混匀,12000 rpm离心10 min;取上清液,加入等体积的氯仿,12000 rpm离心10 min;取上清液,加0.8倍体积的异丙醇,上下颠倒混匀,12000 rpm离心10 min;取沉淀,加入1 mL 70%乙醇,涡旋,12000 rpm离心10 min;弃上清,取沉淀,于DNA浓缩仪中干燥;加200 mu;L TE溶液(pH8.0),65℃温育至沉淀完全溶解;取2 mu;L DNA溶液于超微量核苷酸定量测定仪中测定浓度,用TE溶液(pH8.0)稀释至100 ng/mu;L,-20℃保存备用。
1.3.2 SYBR Green荧光PCR
PCR反应体系:SYBR Premix Ex Taq(2times;)12.5 mu;L,正、反向引物(10 mu;mol/L)各0.5 mu;L,DNA 2 mu;L,ddH2O 9.5 mu;L。
大米-F、大米-R引物对的PCR反应条件:95℃ 1 min;95℃ 10s,60℃1 min,40个循环;65℃~95℃熔解曲线分析。
表1 用于籼-粳稻鉴定的SYBR PCR引物设计
Table 1 Design of SYBR PCR primers for hsien-keng rice identification
正/反向引物 | 引物名称 | 引物序列(5'- -3') |
正向引物 | 大米-F1 | TAGATGAAATTGTATAAGTGG |
大米-F2 | AGACTTAGACCACGCAAGGC | |
大米-F3 | TTGTTACTAGATGTTCTATAGG | |
大米-F4 | AATCGCAACCCCTTTCCGC | |
反向引物 | 大米-R1 | TTGAGGATTATTCCATGATTCC |
大米-R2 | TCACAGCCGAGGTCGTGG | |
大米-R3 | GTCTCGAAGATAGAAATTTTGC |
其他引物对的PCR反应条件:95℃ 30 s;95℃ 5s,60℃34 s,40个循环;65℃~95℃熔解曲线分析。
PCR产物的长度进行测序或电泳验证。
2 结果与分析
2.1 籼粳稻米的特异性cpDNA片段PCR检测引物设计
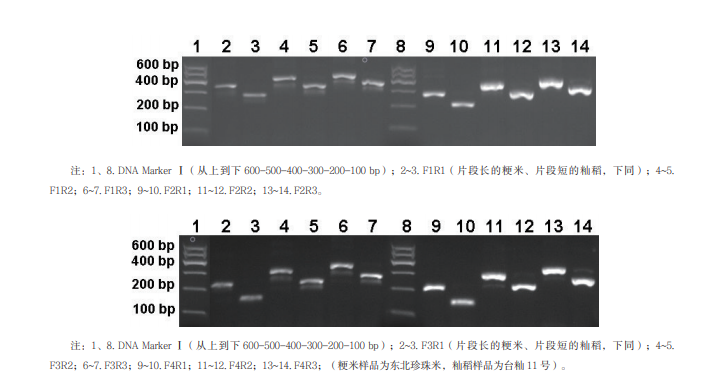
应用文献[7]的正向引物大米-F与反向引物大米-R,对典型的粳稻东北珍珠米与典型的籼稻台籼11号进行cpDNA特异性片段的PCR扩增与测序,测序结果如图1所示。2个亚种的cpDNA在PstⅠ-12片段的ORF 100领域中间部位相差69个碱基,即籼稻亚种与粳稻亚种相比,缺失69个碱基。在2条序列比对基础上,设计7条引物,其中,正向引物4条(F1-F4),反向引物3条(R1-R3)(见图2和表1),组合成12个引物对进行籼稻亚种与粳稻亚种的SYBR-Green 荧光PCR检测鉴定方法的优化。

图1 籼-粳稻叶绿体DNA特异性片段序列比对
Fig.1 Sequence comparison of chloroplast DNA specific fragments in hsien-keng rice

图2 7个引物所在位置
Fig.2 Location of 7 primers
2.2 建立籼-粳稻谷(米)的SYBR-Green 荧光PCR鉴定方法
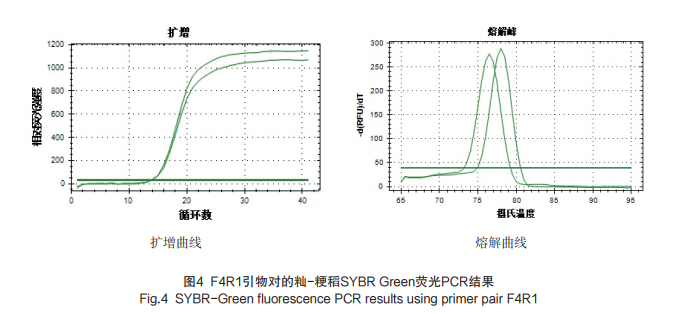
对2.1中组合成的12对引物进行SYBR-Green 荧光PCR熔解温度Tm值测定,结果如表2所示。表2表明,在12对引物中,籼粳稻的熔解峰的间距较大,且熔解温度Tm值差异最大的是F3R1与F4R1,均为1.5,籼、粳稻Tm值分别为76.50和78.00。图3表明,引物对F4R1的PCR扩增产物电泳条带大小135 bp的为cpDNA缺失的籼稻,204 bp为cpDNA不缺失的粳稻,在12对引物中最小,如表2所示,电泳图谱比F3R1扩增产物区分明显、条带清晰,因此,选取F4R1对用于区分籼、粳稻的PCR引物。F4R1引物对的SYBR-Green 荧光PCR扩增曲线和溶解曲线见图4(其他11对引物略)。
优化后建立的基于特异性cpDNA片段的籼-粳稻谷(米)SYBR-Green 荧光PCR鉴定方法:荧光PCR反应体系如1.3.2所述,正向引物F4,反向引物R1,序列如表1所示。PCR反应条件:95℃ 30 s;95℃ 5s,60℃34 s,40个循环。熔解温度Tm值76.50判定为籼稻,Tm值78.00判定为粳稻
表2 各对引物测得的籼粳稻谷PCR产物大小和Tm值比较
Table 2 Comparison of PCR product size and TM values of hsien-keng rice by primer pairs
片段 | 粳米(东北珍珠米) | 籼稻(台籼11号) | Tm差值 | ||
片段大小/bp | Tm值 | 片段大小/bp | Tm值 | ||
F1R1 | 327 | 78.50 | 258 | 77.50 | 1.00 |
F1R2 | 405 | 78.50 | 336 | 78.00 | 0.50 |
F1R3 | 442 | 79.00 | 373 | 78.50 | 0.50 |
F2R1 | 288 | 78.50 | 219 | 77.50 | 1.00 |
F2R2 | 366 | 78.50 | 297 | 78.00 | 0.50 |
F2R3 | 403 | 78.50 | 334 | 78.50 | 0 |
F3R1 | 236 | 78.00 | 167 | 76.50 | 1.50 |
F3R2 | 314 | 78.50 | 245 | 77.50 | 1.00 |
F3R3 | 351 | 78.50 | 282 | 78.00 | 0.50 |
F4R1 | 204 | 78.00 | 135 | 76.50 | 1.50 |
F4R2 | 282 | 78.50 | 213 | 77.50 | 1.00 |
F4R3 | 319 | 78.50 | 250 | 78.00 | 0.50 |
2.3 SYBR-Green荧光PCR方法在稻谷/米的籼-粳种类鉴定中的应用验证
2.2中所述建立的SYBR-Green 荧光PCR方法应用在供试538个稻谷样品的籼粳亚种鉴定检测,结果如表3所示:在177个粳稻谷(米)样品中,检出cpDNA非缺失型片段长度为204 bp、Tm值为78.00的有122个样品,cpDNA缺失型片段长度为135 bp、Tm值为76.50的有7个样品,与预想的粳稻谷(米)符合率为96. 05%,不符合率3. 95%;在361个籼稻谷(米)样品中,检出片段长度为135 bp的cpDNA缺失型、Tm值为76.50的有331个样品,cpDNA非缺失型片段长度为204 bp、Tm值为78.00有30个样品,与预想的籼稻谷(米)符合率为91. 69%,不符合率为8. 31%。经国家水稻数据中心(China Rice Data Center)查询,具有非缺失型DNA片段的30个品种中,22个籼型杂交组合样品具有粳稻cpDNA 非缺失型母系血缘,其余8个是常规品种。由此可见,本方法用于籼-粳稻谷(米)的鉴定,籼、粳稻谷(米)准确率分别高达91%以上和96%以上。
3 讨论
日本学者Kanno等(1993)发现,在籼稻(hsien/indica)cpDNA的PstⅠ-12片段的ORF100区域中存在长度为69 bp的缺失片段,而在粳稻(keng/japonica)cpDNA中则不存在缺失[27]。本文基于该发现,经过引物设计筛选,建立了适用于实验室快速鉴定籼-粳稻谷(米)亚种SYBR Green荧光PCR方法。其中,籼稻谷(米)与粳稻谷(米)的PCR产物长度各为135 bp与204 bp,熔解温度Tm值分别为76.50和78.00。该SYBR Green荧光PCR方法在538个已知籼、粳类别的稻谷(米)样品鉴定中得到验证,鉴定结果与水稻的籼、粳稻类别高度吻合。其中,供试177个粳稻谷(米)样品,检测结果与预想的粳稻谷(米)符合率为96.83%,不符合率为3.17%。361个籼稻谷(米)样品的检测结果与预想的籼稻谷(米)符合率为91. 69%,不符合率为8.31%。经国家水稻数据中心(China Rice Data Center)数据库查询,具有非缺失型DNA片段、Tm值为78.00的30个预计为籼稻的样品中,有22个籼型杂交组合具有粳稻cpDNA非缺失型母系血缘。上述存在的cpDNA的SYBR Green荧光PCR鉴定结果与预计的籼或粳稻亚种不符合的原因可能是传统上误名,或是以粳稻为母本、以籼稻为父本进行多代回交而形成具有粳稻细胞质的籼稻品种,又或者是相反情况。蔡星星等(2006)通过DNA插入缺失差异片段研究水稻籼-粳分化,揭示籼稻和粳稻品种存在渐渗杂交(introgression)的过程,籼稻品种中存在粳稻的血缘,虽然它们表现了明显的籼稻表型性状,但在分子水平上还保留了一些粳稻的特征位点[17]。另外,籼稻特别是籼型杂交稻的符合率不如粳稻高,原因可能与籼稻种植地域广,且杂交水稻组合的不育系培育引进粳稻父本细胞核和保留籼稻细胞质的遗传成分有关。而粳稻主要分布在高海拔、高纬度冷凉相对封闭地区[16,19],遗传物质交流少,变异频率低,因此cpDNA的PCR检测符合率高。已发表的基于核DNA[9-20]与细胞质的叶绿体与线粒体DNA的普通PCR方法[21-25],检测的DNA位点多个到上百个,而本文采用的SYBR Green荧光PCR方法未见报道,且只需检测一个遗传上相对稳定的细胞质cpDNA位点,相比之下更加经济简便、准确高效。本研究建立的籼-粳稻谷(米)样品鉴定方法的准确率还有提升空间,有待技术改进,同时期待进一步筛选籼-粳稻亚种特异性强的细胞核单基因位点进行荧光PCR扩增,与基于细胞质叶绿体的DNA标记的本方法互补,以提高籼-粳稻谷(米)亚种鉴定的准确率。
4 结论
建立的基于特异性cpDNA片段的籼-粳稻谷(米)SYBR-Green 荧光PCR鉴定方法:PCR反应体系为SYBR Premix Ex Taq(2times;)12.5 mu;L,正、反向引物(10 mu;mol/L)各0.5 mu;L,DNA 2 mu;L,ddH2O 9.5 mu;L。PCR反应条件:95℃ 30 s;95℃ 5s,60℃34 s,40个循环;65℃~95℃熔解曲线分析。正向引物F4序列为:5'-AATCGCAACCCCTTTCCGC-3',反向引物R1序列为:5'-TTGAGGATTATTCCATGATTCC-3',测得的熔解温度Tm值为76.50判定为籼稻,Tm值为78.00判定为粳稻。本方法经对538个稻谷(米)样品的籼-粳亚种鉴定验证,结果表明,籼、粳稻谷(米)准确率分别高达91%以上和96%以上,比已报道的籼-粳稻鉴别方法更准确、便宜、快速,可以应用推广。该方法的建立,将为维护正常的国内水稻种子与大米及其制品的贸易市场秩序,维护相关企业、消费者权益,以及为口岸执法,提供科学技术支撑与保障。
【该文经CNKI学术不端文献检测系统检测,总文字复制比为0.8%。】
注:1、8. DNA Marker Ⅰ(从上到下600-500-400-300-200-100 bp);2~3. F1R1(片段长的粳米、片段短的籼稻,下同);4~5. F1R2;6~7. F1R3;9~10. F2R1;11~12. F2R2;13~14. F2R3。
注:1、8. DNA MarkerⅠ(从上到下600-500-400-300-200-100 bp);2~3. F3R1(片段长的粳米、片段短的籼稻,下同);4~5. F3R2;6~7. F3R3;9~10. F4R1;11~12. F4R2;13~14. F4R3;(粳米样品为东北珍珠米,籼稻样品为台籼11号)。
参考文献
[1] 丁颖. 中国古来粳籼稻种栽培及分布之探讨与栽培稻种分类法预报[J]. 中山大学农学院农林研究委员会丛刊: 农艺专刊, 1946(6): 21-30.
[2] 丁颖. 中国栽培稻种的起源及其演变[J]. 农业学报, 1957, 8(3): 243-260.
[3] 程侃声. 亚洲稻籼粳亚种的鉴别[M]. 昆明: 云南科技出版社, 1993: 1-23.
[4] 陈文炳. 亚洲栽培稻起源分化的遗传学研究(综述)[J]. 上海农业学报, 1999, 15(3): 42-48.
[5] 加藤茂苞, 小坂博, 原史六. 杂种植物の结实度より见たる稻品种の类缘に就いて[J]. 九州帝大农学杂志, 1928, (2): 241-276.
[6] 凌启鸿,张洪程,丁艳锋. 关于亚洲栽培稻(Oryza sativa L.)两个亚种命名的商榷[J]. 中国农业科学, 2013, 46(2): 250-256.
[7] CHEN Wen-Bing, SATO Yo-Ichiro, NAKAMURA Ikuo, et al. Indica-japonica differentiation in Chinese rice landraces[J]. Euphytica, 1994, 74(3): 195-201.
[8] 汤圣祥, 魏兴华, 李自超, 等. 中国栽培稻同工酶的遗传多样性[J]. 中国农业科学, 2002, 28(2) : 203-207.
[9] Zhang Q F, Saghai M A, Lu T Y. Genetic diversity and differentiation of indica and japonica rice detected by RFLP analysis[J]. Theor Appl Genet, 1992, 83: 495-499.
[10] 庄杰云, 钱惠荣, 林鸿宣, 等. 应用RFLP标记研究亚洲栽培稻的起源与分化[J] . 中国水稻科学, 1995, 9(3):135-140.
[11] 孙传清, 王象坤, 吉村淳, 等. 普通野生稻和亚洲栽培稻核基因组的RFLP分析[J].中国农业科学, 1997, 30(4): 37-44.
[12] 龙雯虹, 许明辉. 籼稻和粳稻品种在RAPD上的遗传差异[J].云南农业大学学报, 2002, 17(3): 245-247.
[13] YAMANAKA Shinsuke, NAKAMURA Ikuo, NAKAI Hirokazu et. al. Dual origin of the cultivated rice based on molecular markers of newly collected annual and perennial strains of wild rice species, Oryza nivara and O. rufipogon[J]. Genetic Resources and Crop Evolution, 2003, 50: 529-538.
[14] 何予卿, 张宇, 孙梅, 等. 利用ISSR 分子标记研究栽培稻和野生稻亲缘关系[J] . 农业生物技术学报, 2001, 9(2): 123-127.
[15] 张晓丽, 郭辉,王海岗,等. 中国普通野生稻与栽培稻种SSR多样性的比较分析[J] . 作物学报, 2008, 3(4): 591-597.
[16] 徐建欣, 王云月, 姚春, 刘云霞, 汤淼. 籼粳稻品种在海拔梯度上的分化与分布[J] . 科学通报, 2012, 57(28-29): 2705-2714.
[17] 蔡星星, 刘晶, 仇吟秋, 等. 籼稻 93-11和粳稻日本晴DNA插入缺失差异片段揭示的水稻籼-粳分化[J]. 复旦学报(自然科学版), 2006, 45(3): 309-315.
[18] 赵伟, 夏寒冰, 章淑杰, 蔡星星. 籼-粳稻特异插入/缺失分子标记揭示的稻属植物遗传分化[J]. 复旦学报(自然科学版), 2008, 47(3): 281-287.
[19] 熊志远. 亚洲栽培稻籼—粳遗传分化及其与地理分布的关系[D]. 上海: 复旦大学, 2010.
[20] LIU Ping., CAI Xing-Xing, LU BaoRong. Single-seeded InDel fingerprints in rice: An effective tool for indicandash;japonica rice classification and evolutionary studies. Journal of Systematic and Evolution[J] . 2012, 50: 1-11.
[21] CHEN Wen-Bing, NAKAMURA Ikuo, SATO Yo-Ichiro, et al. Distribution of deletion type in cpDNA of cultivated and wild rice[J]. Japanese Journal of Genetics, 1993, 68(6) : 579-603.
[22] 杨杰, 王军, 曹卿, 等. 江淮流域杂草稻叶绿体DNA的籼粳分化[J]. 中国水稻科学, 2009, 23(4): 391-397.
[23] 王荣升, 魏鑫, 曹立荣, 等. 基于叶绿体基因多样性的中国水稻起源进化研究[J] . 植物遗传资源学报, 2011, 12(5): 686-693.
[24] 曹立荣, 魏鑫, 黄娟, 等. 基于线粒体基因片段核苷酸多态性的亚洲栽培稻起源进化研究[J] . 植物遗传资源学报, 2013,14(1): 18-24.
[25] HU, H., J. MU, H.J. ZHANG, et. al. Differentiation of a Miniature Inverted Transposable Element (MITE) System in Asian Rice Cultivars and Its Inference for a Diphyletic Origin of Two Subspecies of Asian Cultivated Rice[J]. Journal of Integrative Plant Biology, 2006, 48 (3): 260-267.
[26] SEO Jeonghwan, LEE Gileung, JIN Zhuo, et. al. Development and application of indicandash;japonica SNP assays using the Fluidigm platform for rice genetic analysis and molecular breeding[J]. Mol Breeding, 2020, 40(4): 1-16.
[27] KANNO A., WATANABE N., NAKAMURA I.,et al. Variations in chloroplast DNA from rice (Oryza sativa): differences between deletions mediated by short direct-repeat sequences within a single species[J]. Theor. Appl. Genet. 1993, 86: 579-584.
(文章类别:CPST-B)







