CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
利用gyrB基因鉴定芽孢杆菌近缘种
作者:宗 凯1 盛 旋1 尤征合1 吕侠影1 孙娟娟1 李云飞1 张 萍1 余晓峰1*
宗 凯1 盛 旋1 尤征合1 吕侠影1 孙娟娟1 李云飞1 张 萍1 余晓峰1*
摘 要 本试验从一批药品中分离到11株疑似芽孢杆菌菌株,采用VITEK 2 systems compact 生化鉴定系统和16S rDNA基因测序技术对其进行鉴定。结果显示,除2株株菌(6号菌株鉴定为Microbacterium oleivorans,8号菌株鉴定为Moraxella osloensis),其他9株株菌均属于芽孢杆菌属(Bacillus)。但仅通过VITEK 2 systems compact鉴定和16S rDNA测序比对,均不能鉴定到种。因此,进一步选择gyrB基因以及其blast分析结果构建系统发育树,最终将1~5号菌株分别鉴定为B. oceanisediminis(99.08%),B. korlensis(90.71%),B. licheniformis(100%),B. marisflavi(97.26%),B. oceanisediminis(99.22%),9~12号菌株分别鉴定为B. megaterium(99.83%),B. boroniphilus(81.20%),B. jeotgali(90.70%),B. humi(81.39%)。实验表明,利用16S rDNA与gyrB基因结合的方式能较好地将芽孢杆菌属鉴定到种,是一种快速准确的芽孢杆菌种间鉴定方法,适合于药品中分离菌株的种属及近缘种的鉴定与区分。
关键词 芽孢杆菌;16S rDNA;gyrB基因;鉴定
Identification of Related Species of Bacillus by gyrB Genes
ZONG Kai1 SHENG Xuan1 YOU Zheng-He1 LV Xia-Ying1
SUN Juan-Juan1 LI Yun-Fei1 ZHANG Ping1 YU Xiao-Feng1*
Abstract Eleven suspected Bacillus strains isolated from a batch of drugs were identified by VITEK 2 systems compact biochemical identification and 16S rDNA gene sequencing. The results showed that except for two strains (strain 6 was identified as Microbacterium oleivorans and strain 8 as Moraxella osloensis), the other 9 strains belonged to bacillus. However, the species could not be identified only by VITEK 2 systems compact identification and 16S rDNA sequencing. The phylogenetic tree was constructed by further selection of gyrB gene and its blast analysis results. Finally, strains 1 to 5 were identified as B. oceanisediminis (99.08%), B. korlensis (90.71%), B. licheniformis (100%), B. marisflavi (97.26%), B. oceanisediminis (99.22%), respectively, and strains 9 to 12 were identified as B. megaterium (99.83%), B boroniphilus (81.20%), B. jeotgali (90.70%) and B. humi (81.39%) respectively. The results showed that 16S rDNA combined with gyrB gene was a rapid and accurate method for species identification of Bacillus, suitable for the identification and differentiation of strains and their relatives.
Keywords Bacillus; 16S rDNA; gyrB gene; identification
通讯作者:余晓峰(1967&—),男,汉族,安徽合肥人,硕士,高级工程师,主要从事微生物分离和鉴定,E-mail: 1225987091@qq.com
1.合肥海关技术中心 合肥 230022
1. Technical Center of Hefei Customs, Hefei 230022
芽孢杆菌属(Bacillus)是一类产芽孢的广泛分布在自然界中具有抗逆能力的细菌,多数为革兰氏阳性菌株,好氧或兼性厌氧[1-2]。在《伯杰氏系统细菌学手册》第二版中列述了芽孢杆菌22个属,包含212种芽孢杆菌,因此,芽孢杆菌属是拥有庞大家族种的细菌。目前,大部分芽孢杆菌是根据《伯杰氏系统细菌学手册》测定菌种的基本特征和测定16S rDNA基因序列来鉴定的。虽然16S rDNA 序列作为一种重要的分子鉴定手段被广泛应用于细菌鉴定中,但是由于种间16S rDNA序列同源性太高,常常不能将分离到的芽孢杆菌鉴定到种[3-4]。本研究在一批药品的微生物限度检查中发现有疑似芽孢杆菌检出,经营养琼脂培养基分离、纯化得到单菌落,结合生化鉴定系统和16S rDNA测序进行鉴定难以确定其分类地位,随后根据16S rDNA测序和blast比对结果选择gyrB基因进行鉴定,通过构建系统发育树可以对其进一步分类和鉴定。
1 材料与方法
1.1 材料与试剂
营养琼脂培养基(北京陆桥生物有限公司);PCR mix预混液(上海近岸蛋白科技有限公司);PCR引物由合肥通用生物科技有限公司合成。
1.2 仪器与设备
VITEK 2 Systems compact(法国生物梅里埃公司);PCR仪(美国ABI公司);DNA/RNA全自动电泳系统(德国凯杰公司)。
1.3 从药品中分离疑似芽孢杆菌及纯化
挑取样品中的典型菌落,划线于营养琼脂培养基;于30℃~35℃培养18 h~24 h,均培养至出现单菌落为止。
1.4 VITEK 2 Systems compact生化鉴定
选用VITEK 2 Systems compact微生物全自动分析系统配套的鉴定卡及试剂,使用的鉴定卡包括革兰氏阴性细菌鉴定卡GN(货号21341)、革兰氏阳性细菌鉴定卡GP(货号21342)和芽孢杆菌鉴定卡(货号21345)。VITEK 2 Systems compact鉴定可信度打分高于85%的为可信鉴定结果,低于85%的不采信。
1.5 基因测序与系统发育树构建
提取菌落DNA基因组:从培养基上挑取单个菌落于1.5 mL EP管,65℃孵育1 h后采用磁珠法提取菌落DNA。
表1 扩增用引物
Table 1 Primers for PCR amplification
引物名称 | 引物序列 | 靶基因 | 扩增片段长度/bp |
16s rDNA-F | 5’-AGA GTT TGA TCA TGG CTC AG-3' | 16s rDNA | 1600 bp左右 |
16s rDNA-R | 5’-GGT ACC TTG TTA CGA CTT-3' | ||
芽孢杆菌gyrB796-F | 5’-GHCGBGGKATYCCDGTY-3' | gyrB | 796 bp左右 |
芽孢杆菌gyrB796-R | 5’-TTYGTYTTBGTYTGNCC-3' |
16S rDNA及gyrB基因扩增条件:预变性95℃ 5 min;变性94℃ 1 min,退火(16S rDNA基因退火条件60℃ l min;gyrB基因退火条件55℃ 30 s),延伸72℃ 2 min,30个循环;72℃ 10 min。待电泳有目的带后,送测序公司测序。
测序结果采用blast比对与分析,未能鉴定到种的菌株进一步采用gyrB基因等进行扩增测序与鉴定,具体引物序列如表1所示。
采用MEGA7.0软件邻位相连法(Neighbor-joining, NJ)构建系统发育树,选择最大复合似然法(Maximum composite likelihood, MCL)构建模型,使用1000次重复检测的Bootstrap值表示树上各节点的支持率。
2 结果与分析
2.1 生化特征鉴定结果
VITEK 2 systems compact全自动生化鉴定仪鉴定结果如表2所示。1~5号菌株鉴定结果与数据库匹配可信度不到90%;9号和11号菌株鉴定结果与数据库匹配可信度分别为95%和97%;10号菌株和12号菌株鉴定结果打分低于85%,结果不采信,即未能鉴定出结果。
2.2 基于16S rDNA基因鉴定并构建系统发育树
将16S rDNA测序序列进行blast比对分析,1号菌株得到Query coverage 99%和Max ident 99%的结果100个,其中,B.firmus 33个,B.oceanisediminis 31个,B.subtilis 1个,其余只能显示到属B.sp.。
2号菌株得到Query coverage 100%和Max ident 96%以上的结果49个,其中,B.korlensis 5个,B.beringensis 6个,其余只能显示到属B.sp.或显示Uncultured bacterium。
3号菌株得到Query coverage 100%和Max ident 100%的结果63个,B.aerius 2个,B.licheniformis 29个,B.tequilensis 1个,B.haynesii 13个,B.paralicheniformis 10个,其余只能显示到属B.sp.。
4号菌株得到Query coverage 100%和Max ident 99%的结果100个,其中,B. aquimaris 15个,B.marisflavi 31个,其余只能显示到属B.sp.。
5号菌株得到Query coverage 99%和Max ident 99%的结果100个,其中,B.oceanisediminis 32个,B.firmus 30个,B.subtilis 1个,其余只能显示到属B.sp.。7号菌株与5号菌株基因序列完全一致,应为同一株菌,因此合并为一株菌分析和进一步鉴定。
6号菌株得到Query coverage 99%和Max ident 99%的结果100个,鉴定到种的仅有Microbacterium oleivorans。
8号菌株得到Query coverage 99%和Max ident 99%的结果100个,Moraxella osloensis 34个,其余未显示种属。
9号菌株得到Query coverage 100%和Max ident 100%的结果100个,B.aryabhattai 27个,B.megaterium 21个,其余只能显示到属B.sp.。
10号菌株得到Query coverage 100%和Max ident 100%的结果100个,B.subterraneus 35个,B.thioparans 7个,B.boroniphilus 5个,B.cohnii 1个,其余只能显示到属B.sp.。
11号菌株得到Query coverage 100%和Max ident 100%的结果100个,B.boroniphilus 11个, B.haynesii 13个,B.jeotgali 12个,B.paralicheniformis 10个,B.subterraneus 5个,其余只能显示到属B.sp.。
12号菌株得到Query coverage 100%和Max ident 100%的结果100个,B.humi 12个,B.timonensis 15个,B.humi 12个,B.sinesaloumensis 1个,其余只能显示到属B.sp.。
从blast比对结果看,除6号菌株鉴定为Microbacterium oleivorans、8号菌株鉴定为Moraxella osloensis,其他9株菌均为芽孢杆菌属。从NCBI数据库下载37条芽孢杆菌属包含约20个种的16S rDNA基因序列,与1~5号菌株、9~12号菌株构建系统发育树,如图1(A)所示。1号和5号菌株与B.subtilis,B.oceanisediminis,B.firmus有较近的亲缘关系(Bootstrap值为100,Bootstrap值代表归类成簇的可信度,大于70表示可信),完全可以归为一簇。同理,2号菌株与B.korlensis,B.beringensis(Bootstrap值为100),3号菌株与B.haynesii,B. paralivheniformis,B.licheniformis,B.aerius,B.tequilensis(Bootstrap值为100),4号菌株与B.aquimaris,B.marisflavi(Bootstrap值为100),9号菌株与B.aryabhattai,B.megaterium(Bootstrap值为100),10号菌株与B.subterraneus,B.thioparans B.cohnii,B.boroniphilus(Bootstrap值为100),11号菌株与B.boroniphilus,B.subterraneus,B.jeotgali,B.selenatarsenatis(Bootstrap值为100),12号菌株与B.humi,B.timonensis(Bootstrap值为100),均各成簇。
2.3 基于gyrB基因鉴定并构建系统发育树
设计一对以gyrB基因为靶目标基因的通用引物,扩增片段长度约796 bp,经芽孢杆菌gyrB796-F/芽孢杆菌gyrB796-R扩增,1~5号菌株、9~12号菌株均能扩出目的片段,通过测序与blast比对,1号菌株与B.oceanisediminis相似度99.08%(Query coverage 99%),2号菌株与B.korlensis相似度90.71%(Query coverage 99%),3号菌株与B.licheniformis相似度100%(Query coverage 100%),4号菌株与B.marisflavi相似度97.26%(Query coverage 100%),5号菌株与B.oceanisediminis相似度99.22%,9号菌株与B.megaterium相似度99.83%(Query coverage 100%),11号菌株与B.jeotgali相似度90.70%(Query coverage 100%),10号菌株、12号菌株测序序列经blast比对未得到相似度大于90%的结果,10号菌株与B.boroniphilus相似度81.20%,12号菌株与B.humi相似度81.39%。
表2 11株菌株生理生化、16S rDNA测序及结合gyrB基因鉴定结果
Table 2 Identification results of VITEK 2 analysis, sequencing of 16S rDNA and gyrB genes of 11 B. strains
菌株编号 | VITEK 2 systems compact鉴定结果 | 16S rDNA测序比对鉴定结果 | 采用gyrB基因进一步鉴定结果 |
1 | Geobacillus toebii(87%) | B.subtilis, B.oceanisediminis, B.firmus | B.oceanisediminis(99.08%) |
2 | Bacillus firmus(87%) | B.korlensis, B.beringensis, B.levoglucosan | B.korlensis(90.71%) |
3 | Bacillus licheniformis(85%) | B.licheniformis, B.sonorensis | B.licheniformis(100%) |
4 | Bacillus megaterium(85%) | B.aquimaris, B.marisflavi | B.marisflavi(97.26%) |
5 | Bacillus firmus(85%) | B.oceanisediminis, B.firmus | B.oceanisediminis(99.22%) |
6 | / | Microbacterium oleivorans | - |
8 | / | Moraxella osloensis | - |
9 | Bacillus megaterium(95%) | B.aryabhattai, B.megaterium | B.megaterium(99.83%) |
10 | / | B.cohnii, B.boroniphilus | B.boroniphilus(81.20%) |
11 | Brebibacillus choshimemsis(97%) | B.boroniphilus, B.subterraneus, B.jeotgali, B.selenatarsenatis, B.thioparans | B.jeotgali(90.70%) |
12 | / | B.humi, B.timonensis, B.sinesaloumensis | B.humi(81.39%) |
注:/表示未能鉴定出结果;-表示未进行鉴定,其中,6号和8号菌株已采用16S rDNA鉴定到种,无需进一步鉴定。
从NCBI中下载25条芽孢杆菌属gyrB基因序列,结合1~5号菌株、9~12号菌株gyrB基因扩增产物测序序列构建了系统发育树,如图1(B)。基于gyrB基因序列blast结果构建的菌株系统发育树,可以将1号菌株、5号菌株鉴定为B.oceanisediminis,2号菌株鉴定为B.korlensis,3号菌株鉴定为B.licheniformis,4号菌株鉴定为B.marisflavi;9号菌株与B.megaterium,B.aryabhattai,10号菌株与B.boroniphilus,11号菌株与B.boroniphilus,12号菌株与B.humi,B.timonensis亲缘关系较近。
3 结论
从药品中分离到的菌株首先采用VITEK 2 systems compact全自动生化鉴定仪进行鉴定,鉴定可信度打分以85%作为临界值点,1~5号菌株、9号菌株、11号菌株鉴定可信度打分超过85%,10号菌株、12号菌株鉴定结果低于85%,不予采信。总体而言,1~5号菌株鉴定可信度打分在85%,处于临界值,与16S rDNA基因序列鉴定结果相比,可信度打分均低于16S rDNA基因序列鉴定。VITEK 2 systems compact全自动生化鉴定仪鉴定结果与16S rDNA基因序列鉴定结果一致的仅有3号菌株和9号菌株。采用VITEK 2 systems compact全自动生化鉴定仪因数据库受限,对菌株鉴定总体可信度得分不高,因此,采用VITEK 2 systems compact全自动生化鉴定仪很难将样品中分离的芽孢杆菌菌株鉴定到种。
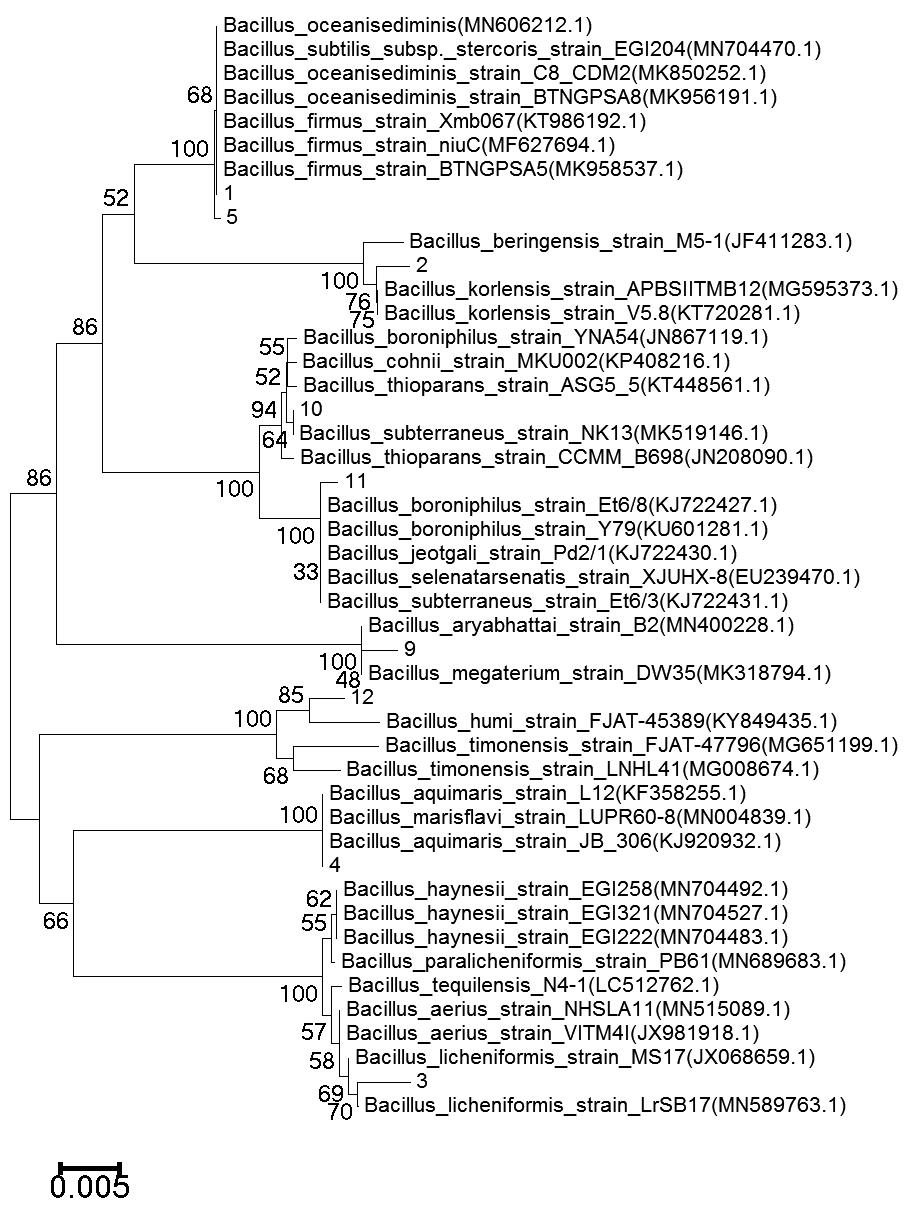
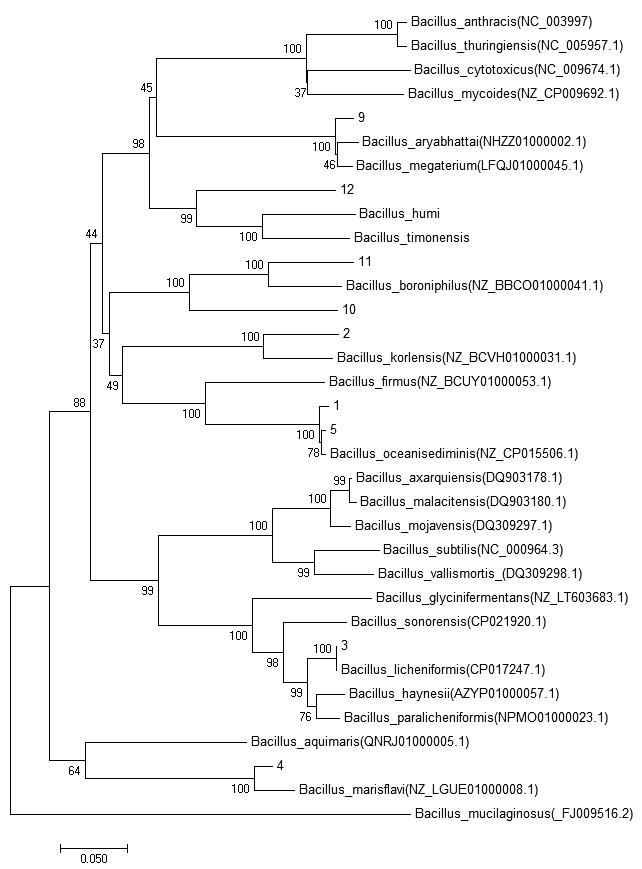
(A ) (B)
图1 基于16S rDNA基因(A)和gyrB基因(B)序列blast结果构建的菌株的系统发育树
Fig.1 Phylogenetic tree of Bacillus strain based on blast results of 16S rDNA and gyrB sequence
根据16S rDNA基因序列分析,初步鉴定自药品中分离的11株菌株中,9株经鉴定为芽孢杆菌属,另外2株分别是Microbacterium oleivorans菌、Moraxella osloensis菌。但由于16S rDNA基因难以区分亲缘关系接近的菌种,如1号菌株采用16S rDNA 测序比对有B.subtilis,B.oceanisediminis,B.firmus等3个种100%匹配,9株菌至少有2个种与之100%匹配,难以鉴定到种。为进一步鉴定芽孢杆菌属9株菌,引入了gryB基因[5-6]。
gyrB基因是普遍存在于细菌中编码促旋酶B亚单位的基因,该基因序列长度约1.2 kb~1.4 kb,很多文献研究表明该基因序列在区分和鉴定细菌近缘种方面比16S rDNA基因更有优势。文献[7]通过对枯草芽孢杆菌类的gyrB基因序列、16S rDNA基因序列比较分析,结果表明8种近缘枯草芽孢杆菌的16S rDNA序列的相似性在98.1%~99.8%之间,难以区分;而gyrB基因的相似性则在75.4%~95.0%之间,结果表明gyrB基因是在种水平上区分枯草芽孢杆菌近缘种的有效靶标基因。文献[8]采用16S rDNA基因序列比较分析,18株H血清型蜡状芽孢杆菌间的相似率均在99%以上,16S rDNA基因不能区分各菌株,而基于gyrB基因序列的系统发育树明显将其分为4组,这也表明与16SrDNA相比,gyrB基因在蜡状芽孢杆菌近缘种具有更高的差异性。
本课题实验经16S rDNA鉴定为芽孢杆菌属的9株菌,由gyrB基因进一步鉴定,1号菌株与B.oceanisediminis相似度99.08%(Query coverage 99%),2号菌株与B.korlensis相似度90.71%(Query coverage 99%),3号菌株与B.licheniformis相似度100%(Query coverage 100%),4号菌株与B.marisflavi相似度97.26%(Query coverage 100%),5号菌株与B.oceanisediminis相似度99.22%,9号菌株与B.megaterium相似度99.83%(Query coverage 100%),11号菌株与B.jeotgali相似度90.70%(Query coverage 100%),10号菌株、12号菌株测序序列经blast比对未得到相似度大于90%的结果;除10号菌株、12号菌株未能被鉴定,1~5号、9号、11号共7株株菌基本得到鉴定,除11号菌株相似度为90.70%,其他菌株相似度均在97%以上。
综上所述,将未知芽孢杆菌菌株鉴定到属,采用生化鉴定或16S rDNA基因鉴定都可以,但要做种间鉴定需要利用16S rDNA结合特异性基因信息进行,比如16S rDNA和gyrB基因结合进行blast比对和系统发育树分析,可以快速、准确地将目标菌株鉴定到种。
【该文经CNKI学术不端文献检测系统检测,总文字复制比为2%。】
参考文献
[1]喻国辉, 牛春艳, 陈远凤, 等. 利用16S rDNA结合gyrA和gyrB基因对生防芽孢杆菌R31的快速鉴定[J]. 中国生物防治学报, 2010, 26(002): 160-166.
[2]严婉荣, 肖敏, 陈圆, 等. 芽孢杆菌基本特征, 16SrRNA对比分析及特异性基因挖掘[J]. 基因组学与应用生物学, 2017, 036(011): 4686-4692.
[3]安然, 易图永, 肖启明, 等. gyrB基因在细菌分类和检测中的应用[J]. 江西农业学报, 2010, 22(004): 18-20.
[4]郝云婕, 韩素贞. gyrB基因在细菌系统发育分析中的应用[J]. 生物技术通报, 2008, (002): 39-41.
[5]李献梅, 王小芬, 杨洪岩, 等. 促旋酶(gyrase) B亚单位基因gyrB在鉴别细菌近缘种中的应用[J]. 微生物学报, 2008, 48(5): 701-706.
[6]申应德, 张新, 李杰, 等. 药品中非脱羧莱克勒菌的分离与鉴定[J]. 药学研究, 2013(10): 566-568.
[7] Wang L T, Lee F L, Tai C J, et al. Comparison of gyrB gene sequences, 16S rRNA gene sequences and DNA-DNA hybridization in the Bacillus subtilis group[J]. International Journal of Systematic Evolutionary Microbiology, 2007, 57(8): 1846-1850.
[8] La Duc MT, Satomi M, Agata N, et al. gyrB as a phylogenetic discriminator for memhers of the Bacillus anthracis-cereus-thuringiensis group[J]. Journal of Microbiological Methods, 2004, 56(3): 383-394.
(文章类别:CPST-A)







