CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
COⅠ和16S rDNA基因在台湾蠛蠓鉴定中的应用
作者:黄恩炯1 张建庆1 蔡怡珊1 钟艳红1,2 高 博1*
黄恩炯1 张建庆1 蔡怡珊1 钟艳红1,2 高 博1*
摘 要 本文对台湾蠛蠓(Lasiohelea taiwana)的COⅠ和16S rDNA基因序列进行了测定,并分析2个基因在台湾蠛蠓分子鉴定中的应用前景。结果表明,台湾蠛蠓COⅠ基因片段长度为709 bp,与台湾铗蠓(Forcipomyia taiwana) 序列同源性最大(98%~99%),分子鉴定与传统形态学鉴定结果一致,而16S rDNA基因片段长度为542 bp,与库蠓属的Culicoides tuttifrutti较为相似(82%)。COⅠ基因还可用于蠓类不同属分类鉴定。因此,COⅠ和16S rDNA基因适用于蠓类的分子鉴定。
关键词 台湾蠛蠓;COⅠ;16S rDNA
The Application of COⅠand 16S rDNA Genes in the Identification of Lasiohelea taiwana
HUANG En-Jiong1 ZHANG Jian-Qing1 CAI Yi-Shan1 ZHONG Yan-Hong1,2 GAO Bo1*
Abstract The paper used COⅠand 16S rDNA sequences to identify Lasiohelea taiwana. It continued to discuss the application prospect of the two genes in the identification of L. taiwana. The result showed that the fragment length of COⅠ was 709 bp, with 98%~99% homology of Forcipomyia taiwana; and 16S rDNA was 542 bp, with 82% homology of Culicoides tuttifrutti. It also demonstrated that the molecular identification based on COⅠgene sequence was consistent with the morphological identification. Additionally, COⅠcould be used for potential identification and classification of different kinds of genus in Ceratopogonidae. Therefore, the sequences of COⅠ and 16S rDNA could be employed in the molecular identification of biting midges.
Keywords Lasiohelea taiwana; COⅠ; 16S rDNA
台湾蠛蠓(Lasiohelea taiwana)是重要的吸血蠓类,广泛分布于福建、浙江、台湾等地,不仅刺吸人畜引起虫咬性皮炎,而且传播病原体造成人畜共患病的暴发[1],对人类健康和畜牧业发展造成很大危害。20世纪50年代,福建和广东两省日本乙型脑炎疫情严重流行期间,人们曾从两地自然界中捕获的台湾蠛蠓体内分离出日本乙型脑炎病毒[2-3],由此,台湾蠛蠓被认为是南方地区日本乙型脑炎的潜在媒介之一。此外,该蠓被证实会传播其他多种细菌性、病毒性、丝虫性和原虫性疾病[4]。因此,对台湾蠛蠓的准确鉴定是监测与控制蠓传疾病发生的重要前提。
当前,蠓的鉴定仍主要通过制作玻片标本进行形态学鉴定,但其鉴定结果易受玻片质量影响,且整个过程周期长、步骤繁琐,一旦遇到近源种或形态特征不完整的标本,难以准确鉴定。为此,亟须找到一种更为准确、便捷的鉴定方法。
随着分子生物学的不断发展,基于分子层面的鉴定手段已融入传统的分类研究中[5]。分子分类鉴定与形态鉴定相比,不仅不受其环境因素、个体生长阶段的影响,而且还可提供直接、充分、定量的生物进化信息,因此受到分类学家的青睐。常见的分子鉴定技术有DNA条形码技术[6]、PCR技术[7]、高通量测序技术、生物信息学分析等,这些技术的发展和应用,佐证了传统的形态分类鉴定,加快了物种分类鉴定的脚步,已成为现代物种分类研究的一个新方向。
细胞色素C氧化酶亚基Ⅰ(COⅠ)两端的序列相对保守,序列插入和缺失较少,同时有足够的变异可区别不同物种。16S rDNA基因进化速率缓慢,是昆虫线粒体较为保守的基因,国内外学者在昆虫的高级分类阶元的分析中对其研究较多[8-9]。因此 ,COⅠ和16S rDNA是昆虫种、属间系统发育研究中频率使用的分子标记[10]。
本文以台湾蠛蠓为研究对象,对其COⅠ和16S rDNA基因进行序列测定和分析,构建系统发育树,以期探索COⅠ和16S rDNA基因在台湾蠛蠓分子鉴定应用的适用性,为吸血蠓的分子分类奠定基础。
1 材料与方法
1.1 供试标本
供试标本采自福建省泉州地区。标本制作参照《中国蠓科昆虫》(虞以新主编,2006)[11]。玻片标本应与用于DNA提取的标本一一对应,准确无误,才能确保形态和分子特征双重鉴定的有效性。鉴定结果经国内权威专家复核后用于试验。
1.2 台湾蠛蠓基因组DNA提取法
参照基因组DNA小量抽提试剂盒(离心柱式)(Genomic DNA Mini Preparation Kit with Spin Column)说明书,略有改动。
1.3 PCR
1.3.1 COⅠ基因
采用COⅠ基因通用引物LCO 1490 (5'–GGT CAA CAA ATC ATA AAG ATA TTG G–3')和HCO2198(5'–TAA ACT TCA GGG TGA CCA AAA AAT CA–3') (Folmer et al. 1994)[12]。反应体系:DNA模板10μL,上下游引物各1μL,2×MasterMix 25μL,加双蒸水补至50μL。反应条件:94℃预变性2 min,94℃变性40 s,48℃退火40 s,72℃延伸40 s,35个循环,最后72℃再延伸10 min。
1.3.2 16S rDNA基因
引物由实验室自行设计,即16S21:5'-GCCTGTTTATCAAAAACAT-3';16S22:5'-CCGGTCTGAACTCAGATCA-3'。 PCR反应体系和反应条件同COⅠ基因。
取5 μL PCR产物,在1.0%琼脂糖凝胶中电泳,电泳结束后于凝胶成像仪中拍照。
1.4 序列测定及分析
委托生工生物工程(上海)股份有限公司进行PCR产物的纯化与测序。采用MEGA 6.06、DNAMAN等软件进行序列分析并构建系统进化树。
2 结果与分析
2.1 PCR产物电泳
台湾蠛蠓COⅠ、16S rDNA基因PCR扩增产物经1.0%琼脂糖凝胶电泳后,两个基因分别在约750 bp和约630 bp左右处有1条特异性的明亮条带,如图1所示。
2.2 COⅠ和16S rDNA序列的基本特征
共获得5条台湾蠛蠓COⅠ基因序列,扩增长度为709 bp,为1个完整的阅读框,编码236个氨基酸残基,无碱基的插入与缺失。在COⅠ基因的709个核苷酸位点中,变异位点18个,简约信息位点10个,T、C、A、G碱基的平均含量分别为40.3%、15.2%、27.9%和16.6%。在密码子的不同位点,碱基组成出现较大差异。
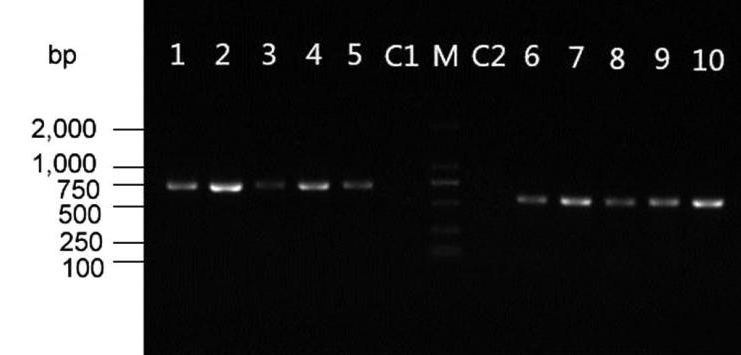
M: DL-2000 Marker; 泳道1~5: COⅠ基因; 泳道6~10: 16S rDNA基因
图1 台湾蠛蠓COⅠ、16S rDNA基因PCR电泳图
Fig.1 PCR Electrophoresis of COⅠ and 16S rDNA gene of L. taiwana
M:DL-2000 Marker, Lane 1~5:PCR amplification of COⅠ, Lane 6~10:PCR amplification of 16S rDNA
在236个氨基酸残基中,包含19种氨基酸,其中,使用频率最高的是Leu,占总数的27.7%;其次是Phe(11.5%)、Tyr(10.0%)、Glu(9.0%);使用频率最少的是Ala与Asn,均是0.5%。
共获得5条台湾蠛蠓16S rDNA基因序列,扩增长度为542 bp。在16S rDNA基因的542个核苷酸位点中,变异位点18个,简约信息位点6个,T、C、A、G碱基的平均含量分别为40.1%、8.9%、37.4%和13.6%。
2.3 COⅠ、16S rDNA序列的联机检索及系统发育分析
不同台湾蠛蠓COⅠ序列地理株间相似性为98%~100%,与Forcipomyia taiwana 序列同源性最大(98%~99%)。采用邻接法(NJ),以台湾蠛蠓的COⅠ基因序列构建系统进化树。采用MEGA 6.06 软件,以Kimura双参数模型为碱基替代模型建树,1000次bootstrap法进行检验,如图2所示。
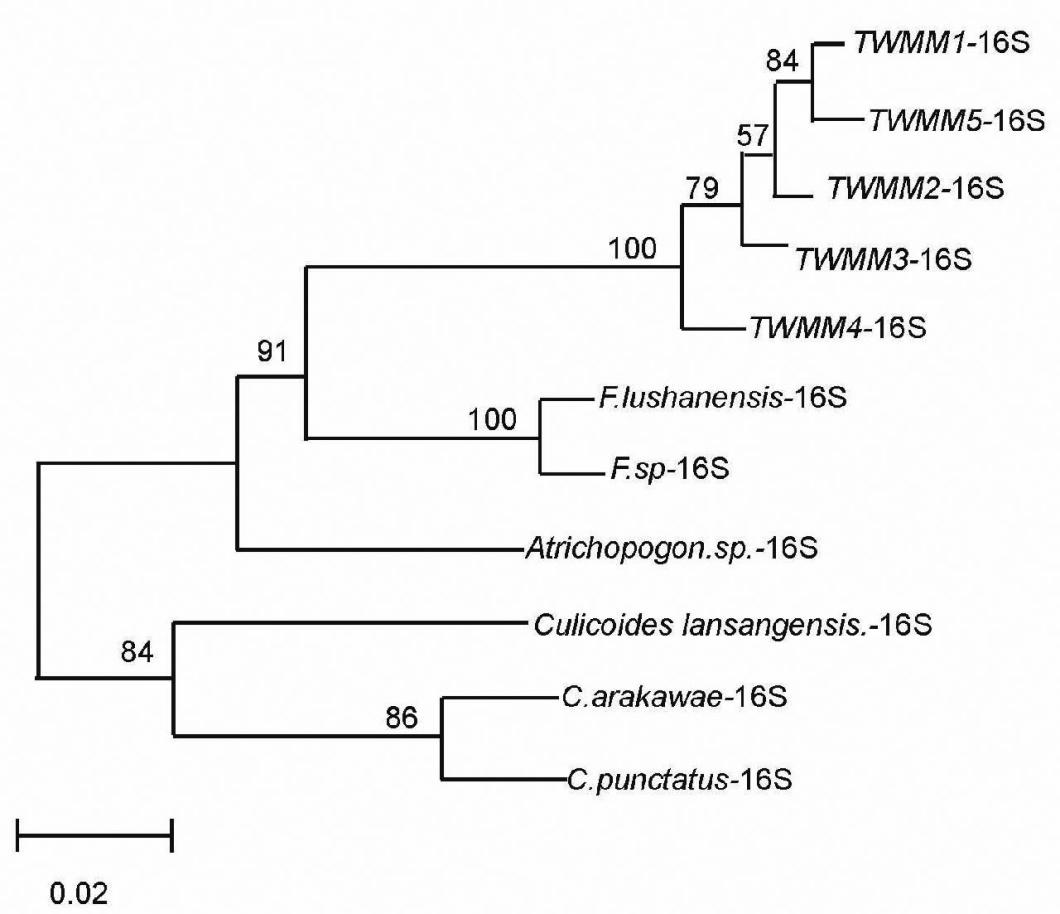
图2 台湾蠛蠓COⅠ基因系统进化树
Fig.2 Phylogenetic tree of COⅠ gene sequences of L. taiwana
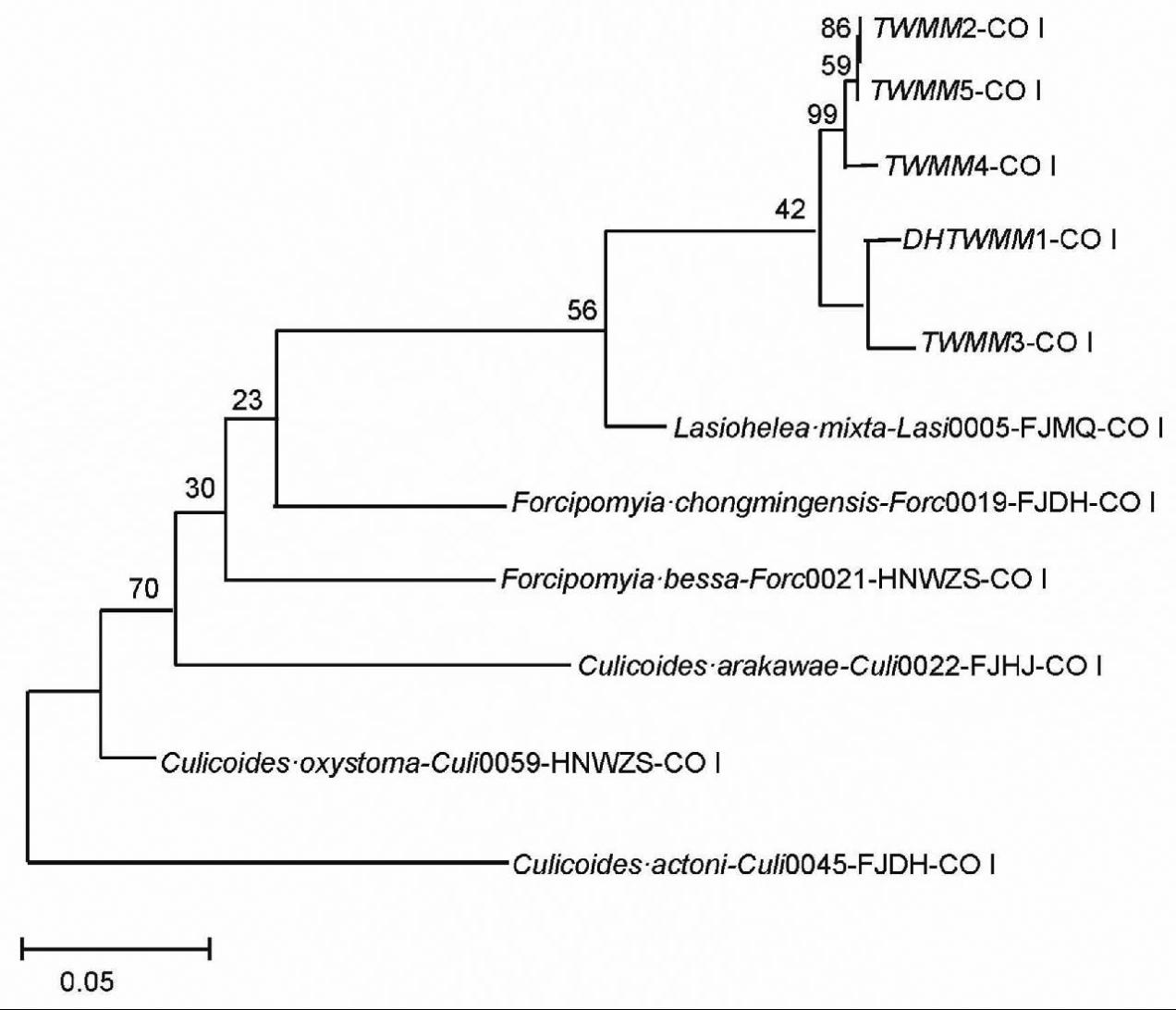
图3 台湾蠛蠓16S rDNA基因系统进化树
Fig.3 Phylogenetic tree of 16S rDNA gene sequences of L. taiwana
不同台湾蠛蠓16S rDNA地理株的亲缘性同样为98%~100%,与 Culicoides tuttifrutti的同源性为82%。
采用邻接法(NJ),以台湾蠛蠓的16S rDNA基因序列构建系统进化树。采用MEGA 6.06软件,以Kimura双参数模型为碱基替代模型建树,1000次bootstrap法进行检验,如图3所示。
以台湾蠛蠓的COⅠ、16S rDNA基因序列构建的系统进化树均表明,台湾蠛蠓和铗蠓属聚类在进化树上的同一分支,这也从分子生物学角度佐证了形态鉴定结果。
3 讨论
台湾蠛蠓COⅠ基因和16S rDNA基因地理株间亲缘性均为98%~100%。序列分析结果表明,台湾蠛蠓COⅠ基因与台湾铗蠓F. taiwana 序列同源性最大,为98%~99%;台湾蠛蠓16S rDNA基因与蠓科昆虫的C. tuttifrutti同源性最大,为82%。
从系统进化树可以看出,台湾蠛蠓和铗蠓属聚类于进化树上的同一分支,而与库蠓属在不同分支上。蠛蠓属原是铗蠓属的亚属,但因其特有口甲齿等形态,且具有吸血习性,而铗蠓属其他亚属均无以上特点,因此,被分类学家分成独立的一个属[13]。本文的实验结果从分子生物学角度上佐证了形态分类学的结果。虽然基于线粒体基因的DNA条形码技术及其应用前景被众多学者看好,但也有人认为,如此短的DNA片段难以提供物种水平的可靠信息,完全依靠DNA条形码会导致鉴定错误[14-15]。因此, 在利用单基因或多个基因进行物种分类和种属鉴定时, 不能只考虑所选用基因片段的长度, 而应结合样本来源、鉴定者、序列组成及其同源性等进行综合评估[16]。
4 结论
据报道,不同物种的种间遗传距离大于2%,而同种的遗传距离小于2%[17]。台湾蠛蠓不同地理株间COⅠ和16S rDNA基因序列同源性均在2%之内,而与不同种吸血蠓的遗传距离也均超过2%,因此,COⅠ和16S rDNA基因可应用于台湾蠛蠓的分子鉴定,尤其是对于不同种属的种类鉴定具有较好的效果,可从分子水平上佐证形态鉴定的结果。但分子分类应与传统形态学鉴定结果相互佐证,从而有利于进一步提高蠓类鉴定结果的准确性。
【该文经CNKI学术不端文献检测系统检测,总文字复制比为3.1%。】
参考文献
[1] 虞以新, 刘金华. 中国蠓科昆虫研究的发展(双翅目)[J]. 寄生虫与医学昆虫学报, 2000, 7 (01): 4-9.
[2] 吴皎如, 吴树吟. 由糠蚊科蠛蠓属Lasiohelea台湾蠛蠓分离出乙型脑炎病毒[J]. 微生物学报, 1957, (05) : 22-26.
[3] 容瓘, 柯小麟. 从广州市天然界蠛蠓分离流行性乙型脑炎病毒的试验[J]. 微生物学报, 1962, 8 (03) : 280-284.
[4] 王飞鹏, 黄恩炯, 蔡亨忠, 等. 吸血蠓及其传播的疾病[J]. 昆虫知识, 2010, 47( 06 ) : 1270-1273.
[5] 黄原, 袁锋, 周尧. 昆虫核酸分子系统学研究进展[J]. 昆虫分类学报, 1995, 17(03): 180-184.
[6] 程希婷, 王爱民, 顾志峰, 等. DNA条形码研究进展[J]. 基因组学与应用生物学, 2011, 30(06), 748-758.
[7] 杨东升, 邓兵, 夏耕田. 聚合酶链式反应(PCR)用于蚊虫分类鉴定的研究概况[J]. 医学动物防制, 2002, 18 (06): 317-319.
[8] 王德明, 林杨. 线粒体DNA基因序列在昆虫分子系统学研究中的应用[J]. 广东农业科学, 2010, (06 ): 188-190.
[9] 刘会, 李正西, 李淑娟, 等. 线粒体DNA序列在半翅目异翅亚目昆虫分子系统学上的应用[J]. 昆虫分类学报, 2007, 29 (04): 265-274.
[10] 王飞鹏, 黄恩炯, 肖武, 等. 吸血蠓分子分类研究进展[J]. 中国人兽共患病学报, 2012, 28 (12): 1227-1232.
[11] 虞以新主编. 中国蠓科昆虫[M]. 北京:军事医学科学出版社, 2006: 1682-1683.
[12] Folmer O, Black M, Hoeh W, et al. (1994). DNA primers for amplif ication of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates [J]. Molecular Marine Biology and Biotechnology, 2010, 3(6) : 294-299.
[13] 虞以新, 刘康南.中国蠛蠓的研究[M]. 北京: 科学出版社, 1982: 1-84.
[14] Will KW, Rubinoff D. Myth of the molecule: DNA barcodes for species cannot replace morphology for identification and classification.Cladistics, 2004, 20(1): 47-55.
[15] Ebach MC, Holdrege C. DNA barcoding is no substitute for taxonomy. Nature, 2005, 434 (7034 ): 697.
[16]王崇财, 陈敏, 杨美琼, 等. rDNA-ITS序列在吸血蠓分子鉴定中的应用研究[J]. 寄生虫与医学昆虫学报, 2018, 25(02): 92-100.
[17] Hebert PD, Stoecklem MY, Emlak TS, et al. Identification of birds through DNA barcodes: [J]. PLoS Biology, 2004, 2(10): e312.
(文章类别:CPST-C)







