CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
应用DNA条形码技术鉴定宁波口岸本底非常见蝇类
作者:刘永磊1 郭立新1 邱佳琦2 吴 薇1*
刘永磊1 郭立新1 邱佳琦2 吴 薇1*
Application of DNA Barcoding to Identify Uncommon Flies in Ningbo Port
LIU Yong-Lei1 GUO Li-Xin1 QIU Jia-Qi2 WU Wei1*
Abstract Species identification was conducted for five local uncommon flies captured in the fly monitoring at Ningbo port. Subjected to CO Ⅰ gene amplification and sequencing, the sequences was aligned in BOLD and GenBank databases, and compared with their similar sequences to calculate the genetic distance and build a phylogenetic tree. Four of the five flies were identified to specific species by sequence analysis combined with morphological features, and one CO Ⅰ gene sequence was not successfully identified without high-quality matches in the database. The study shows that DNA barcoding technique combined with morphological identification can identify flies of unknown species quickly and efficiently.
Keywords DNA barcoding; cytochrome C oxidase subunit Ⅰ gene; fly species identification
蝇类作为一种重要的病媒昆虫,其医学重要性在于能够机械传播病原体。由于不同种类蝇的孳生环境及生存习性存在差异,其携带的病原体种类也千差万别,因此蝇类种类鉴定是蝇类防制的首要工作内容。蝇类种类鉴定对于了解口岸本底病媒生物构成及对外来入侵物种识别具有重要意义。
目前,蝇类种类鉴定主要采用形态学鉴定方法。对于常见蝇种,形态学鉴定方法具有操作简便且效率较高的优点,主要依赖鉴定人员的经验。对于出现频次较低的非常见蝇种,由于蝇类的形态学特征相对复杂,种间差异较小,存在较大的鉴定难度,对鉴定人员专业水平要求更高。此外,对于形态残缺或者处于特定发育时期而不具备必要鉴定特征的对象,形态学鉴定方法同样不适用[1]。
自 2003 年 DNA 条形码(DNA barcoding)技术问世以来,受到了生物分类学领域的广泛关注[2]。DNA条形码技术选用各物种间有一定保守性,且具有足够种间变异性的基因序列作为目标片段。该片段能够使用通用引物在不同物种间方便地扩增,并用扩增产物的基因测序结果来区分不同物种。其中,线粒体细胞色素C氧化酶亚基Ⅰ(CO Ⅰ)基因能够满足大多数物种鉴定的需求,被作为通用型分子标记。邱德义等[3]运用DNA条形码技术对国内49种丽蝇科常见蝇种进行了分类鉴定,鉴定结果与形态学鉴定一致。吴薇等[4]对宁波口岸进境的24种有瓣蝇类,采用DNA条形码技术并结合形态学鉴定进行了有效的种类鉴定。杜鹃等[5]对上海口岸截获的粉蝇属干制标本进行CO Ⅰ条形码研究,结果表明条形码技术可以弥补传统形态学分类方法的不足,提高蝇类干制标本鉴定的成功率。
本研究通过对宁波口岸捕获的5只蝇进行DNA条形码鉴定,探讨了DNA条形码技术在非常见蝇种鉴定中的应用。通过与传统形态学鉴定方法比对与结合,以期建立高效准确的蝇类分类鉴定方法,为口岸本底蝇类及进境蝇类鉴定提供有效技术支撑。
1 材料与方法
1.1 材料
本研究所用样本为2021年4月宁波海关所属梅山海关在梅山保税港区围网外荒地上捕获的5只本底非常见蝇种。这5只蝇经过形态学初步鉴定均未鉴定到具体种类,将其分别编号为210415-1、210415-2、210415-3、210415-4、210415-5。DNA提取使用Qiagen血液/组织基因组DNA提取试剂盒;PCR试剂采用TaKara PrimeSTAR Max 预混液;DNA Marker(B500350)、琼脂糖、GoldView核酸染料和TAE缓冲液干粉均购自生工生物工程(上海)股份有限公司。
1.2 DNA提取及基因片段扩增
操作方法主要参照SN∕T 4278-2015《国境口岸医学媒介昆虫DNA条形码鉴定操作规程》。首先,采集蝇一侧中足,置于1.5 mL离心管中,加入试剂盒附带的裂解液,并用研磨棒碾碎。然后,按试剂盒使用说明书提取蝇类总DNA,将提取后的DNA样品置于-20℃保存备用。
CO Ⅰ通用引物LCO1490 5'-GGTCAA CAAATCATAAAGATATTG-3'、HCO2198 5'-TAAACTTCAGGGTGACCAAAAAATCA-3' [6]由生工生物工程(上海)股份有限公司合成。
在PCR反应管中,加入2×预混液体积25 μL,上下游引物各2 μL,DNA模板3 μL,补充去离子水至50 μL。
反应条件:95℃预变性5 min;95℃变性30 s,55℃退火30 s,72℃延伸1 min,共30个循环;72℃最终延伸10 min。
PCR产物使用浓度为1.0%琼脂糖凝胶,在5 V/cm的电压下进行电泳,电泳结束后拍照分析。选取电泳条带清晰且单一的PCR产物交由生工生物工程(上海)股份有限公司进行测序。
1.3 序列分析
PCR产物进行测序后,去除序列两端的测序不稳定区域,使用BOLD在线系统对测序序列进行物种信息比对[7]。在GenBank数据库中通过BLAST进行同源搜索,选取同源序列。使用MEGA7.0软件,利用Kimura 2-parameter模型计算遗传距离,采用邻接法构建系统发育树,Bootstrap值设定为1000。
2 结果
2.1 PCR扩增结果
5只蝇提取的DNA样品,经过PCR扩增后,再将产物经1.0% 琼脂糖凝胶电泳分析,得到长度约700 bp的扩增产物片段,与预期大小一致,如图 1所示。
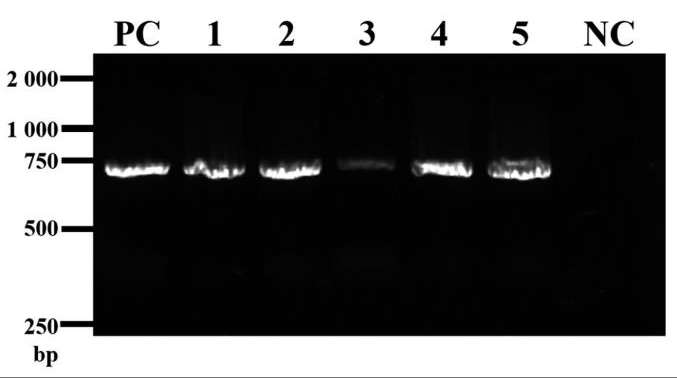
PC:阳性对照(Positive control);1:210415-1;2:210415-2;3:210415-3;4:210415-4;5:210415-5;NC:阴性对照(Negative control)
图1 宁波口岸监测5只蝇CO Ⅰ基因扩增产物电泳图
Fig.1 Electrophoresis of CO Ⅰ gene amplification products of five flies monitored at Ningbo port
2.2 序列在数据库中的比对分析
PCR产物进行测序后,去除序列两端的测序不稳定区域,长度为635 bp,将上述序列提交至BOLD数据库进行物种信息比对。210415-1序列与绿蝇属(Lucilia)的同源性为100%,与Melinda pusilla基因相似性为99%以上;210415-2样本在系统中无匹配序列;与210415-3序列有最高同源性的为铜腹重毫蝇(Dichaetomyia bibax);与210415-4和210415-5序列匹配度最高的是拟新月陪丽蝇(Bellardia menechmoides)。5只蝇中的4只CO Ⅰ基因在GenBank数据库中存在98%以上的同源性序列,而210415-2样本的CO Ⅰ基因序列在GenBank数据库中同源性最高的序列仅为93.88%,具体情况见表1。
2.3 序列进化分析
将5条序列采用Kimura 2-parameter模型进行计算,得出5只蝇序列间的种间遗传距离,其中,210415-4与210415-5之间遗传距离为0,进一步说明2只蝇为同一蝇种;剔除210415-5后,其余4只蝇的平均遗传距离为0.133,显著高于种间平均遗传距离的阈值0.02,见表2。序列信息利用BLAST工具和GenBank数据库中序列进行比对。收集与每个样本CO Ⅰ基因相匹配的前6条序列,去除重复序列后与原序列一并构建进化树,如图2所示。从聚类结果观察,210415-1、210415-3、210415-4、210415-5与其序列相似性较高的序列聚类在同一枝,自展值均在80%以上,除210415-3自展值为82%之外,其余各枝均在90%以上,具有较高可信度;210415-2与其序列相似性最高的序列聚类分枝自展值仅为40%,且该序列只标明来源于双翅目。
2.4 蝇种鉴别判定
210415-1样本虽然在BLOD数据库中最佳匹配结果是绿蝇属,但在匹配前6位的结果中有4个是Melinda pusilla,在GenBank比对结果列表的前3位均为Melinda pusilla,且序列相似度均在99%以上。经查阅相关文献,Melinda pusilla实为Gymnodichosia pusilla(黄足裸变丽蝇)的同种异名[8],在GenBank中收录的Melinda pusilla序列在文献中的描述也同样为黄足裸变丽蝇[3]。通过比对形态学特征,210415-1形态符合黄足裸变丽蝇的描述[8],鉴定为黄足裸变丽蝇。至于在BOLD数据库匹配度最高的绿蝇属序列,应为序列上传者在蝇种鉴定中存在偏差。210415-2在BLOD数据库中无相对应的物种,在GenBank数据中最接近的序列基因相似度为93.88%,无具体物种信息描述,其次接近的是净妙蝇(Myospila lauta)CO Ⅰ基因序列,相似度仅为93.38%,另有点池蝇属(Spilogona)、须泉蝇属(Pegoplata)等CO Ⅰ基因序列与之相似度为90%以上,经形态学特征比对分析,无法具体鉴定其种类。210415-3、210415-4、210415-5号样本在BOLD条码数据库中有较好的匹配结果,序列相似度均高于98%,同时GenBank数据库中序列相似度均高于98%,结合形态学特征鉴定,可以确定与DNA条码初步结果完全符合。综上所述,对于某些非常见蝇种,在DNA条形码进行蝇种鉴定过程中,有必要结合BOLD和GenBank数据库进行交叉验证,对于提升鉴定结果的可靠性有一定帮助。
3 讨论
据世界卫生组织(WHO)数据显示,全球有80%的人口处于病媒生物传染病风险之中,由病媒生物传播的寄生虫、细菌、病毒所引发的传染病,导致全球每年有70多万人死亡,占全部传染病的17%以上[9]。因此,对于口岸病媒生物的监测和控制是病媒防制的核心内容。
口岸本底蝇类监测中捕获的蝇类多为常见种,但仍有少量是非常见种。非常见种蝇类鉴定工作,特别考验鉴定人员的分类学知识和鉴定经验。在样本鉴定时,可能遇到样本形态破损,发育时期差异、雌雄异型等不满足形态学鉴定要求的情况。此外,形态学鉴定过程容易受蝇类个体表型可塑性和遗传可变性干扰影响鉴定结果。而使用DNA条形码技术,则可以有效规避上述缺点,再加上标准化操作流程,使得鉴定人员更易操作,避免形态学鉴定过程中产生的人为误差。但是DNA条形码技术在应用中也存在一些问题:(1)DNA条形码数据库收录的某物种基因序列数量与该物种在自然环境中出现的频次成正相关,对于非常见蝇种使用DNA条形码鉴定时,最常出现的问题就是在数据库中比对不到对应的序列。本研究中使用的5只蝇,初步鉴定结果就有2只比对不到具体蝇种。但序列对比的结果能提示形态学鉴定的方向,缩小资料查找范围,提高鉴定效率。(2)数据库中收录的序列质量难以保证,这一点也与序列数量相关。出现频次较低的蝇类,本身就存在形态学鉴定困难的问题,数据库中的序列结果难以具体分类到种或者出现差错,由于数量的缺乏导致缺少数据间的相互印证。本研究中的210415-1样本在BOLD数据库中初步比对结果为绿蝇属,通过与GenBank数据库中序列的交叉验证,再经形态学比对最终才得以确定结果。
4 结语
本研究通过对宁波口岸本底监测捕获的蝇类进行DNA条形码鉴定,对DNA条形码技术在非常见蝇种鉴定中的应用进行探讨,认为在蝇类鉴定的实践应用中,有必要将形态学鉴定和DNA条形码技术有机结合,实现蝇类的快速准确鉴定,对于保障国门生物安全具有重要意义。
第一作者:刘永磊(1989—),男,汉族,山东临沂人,博士,高级农艺师,主要从事病媒生物种类鉴定及病原检测,E-mail: toxinx@163.com
通讯作者:吴薇(1971—),女,汉族,黑龙江穆棱人,硕士,副主任医师,主要从事医学媒介生物分类研究,E-mail: 71937549@qq.com
1. 宁波国际旅行卫生保健中心 宁波 315800
2. 梅山海关 宁波 315800
1. Ningbo International Travel Health Care Center, Ningbo 315800
2. Meishan Customs, Ningbo 315800
表1 宁波口岸监测5只蝇CO Ⅰ基因在两个数据库中的比对结果
Table 1 Comparison results of CO Ⅰ genes of five flies in two databases
样本编号 | BLOD数据库中最高相似度 (%) | GenBank数据库中最高相似度 (%) | DNA条形码初步结果 |
210415-1 | 100 | 99.84 | 绿蝇属Lucilia |
210415-2 | N/A | 93.88 | N/A |
210415-3 | 98.78 | 98.58 | 铜腹重毫蝇Dichaetomyia bibax |
210415-4 | 99.22 | 98.90 | 拟新月陪丽蝇Bellardia menechmoides |
210415-5 | 99.22 | 99.05 | 拟新月陪丽蝇Bellardia menechmoides |
注:N/A表示不适用(Not available) | |||
表2 宁波口岸监测5只蝇CO Ⅰ基因序列两两比对的遗传距离
Table 2 Genetic distance of CO Ⅰ gene sequences of five flies monitored at Ningbo port
编号 | 210415-1 | 210415-2 | 210415-3 | 210415-4 |
210415-1 | ||||
210415-2 | 0.127 | |||
210415-3 | 0.138 | 0.119 | ||
210415-4 | 0.127 | 0.131 | 0.156 | |
210415-5 | 0.127 | 0.131 | 0.156 | 0.000 |

图2 宁波口岸监测5只蝇及其近缘序列的系统发育树
Fig.2 Phylogenetic tree of five flies monitored at Ningbo port and their high similarity sequences
参考文献
[1]张瑞玲, 王晓旭, 张忠. DNA条形码技术在医学媒介生物鉴定中的应用[J]. 中国热带医学, 2017, 17(8): 831-837.
[2] Hebert P D, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society of London Series B: Biological Sciences, 2003, 270(1512): 313-321.
[3]邱德义, 廖俊蕾, 魏晓雅, 等. 基于DNA条形码丽蝇科49种常见种的分类鉴定[J]. 中国媒介生物学及控制杂志, 2017, 28(4): 322-331.
[4]吴薇, 夏德峰, 郑炜. 应用DNA条形码技术鉴定24种入境有瓣蝇类 [J]. 农业科学与技术:英文版, 2015(2): 235-241.
[5]杜鹃, 邓耀华, 张子龙,等. 上海口岸截获的粉蝇属DNA条形码鉴定及系统发育研究[J]. 寄生虫与医学昆虫学报, 2019, 26(1): 44-49.
[6] Folmer O, Black M, Wr H, et al. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates[J]. Molecular Marine Biology and Biotechnology, 1994, 3(5): 294-299.
[7] Ratnasingham S, Hebert P D. BOLD: The Barcode of Life Data System (http://www. barcodinglife. org)[J]. Molecular ecology notes, 2007, 7(3): 355-364.
[8]范滋德. 中国常见蝇类检索表[M]. 2版. 北京: 科学出版社, 1992: 516-517.
[9] World Health Organization & UNICEF. Global vector control response 2017-2030 [R]. Geneva: WHO, 2017.
(文章类别:CPST-A)
第3卷 第9期
2021年9月
Applied Research / 应用研究







