CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
茶树C4H基因的克隆及生物信息学分析
作者:汪文俊 刘青山 罗纪军 朱林 吴琼 谢婷 宗凯 余晓峰
汪文俊 刘青山 罗纪军 朱林 吴琼 谢婷 宗凯 余晓峰
Abstract To clone and verify the cinnamate 4-hydroxylase (C4H) gene in tea plant and predict the biological function of C4H gene. Extracting total RNA from fresh tea leaves, and the C4H gene of tea was cloned by RT-PCR. The conserved domain, physical and chemical properties, subcellular localization, transmembrane domain and signal peptide, homology and phylogeny of the protein encoded by C4H gene were analyzed by bioinformatics software. The results showed that the C4H gene of tea contained a complete open reading frame (ORF) of 1518 bp, encoding 505 amino acids; conserved domain analysis showed that the protein encoded by tea C4H gene contained a domain of P450 superfamily protein (PLN02394), with a molecular weight of 58.16 kDa and a theoretical isoelectric point of 9.29. It was a hydrophilic and unstable protein, localized in the endoplasmic reticulum, without signal peptide and transmembrane domain. The secondary structure of the protein was mainly a-helix and random coil. The C4H gene had the highest homology with oil tea at the amino acid level (92%). In this study, the C4H gene of tea was successfully cloned and its biological function was analyzed, which laid a preliminary theoretical foundation for further study on the function of C4H gene and the molecular mechanism of tea synthesis.
Keywords camellia sinensis; cinnamate-4-hydroxylase (C4H); bioinformatics analysis
茶树肉桂酸-4-羟基化酶基因是植物P450单加氧酶超家族基因之一,可以编码和翻译肉桂酸-4-羟基化酶[1]。该酶在苯丙烷生物合成途径中参与一步重要的氧化反应,即将反式肉桂酸转换为4-香豆酸[2]。植物体内许多重要的化合物都是经由苯丙烷次生代谢途径衍生而成的,如木质素、黄酮类、抗毒素和色素类,这些物质对于植物适应外界环境、抵御病虫害侵扰和逆境胁迫具有重要意义[3]。
据报道,许多植物中的C4H基因已经被克隆,如拟南芥(Arabidopsis thaliana)[4]、荸荠(Water-chestnut)[5]、龙血树( Dracaena cambodiana)[6]、毛竹(Phyllostachys edulis)[7]等。但关于茶树C4H 基因的研究报道较少,仅有姚胜波等[8]以舒早茶为材料,采用5'-RACE技术对茶树C4H基因进行克隆。另外,在NCBI数据库中检索茶树C4H基因 mRNA序列,较完整的只有2条(AY641731.2和KP990541.1)序列。本研究通过提取黄山柿大叶种茶树叶片的总RNA,利用RT-PCR技术克隆和验证了C4H基因序列,并通过生物信息学手段对肉桂酸-4-羟基化酶基因的理化性质、保守结构域、信号肽、跨膜结构、亚细胞定位、二级和三级结构等进行了预测分析,旨在进一步为肉桂酸-4-羟基化酶基因的功能和苯丙烷代谢途径的研究提供参考。
1 材料与方法
1.1 材料与试剂
茶树鲜叶采自黄山海关茶树种质资源圃,品种为柿大叶种(Camellia sinensis (L.) O. Ktze.),样品采集后迅速用液氮冷冻,后转移至-80℃冰箱保存。总RNA提取试剂盒和反转录试剂盒均购自天根生化试剂公司,RT-PCR扩增试剂购自上海近岸生物公司。其他有机溶剂均为国产分析纯。
1.2 仪器与设备
Eppendorf 5424/5424R离心机(Eppendorf公司);veriti PCR扩增仪(美国ABI公司);QIAxcel核酸电泳分析系统(德国QIAGEN公司)。
1.3 实验方法
1.3.1 茶树鲜叶总RNA提取
将从-80℃冰箱保存的鲜叶样品取出,恢复至室温,用不含RNA酶的研钵研磨鲜叶,按照总RNA提取试剂盒说明书方法提取鲜叶总RNA,提取结束立即用反转录试剂盒将RNA转录为可以长期保存的cDNA。
1.3.2 RT-PCR
参考NCBI数据库中茶树C4H基因(KP990541.1)序列,设计一对覆盖CDS编码区的特异性引物,C4H-F:5′-ATGGATCTTCTCCTCCTA GAGAAG-3′,C4H-R:5′-TCAGAACGATCTTG GTTTCAGAAC-3′。以cDNA为模板,扩增条件如下:95℃预变性30 s;95℃变性15 s,60℃退火30 s,72℃延伸100 s,该步骤进行30个循环;最后72℃延伸10 min。采用琼脂糖凝胶电泳分析扩增产物,切胶回收后连接到T表达载体上,筛选阳性克隆样品并送至滁州通用生物科技有限公司进行测序。
1.4 生物学信息学分析
采用BioXM 2.6软件对C4H基因编码蛋白质进行翻译和开放阅读框分析,用CCD(conserved domain database)在线软件分析保守结构域。对蛋白质序列进行理化性质(Protparam软件)、疏水性(ProtScale软件)、二级结构(SOPMA软件)和三级结构分析预测(SWISS-MODEL软件),同时利用在线分析工具进行跨膜结构域预测(TMHMM 2.0软件)、信号肽分析(SignalP 4.1软件)及亚细胞定位(Euk-mPLoc 2.0软件)。利用Mega 5.0软件进行同源蛋白的氨基酸多序列比对并构建系统进化树[9]。
2 结果与讨论
2.1 茶树C4H基因全长克隆
以茶树鲜叶为实验材料提取其总RNA,随后反转录得到cDNA模板,通过RT-PCR扩增出一条1500 bp左右的电泳条带。如图1所示,核酸电泳分析系统可见电泳图谱H02泳道有一条明显条带,与预期设计产物大小一致。将扩增产物进行测序,测序结果与Genebank:KP990541.1比对,仅有1个碱基差异,证实被扩增成功的确实为茶树C4H基因。
图1 茶树C4H基因RT-PCR扩增电泳图
Fig.1 RT-PCR amplification electropherogram of C4H gene in Camellia sinensis
2.2 茶树C4H基因ORF框和保守结构域分析
采用BioXM2.6软件查找茶树C4H基因的ORF框,发现具有一个长度为1518 bp的ORF框。经NCBI中CCD在线分析,茶树肉桂酸-4-羟基化酶氨基酸序列在第14和505个之间存在一个结构域(PLN02394),编码P450超家族成员。
2.3 茶树C4H蛋白理化性质分析
经Protparam软件计算,发现C4H基因编码505个氨基酸,氨基酸组成信息见表1,总蛋白质分子量为58.16 kDa,分子式为C2646H4189N719O723S16,蛋白质理论等电点为9.29。由表1可知,含量较高的氨基酸残基有Leu(L)、Val(V)、Lys(K)、Glu(E)、Arg(R)、Gly(G)、Ile(I),其中碱性氨基酸个数为80,占比15.84%;酸性氨基酸个数为107,占比21.19%。带正电荷的氨基酸残基(Total number of positively charged residues)(Arg+Lys)有70个,带负电荷的氨基酸残基(Total number of negatively charged residues)(Asp+Glu)有59个,不稳定指数(Instability index)为41.98(大于40为不稳定),表明该蛋白为不稳定状态。软件分析该蛋白质,其脂肪指数(Aliphatic index)为98.59,总平均亲水性(Grand average of hydropathicity,GRAVY)为-0.243,这表明此蛋白为亲水蛋白,且采用ProtScale软件Hydropath. / Kyte & Doolittle参数分析,该蛋白质在400~420位氨基酸序列之间有一个典型的亲水区域,见图3。
2.4 茶树C4H基因跨膜结构域预测、信号肽分析及亚细胞定位
采用TMHMM 2.0软件对茶树C4H基因跨膜结构域进行分析,结果见图4。横坐标轴表示被分析蛋白序列对应的氨基酸残基(1~505个),纵坐标轴为横轴上对应的每个氨基酸位于膜内侧、膜外侧和跨膜螺旋区的概率值。软件分析显示,预测的跨膜螺旋数量(Number of predicted TMHs)分析结果为0,表明没有跨膜螺旋结构。跨膜螺旋氨基酸残基数量的期望值(Exp number of AAs in TMHs)为10.77(小于18一般表示不含跨膜螺旋或信号肽)。蛋白质的前60个氨基酸中跨膜螺旋的氨基酸量的期望值(C4H Exp number, first 60 AAs )为10.23。位于膜细胞质侧的总概率(Total prob of N-in: N-term)为0.49。综合以上分析,该蛋白不含有跨膜结构域。
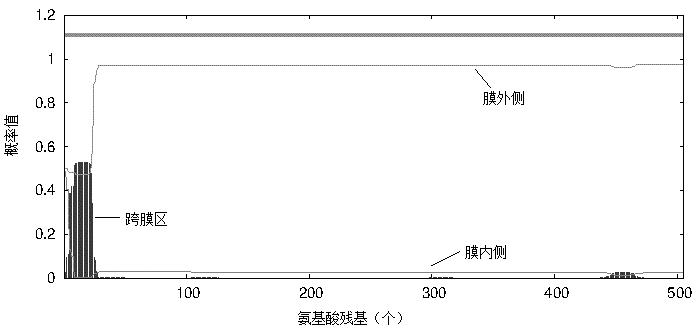
图4 茶树C4H蛋白跨膜结构域预测
Fig.4 Prediction of transmembrane domain of C4H protein in Camellia sinensis
采用SignalP 4.1软件对茶树C4H基因编码蛋白进行分析,软件默认分析前70个氨基酸,结果见图5。SignalP 4.1软件分析信号肽有3个重要的打分项,如图5,C-score(raw cleavage site score)显示是否为剪切位点,剪切位点之后的第一个氨基酸对应的是最高峰值(即成熟蛋白的第一个氨基酸残基);S-score(signal peptide score)显示对应氨基酸位置是否为信号肽区域,Y-score(combined cleavage site score)是C-score和S-score的几何平均数。图5左图为各值的变化趋势曲线,图5右图根据C值、S值和Y值等给出潜在的剪切位点,在信号肽(Signal peptide)列中结果为NO,表明该蛋白无信号肽。茶树C4H基因亚细胞定位采用Euk-mPLoc2.0软件,导入氨基酸序列后分析结果显示茶树C4H基因定位于内质网(Endoplasmic reticulum),见图6。
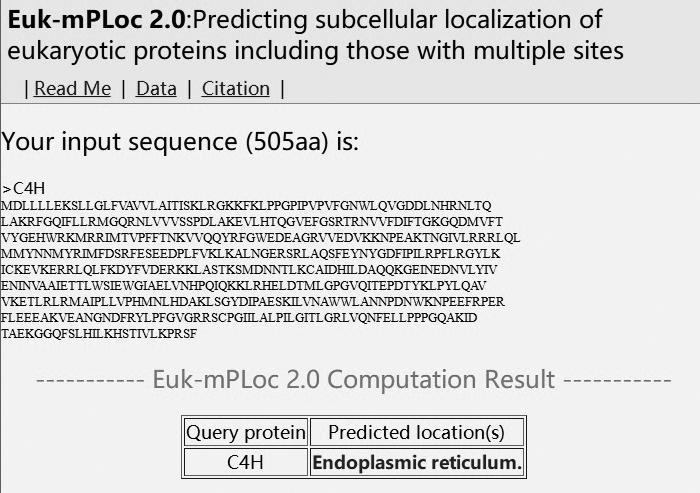
图6 茶树C4H蛋白亚细胞定位预测
Fig.6 Prediction of Subcellular localization of C4H protein in Camellia sinensis
2.5 茶树C4H蛋白的二级和三级结构预测
采用SOPMA软件在线分析茶树C4H蛋白,图7是软件分析后的蛋白质二级结构图,横向坐标是505个氨基酸,图7上部分是用不同长度竖线显示不同二级结构形式和所在位置,下部分是对应不同二级结构形式的峰图。经分析,C4H蛋白包含的二级结构中α-螺旋(Alpha helix)占45.74%,延伸链(Extended strand)占14.65%,无规卷曲(Random coil)占35.45%。此分析结果表明,茶树C4H蛋白的二级结构主要为α-螺旋和无规卷曲形式,占序列的80%以上。
采用SWISS-MODEL软件对茶树C4H蛋白三级结构进行预测,见图8。与数据库中三维模板匹配最佳的是来自Sorghum bicolor的6vby.1.A模板,序列相似率为78.11%。建模质量估值(GMQE)得分为0.91,接近1,表明建模结果较好。
图7 茶树C4H蛋白的二级分析
Fig.7 Secondary analysis of C4H protein in Camellia sinensis
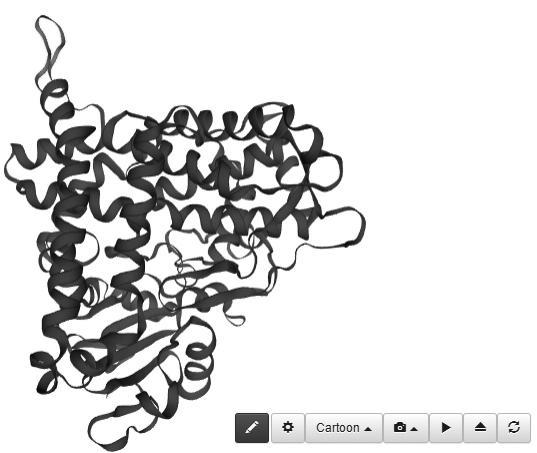
图8 茶树C4H蛋白三级结构图
Fig.8 Tertiary structure of C4H protein in Camellia sinensis
2.6 茶树C4H蛋白系统进化树构建
从NCBI中批量下载部分植物C4H蛋白的氨基酸序列,采用MEGA6.0软件中Clustal W工具进行序列比对,删除序列两端不能完全对齐的序列,进化树构建选择NJ法,test of Phylogeny选择Bootstrap method,重复次数为500;进化模型/方法选择p-distance,Gaps/missing Data Treatment 选择Pairwise deletion,构建的系统发育树见图9。系统发育树的树状结构处的数字为2个相邻植物C4H蛋白序列的相似度,可以看出C4H蛋白是一种高度保守的蛋白质,部分植物之间的氨基酸序列同源性超过90%。其中,与茶树(Camellia sinensis)亲缘关系最近的是油茶树(Camellia chekiangoleosa),序列相似度为92%。
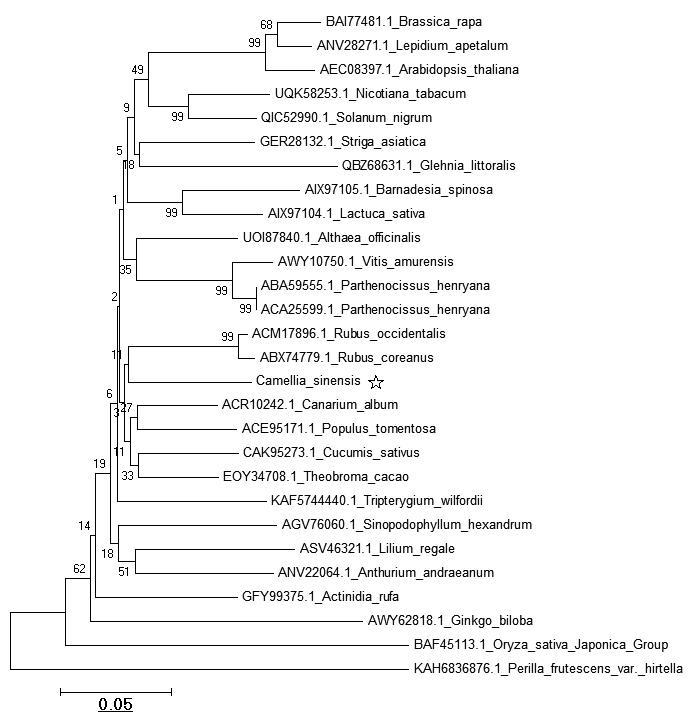
图9 茶树C4H蛋白系统进化树
Fig.9 Phylogenetic tree of C4H protein in Camellia sinensis
3 结论
茶树苯丙烷代谢途径是茶树次生代谢的重要途径,肉桂酸-4-羟基化酶是这个代谢途径的关键酶之一[10],其功能是催化合成p-香豆酸,为植物木质素、类黄酮等的合成提供前体物质[11]。
本研究克隆了茶树肉桂酸-4-羟基化酶(C4H)基因cDNA序列,包含一个1518 bp的ORF框,编码蛋白包含505个氨基酸,与姚胜波等[8]报道的一致。通过生物信息学方法初步分析了茶树C4H蛋白为亲水性蛋白质,且在400~420位氨基酸序列之间有一个典型的亲水区域。该蛋白不含有跨膜结构域,无信号肽结构,亚细胞定位将其定位于内质网,α-螺旋和无规卷曲是该蛋白二级结构的主要形式,利用SWISS-MODEL软件对其三级结构进行建模,预测其三级结构形态。通过进化树分析,发现C4H蛋白是高度保守的蛋白质,部分植物之间的氨基酸序列同源性超过90%,这与刘雄盛等[12]的研究结果一致。
本研究进一步克隆验证了茶树C4H基因序列,为研究分析C4H基因提供了更多丰富的基因信息,为下一步开展C4H基因调控茶树苯丙烷代谢途径的分子机理奠定了一定的基础。
参考文献
[1] 方志. 砀山酥梨肉桂酸4-羟化酶基因克隆及表达分析[D]. 合肥: 安徽农业大学, 2015: 5-6.
[2] 牛卉. 华细辛肉桂酸-4-羟基化酶基因的克隆、生物信息学及表达分析[D]. 上海: 上海交通大学, 2016: 3-4.
[3] 孙晓丹. 膜荚黄芪肉桂酸-4-羟化酶(C4H)基因克隆及植物表达载体的构建[D]. 延边: 延边大学, 2011: 6-8.
[4] 翟逸, 刘钦松, 石党委, 等. 拟南芥4-香豆酰-CoA连接酶基因的克隆与生物信息学分析[J].生物化工,2017, 3(3): 1-8.
[5] 宋慕波, 周伟政, 唐永胜, 等. 荸荠肉桂酸4-羟基化酶基因的克隆及其在鲜切荸荠黄化过程中的表达分析[J]. 基因组学与应用生物学, 2020, 39(2): 666-673.
[6] 梁惠桢, 朱家红, 戴好富, 等. 海南龙血树肉桂酸-4-羟基化酶基因(DcC4H)的克隆及表达分析[J]. 分子植物育种, 2018, 16(24): 7984-7989.
[7] 李广柱, 朱成磊, 杨克彬, 等. 毛竹C4H基因的鉴定及其表达模式分析[J]. 热带亚热带植物学报, 2022, 30(2): 151-160.
[8] 姚胜波, 王文钊, 李明卓, 等.茶树肉桂酸 4-羟基化酶基因的克隆及表达分析[J]. 茶叶科学, 2015, 35(1): 35-44.
[9] 梅菊芬, 徐德良, 汤茶琴, 等. 茶树CsLhcb4基因的电子克隆与生物信息学分析[J]. 江苏农业科学, 2018, 46(9): 51-55.
[10] 王树斌, 全雪丽, 具红光, 等. 膜荚黄芪肉桂酸-4-羟化酶(C4H)基因克隆与序列分析[J]. 延边大学农学学报, 2012, 34(4): 277-281.
[11] 刘声传, 鄢东海, 魏杰, 等. 茶树硒代半胱氨酸甲基转移酶基因生物学分析[J]. 西南农业学报, 2013, 26(6): 2221-2225.
[12] 刘雄盛, 尹国平, 肖玉菲, 等. 枫香C4H基因的克隆及生物信息学分析[J]. 热带农业科学, 2022, 42(6): 45-52.
1 50 100 150 200 250 300 350 400 450 500

图2 茶树C4H蛋白保守结构域分析
Fig.2 Analysis of conserved domain of C4H protein in Camellia sinensis
表1 茶树C4H蛋白的氨基酸组成
Table 1 Amino acid composition of C4H protein in Camellia sinensis
氨基酸种类 | 个数 | 占比 (%) |
Ala (A) | 25 | 4.95 |
Cys (C) | 3 | 0.59 |
Asp (D) | 24 | 4.75 |
Glu (E) | 35 | 6.93 |
Phe (F) | 27 | 5.35 |
Gly (G) | 32 | 6.34 |
His (H) | 10 | 1.98 |
Ile (I) | 31 | 6.14 |
Lys (K) | 36 | 7.13 |
Leu (L) | 62 | 12.28 |
Met (M) | 13 | 2.57 |
Asn (N) | 28 | 5.54 |
Pro (P) | 28 | 5.54 |
Gln (Q) | 20 | 3.96 |
Arg (R) | 34 | 6.73 |
Ser (S) | 18 | 3.56 |
Thr (T) | 20 | 3.96 |
Val (V) | 38 | 7.52 |
Trp (W) | 8 | 1.58 |
Tyr (Y) | 13 | 2.57 |
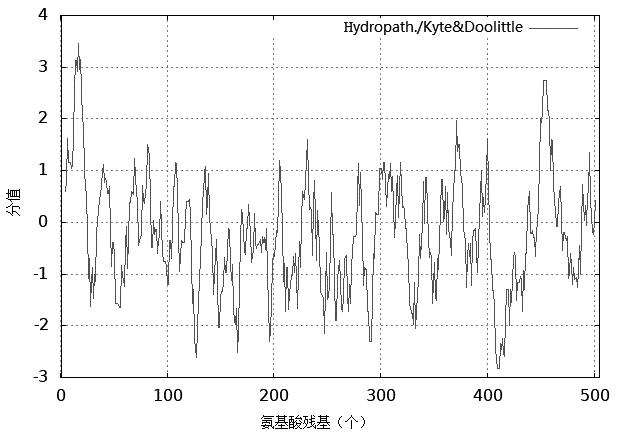
图3 茶树C4H蛋白亲水性预测图
Fig.3 Hydrophilicity prediction map of C4H protein in Camellia sinensis
图5 茶树C4H蛋白信号肽预测
Fig.5 Prediction of signal peptide of C4H protein in Camellia sinensis








