CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
CO Ⅰ和16S rRNA基因条形码在海马物种鉴定中的应用
作者:曾韵颖 陈信忠 朱黄鑫 郭书林 龚艳清 徐淑菲 叶鹏 孔繁德
曾韵颖 陈信忠 朱黄鑫 郭书林 龚艳清 徐淑菲 叶鹏 孔繁德
海马是我国传统的名贵中药材,具有补肾壮阳、活血止痛等功效[1]。Lourie等[2]根据形态学、遗传学和地理学等信息确认已知海马属包括41种有效种,其中多种野生海马已成为濒危物种。2019年,世界自然保护联盟(International Union for Conservation of Nature,IUCN)将海马属所有物种列入濒危物种红色名录,实施严格管制,禁止国际贸易。海马也被我国列为国家二级保护动物,但近年来走私海马的案例越来越多[3]。依据《濒危野生动植物种国际贸易公约》(CITES),海关工作人员需要快速准确地识别濒危海马物种。然而,由于海马种类较多,亲缘关系和形态特征相似,不易区分,给海关监管工作带来了一定的挑战,因此,建立快速准确的海马鉴定技术对打击海马走私、保护海马资源具有重要意义。
目前海马鉴定方法主要包括形态学鉴定和分子生物学鉴定。海马的形态学鉴定方法主要依据外部形态、骨骼或粉末的显微特征等,鉴定者需要具备专业知识,了解海马的形态特征[4-8]。但因为海马的形态十分相似,在贮存、加工时体色和表面纹理等容易发生变化,外观差异不显著,鉴别难度较大。与形态学鉴定方法相比,DNA条形码技术[9-14]、多重PCR[15]、随机扩增多态DNA(Randomly amplified polymorphic DNA,RAPD)技术[16]、基因组超保守元件(Ultraconserved elements,UCEs)分析[17]等分子生物学鉴定方法不受加工方法、产品形态的影响,鉴别结果更准确可靠。尤其是DNA条形码技术具有快速、简便、应用广泛的特点,已经被越来越多地应用于海马分类中。该技术是利用生物体DNA中一段保守片段对物种进行准确鉴定的新技术[18],其利用通用引物扩增一段靶基因的DNA序列,将序列在BOLD(The Barcode of Life Data System)的公共DNA条形码数据库或NCBI(National Center for Biotechnology Information)GenBank数据库中进行分析比对,从而实现物种鉴定。最常用的DNA条形码为线粒体COⅠ、16S rRNA或Cytb基因。Hou等[10]用COⅠ基因在中国3个大型中药市场鉴定了9种海马物种,所有种间遗传变异>2%,且种间平均遗传距离比种内平均遗传距离大17倍。蒋超等[5]用形态学和COⅠ基因相结合的方法,鉴定出23种市售海马物种,其中13种可通过COⅠ序列进行有效鉴定,另10种结合形态学后可进一步区分。Wang等[11]根据COⅠ基因序列和形态学对来自中国4大医药市场的64个海马干样品进行调查,鉴定出13种海马物种,其中11种可通过COⅠ序列有效鉴定,另外2种结合形态学后可进一步区分。Teske等[12]根据核RP1和醛缩酶基因,以及线粒体16S rRNA和Cytb基因序列对海马属32个物种进行分类研究,并确定它们的系统发育关系。Lopez等[13]采用基因组微卫星序列和16S rDNA序列有效鉴别了西班牙海岸的欧洲海马和浅黄海马。Sanders等[14]利用海马Cytb基因,对美国市场上56个海马样品的物种和种群水平进行了分析,其中53个样品被准确鉴定为5种海马。本研究选取COⅠ基因和16S rRNA基因作为DNA条形码的靶基因,建立快速有效的海马物种鉴定方法,以期为打击海马走私提供有力的技术支持。
1 材料与方法
1.1 实验材料
实验所用的6种海马样品为三斑海马(Hippoca mpus trimaculatus)、棘海马(H. spinosissimus)、管海马(H. kuda)、膨腹海马(H. abdominalis)、浅黄海马(H. guttulatus)和克氏海马(H. kelloggi)。其中,三斑海马采自福建东山,棘海马、管海马购自浙江磐安江南药镇,膨腹海马、浅黄海马、克氏海马为海关截获样品。以上样品均经形态学和GB/T 35918—2018《动物制品中动物源性检测基因条码技术 Sanger测序法》确认。
1.2 实验仪器和试剂
动物组织基因组提取试剂盒(美国Omega BIO-TEK公司),台式冷冻离心机(美国Thermo公司),Veriti PCR仪(美国ABI公司),毛细管电泳仪(台湾光鼎公司)。
1.3 实验方法
1.3.1 核酸提取
用洁净解剖刀取干海马肌肉约50 mg,按照试剂盒方法提取DNA。提取的DNA置于-20℃冰箱备用。
1.3.2 PCR引物
海马COⅠ基因通用引物参考Ward等[19]报告的方法,上游引物采用FishF1:5′-TCAACCAACCACAAAGACATTGGCAC-3′,下游引物采用FishR1:5′-TAGACTTCTGGGTGGCCAAAGAATCA-3′,目标片段长约655 bp;16S rRNA基因通用引物参考Palumbi[20]报告的方法,上游引物采用16Sar-5:5′-CGCCTGTTTATCAAAAACAT-3′,下游引物采用16Sbr-3:5′-CCGGTCTGAACTCAGATCACGT-3′,目标片段长约572 bp。
1.3.3 PCR扩增及电泳
采用25 μL反应体系:上下游引物(10 μmol/L)各1.25 μL,2×Taq PCR MasterMix预混液12.5 μL,DNA模板5 μL,超纯水5 μL。COⅠ基因扩增程序:首先94℃预变性5 min;然后94℃ 30 s、51℃ 30 s、72℃ 30 s,40个循环;最后72℃ 10 min。16S rRNA基因扩增程序:首先94℃预变性2 min;然后94℃ 30 s、52℃ 40 s、72℃ 1 min,35个循环;最后72℃ 10 min。使用毛细管电泳仪对扩增产物进行检测。
1.3.4 测序
扩增产物纯化后,委托生工生物工程(上海)股份有限公司使用ABI3730XL基因测序仪进行测序。
1.3.5 序列比对及分析
使用SeqMan软件对测序结果进行拼接、校对,并删除两端引物序列。将处理过的COⅠ序列分别在BOLD数据库和GenBank数据库进行比对鉴定;16S rRNA序列在GenBank数据库进行比对鉴定。
1.3.6 系统发育分析
根据测序比对结果,从GenBank数据库下载其他已知海马物种的COⅠ参考序列和16S rRNA参考序列。将6种海马样品序列和下载的COⅠ参考序列、16S rRNA参考序列,通过MEGA11软件进行多重序列比对,用p-distance的算法构建邻接法系统发育树,Bootstrap自展值设为1000次,据此验证选取的DNA条形码的准确性。
2 结果与分析
2.1 COⅠ基因和16S rRNA基因扩增结果
6种海马样品PCR扩增后电泳结果如图1所示。所有海马均扩增出目的基因片段,其中COⅠ基因目的片段长度约700 bp,16S rRNA基因目的片段约610 bp。
2.2 序列分析和鉴定结果
6种海马样品的COⅠ基因和16S rRNA基因扩增产物均获得良好的测序结果。所有基因序列已提交GenBank数据库,其收录序列号见表1。
6种海马的COⅠ基因和16S rRNA基因的序列比对和物种鉴定结果见表1。COⅠ序列比对结果显示,三斑海马、棘海马、膨腹海马、浅黄海马、克氏海马和管海马6种海马样品的COⅠ序列在BOLD数据库和GenBank数据库均能得到准确鉴定,且鉴定结果一致,物种序列相似度>99.5%。
16S rRNA序列比对结果显示,三斑海马、棘海马、膨腹海马、克氏海马和管海马5种海马样品在GenBank数据库能够得到准确鉴定,物种序列相似度>99.5%。因GenBank数据库尚无浅黄海马16S rRNA基因参考序列,因此浅黄海马无法获得有效鉴定结果。
研究表明,物种间线粒体基因种内遗传距离大部分低于2%,而种间遗传变异一般大于2%[21-22]。因此,通常认为同一物种的基因序列同源性应大于98%。本研究在进行序列比对时,在相似度≥98%的范围内选取相似度最高的判定为同一海马物种。
本研究运用上述方法对实验室收集的67个样品进行了鉴定,其中,三斑海马34个、棘海马19个、克氏海马5个、膨腹海马3个、管海马3个和浅黄海马3个,在GenBank数据库序列相似度均>99.5%。
2.3 系统发育分析
2.3.1 基于COⅠ基因序列的海马系统发育分析
根据6种海马样品和GenBank数据库已经收录的另外26种海马COⅠ基因序列构建了N-J系统发育树,如图2所示,其遗传距离见表2。
该系统发育树和遗传距离表明,海马基于COⅠ基因序列的平均遗传距离为0.122,非洲豆丁海马(H. nalu)和日本海马(H. mohnikei)的遗传距离最大,为0.256。有23种海马与其他海马物种的种间遗传距离均大于0.02,可用COⅠ基因条形码进行有效鉴定。其余9种海马的种间遗传距离很小。其中昆士兰海马(H. queenslandicus)和棘海马(H. spinosissimus)的遗传距离最小,为0.003,实际上在IUCN濒危物种红色名录中被认为是同物异名。Hou等[23]测定了昆士兰海马线粒体基因全序列,也表明与棘海马的亲缘关系非常近。
南非海马(H. capensis)、棕海马(H. fuscus)、西非海马(H. algiricus)和吻海马(H. reidi)4种海马的亲缘关系很近,窄腹海马(H. angustus)、东方多棘海马(H. hendriki)、西澳海马(H. subelo ngatus)3种海马的亲缘关系也很近,其中南非海马和棕海马的遗传距离为0.011,西非海马和吻海马的遗传距离为0.012,南非海马和吻海马的遗传距离为0.018,窄腹海马和东方多棘海马的遗传距离为0.012,东方多棘海马和西澳海马的遗传距离为0.014,这些海马无法依据COⅠ基因条码进行有效鉴别。
近年来陆续发现的小型豆丁海马包括丹尼斯豆丁海马(H. denise)、彭氏豆丁海马(H. pontohi)、巴氏豆丁海马(H. bargibanti)和非洲豆丁海马(H. nalu)聚成一支,但遗传距离均大于0.05。因此可以确认这些海马虽然形态相似,但属不同的物种。
2.3.2 基于16S rRNA基因序列的海马系统发育分析
根据6种海马样品和GenBank数据库已收录的另外24种海马16S rRNA基因序列构建了N-J系统发育树(图3),其遗传距离见表3。
该系统发育树及其遗传距离表明,海马基于16S rRNA基因序列的平均遗传距离为0.082,西澳海马和丹尼斯豆丁海马的遗传距离最大,为0.166。有17种海马与其他海马种间遗传距离>0.02,可用16S rRNA基因条形码进行有效鉴定。其余13种海马的种间遗传距离<0.02,表明种间序列差异很小,仅用16S rRNA基因条形码难以区分。其中,南非海马和棕海马序列完全一致。
管海马(H. kuda)、南非海马、棕海马、西非海马、吻海马、夏威夷海马(H. fisheri)和太平洋海马(H. ingens)7种海马组成的管海马复合体亲缘关系很近,彼此间的平均遗传距离仅为0.012。此外,昆士兰海马和棘海马的遗传距离仅为0.003,虎尾海马(H. comes)和西澳海马遗传距离只有0.014,直立海马(H. erectus)和欧洲海马(H. hippocampus)的遗传距离只有0.019,也无法依据16S rRNA基因条码进行有效鉴别。
管海马复合体在遗传上密切相关,得到一些研究的支持[24-26]。Lourie等[2]的研究表明,它们在冠的形态特征上有一些细微差异,且在地理上有一定的隔离,如管海马被发现于印度—太平洋,西非海马来自西非海岸,吻海马分布在美洲东海岸,太平洋海马则生活在东太平洋。
2.3.3 COⅠ和16S rRNA基因的分辨率比较
COⅠ基因和16S rRNA基因的系统发育树分支拓扑结构总体上趋于一致,进一步验证了DNA条形码鉴定结果的准确性。COⅠ基因比16S rRNA基因更大的平均遗传距离表明其在海马物种鉴定上具有更高的分辨率。其中,对亲缘关系较近的南非海马、棕海马、西非海马、吻海马、管海马、太平洋海马和夏威夷海马这7种海马组成的管海马复合体,16S rRNA基因均无法区别,COⅠ基因能有效分辨其中的3种(管海马、太平洋海马和夏威夷海马);对16S rRNA基因无法区分的直立海马和欧洲海马、虎尾海马和西澳海马,COⅠ基因均可分辨。
2.4 海马物种鉴定方案
COⅠ基因和16S rRNA基因对海马物种鉴定结果表明,DNA条形码技术可以对大部分海马物种进行准确的鉴定,特别是进化速度较快的COⅠ基因具有更高的种间分辨率。但目前海马DNA条形码数据库的基因条码信息不够完整,部分海马物种尚无相关的基因信息,或基因条码数量很少,在一定程度上限制了DNA条形码技术的应用。本研究已将GenBank数据库缺少的浅黄海马16S rRNA基因序列上传至GenBank数据库。基因库中还有少数基因信息因各种原因可能存在谬误,也可能影响鉴定结果的准确性。本研究推荐优先采用COⅠ基因和16S rRNA基因对海马物种进行鉴定,对因亲缘关系较近无法用这两种基因区分的海马,建议采用其他线粒体基因进一步分析和鉴定。目前Genbank等数据库中有关海马其他线粒体基因的信息较少,涵盖的物种数量也比较有限,主要以Cytb和ND4基因序列为主。遗传距离分析表明,对COⅠ基因无法区分的几种海马Cytb和ND4基因也无法分辨或无相关序列数据。
对于现有DNA条形码技术难以区分的海马物种,建议结合形态特征和地理分布信息进行分析。因亲缘关系太近,用COⅠ基因无法有效鉴定的海马主要分成3组:第1组为西非海马、吻海马、南非海马和棕海马,这4种海马属于管海马复合体成员,建议先用形态特征进行区分,蒋超等[5]利用冠型、颊棘、眼棘、吻部形态及体色、斑纹、体型大小等核心形态特征,对棕海马、西非海马、吻海马、管海马、太平洋海马和南非海马6种管海马复合体成员进行了鉴定,对形态上也相似的再结合样品地理来源进行分析;第2组为昆士兰海马和棘海马,这2种海马在IUCN濒危物种红色名录中被标注为同物异名;第3组是西澳海马、窄腹海马、东方多棘海马,其中,窄腹海马和东方多棘海马在IUCN濒危物种红色名录中被标注为同物异名,西澳海马和窄腹海马则可通过形态学进行区分,前者有一个中等大小的刺冠,体表有棘刺,后者刺冠高而圆,体平无棘[2]。
3 结论
基于COⅠ基因和16S rRNA基因的DNA条形码技术可有效应用于大部分海马物种的鉴定,2个基因对同一海马物种的鉴定结果基本一致,其中COⅠ基因具有更高的种间分辨率,推荐优先选用。对于亲缘关系较近的海马物种,应结合形态特征和地理分布信息进一步区分。本研究提供的海马物种鉴定方案可对海马物种进行准确快速的鉴定,为打击海马走私、保护海马资源提供技术支持。下一步,需重点研究基因信息有限的部分海马物种和亲缘关系太近的海马物种复合体,持续完善和修正BOLD和NCBI数据库的相关序列信息,以提高其适用性和准确性。此外,需加强对其他靶基因和多基因联合DNA条形码的研究,探讨基因组超保守元件分析等新兴分子生物学技术在海马物种鉴定上的应用。
参考文献
[1]国家药典委员会. 中华人民共和国药典(一部)[M]. 北京: 中国医药科技出版社, 2015.
[2] Lourie S A, Pollom R A, Foster S J. A global revision of the seahorses Hippocampus Rafnesque 1810 (Actinopterygii: Syngnathiformes): taxonomy and biogeography with recommendations for further research[J]. Zootax, 2016(4146): 1-66.
[3]陈信忠,曾韵颖.海马种类及其鉴定技术分析[J].中国口岸科学技术, 2020(12): 48-54.
[4] Lourie S A, Foster S J, Cooper E C, et al. A Guide to the Identification of Seahorses[M]. Washington, D C: University of British Columbia and World Wildlife Fund, 2004.
[5]蒋超,刘富艳,金艳,等.基于形态和DNA序列分析的海马类药材商品基原调查[J]. 中国中药杂志, 2018, 43(23): 4553-4561.
[6]何旭辉, 张成中, 黄宝康, 等. 以微观性状鉴别为基础的海马分类研究[J]. 中国海洋药物, 2021, 40(4): 18-26.
[7]梁亮堂. 透明化骨骼染色鉴定海马种类的方法[J].中国水产, 2019(5): 88-90.
[8]康延国, 朱丽娃, 苑冬敏, 等. 海马类药材粉末的显微比较鉴别[J].中药材, 2006(3): 224-228.
[9]胡嵘, 杜鹤, 崔丽娜, 等. 海马、海龙基于COⅠ条形码的DNA分子鉴定[J].吉林中医药, 2012, 32(3): 272-273.
[10] Hou F, Wen L, Peng C, et al. Identification of marine traditional Chinese medicine dried seahorses in the traditional Chinese medicine market using DNA barcoding[J]. Mitochondrial DNA A, 2016, 29(1): 107-112.
[11] Wang X, Zhong H, Guo J, et al. Morphology and molecular identification of the zoological origin of medicinal seahorses in Chinese herbal markets[J]. Mitochondrial DNA Part A, 2020, 31(8): 335-345.
[12] Teske P R, Cherry M I, Matthee C A. The evolutionary history of seahorses (Syngnathidae: Hippocampus): molecular data suggest a West Pacific origin and two invasions of the Atlantic Ocean[J]. Molecular Phylogenetics and Evolution, 2004, 30(2): 273-286.
[13] Lopez A, Vera M, Otero-Ferrer F, et al. Species identification and genetic structure of threatened seahorses in Gran Canaria Island (Spain) using mitochondrial and microsatellite markers[J]. Conserv Genet, 2010(11): 2431-2436.
[14] Sanders J G, Jennifer E C, Harris G F, et al. The tip of the tail: molecular identification of seahorses for sale in apothecary shops and curio stores in California[J]. Conserv Genet, 2008, 9(1): 65-71.
[15]刘富艳, 袁媛, 金艳, 等. 多重PCR同时鉴别5种药用海马[J]. 中国中药杂志, 2018, 43(23): 4562-4568.
[16]董世雄,李卫东,王沛政, 等. 海马种类鉴定及群体遗传多样性分析[J]. 技术与市场, 2017, 24(12): 126-128.
[17] Stille J, Short G, Hamilton H, et al. Phylogenomic analysis of Syngnathidae reveals novel relationships, origins of endemic diversity and variable diversifcation rates[J]. BMC Biology, 2022, 20(1): 75.
[18] Hebert P D, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes[J]. The Royal Society, 2003(270): 313-321.
[19] Ward R D, Zemlak T S, Innes B H, et al. DNA barcoding Australia's fish species[J]. Philosophical transactions of the Royal Society of London. Series B, Biological sciences, 2005, 360(1462): 1847-1857.
[20] Palumbi S R. Nucleic acids Ⅱ: the polymerase chain reaction[M]. Sunderl and: Sinauer & Associates Inc., 1996: 205-247.
[21] Hebert P D, Ratnasingham S, deWaard J R. Barcoding animal life: cytochrome coxidase subunit 1 divergences among closely related species[J]. The Royal Society, 2003, 270(S1): 96-99.
[22] Ratnasingham S, Hebert P D. A DNA-based registry for all animal species: the barcode index number (BIN) system[J]. Plos One, 2013, 8(7): e66213. DOI: 10.1371/journal.pone.0066213.
[23] Hou F, Wang S, Peng C, et al. The complete mitochondrial DNA genome of the Queensland seahorse Hippocampus queenslandicus Horne, 2001 (Gasterosteiformes: Syngnathidae)[J]. Mitochondrial DNA B Resour, 2016, 1(1): 923-924.
[24] Casey S P, Hall H J, Stanley H F, et al. The origin and evolution of seahorses (genus Hippocampus): a phylogenetic study using the cytochrome b gene of mitochondrial DNA. [J]. Molecular Phylogenetics and Evolution, 2004, 30(2): 261-272.
[25] Teske, P R, Hamilton H, Palsboll P J, et al. Molecular evidence for long-distance colonization in an Indo-Pacific seahorse lineage[J]. Marine Ecology Progress Series, 2005, 286: 249-260.
[26] Silveira R B, Siccha-Ramirez R, Silva J R, et al. Morphological and molecular evidence for the occurrence of three Hippocampus species (Teleostei: Syngnathidae) in Brazil[J]. Zootaxa, 2014, 3861(4): 317-332.

左:COⅠ基因扩增片段电泳结果; 右: 16S rRNA基因扩增片段电泳结果; M: Marker(20~1000 bp); 1: 三斑海马(H. trimaculatus); 2: 棘海马(H. spinosissimus); 3: 膨腹海马(H. abdominalis); 4: 浅黄海马(H. guttulatus); 5: 克氏海马(H. kelloggi); 6: 管海马(H. kuda)
图1 海马样品COⅠ和16S rRNA基因扩增结果
Fig.1 PCR amplification of COⅠ and 16S rRNA gene of hippocampus
表1 6种海马样品COⅠ基因和16S rRNA基因的物种鉴定结果
Table 1 Species identification results of six hippocampus samples based on COⅠ Gene and 16S rRNA Gene
序号 | 海马物种名称 | COⅠ基因 | 16S rRNA基因 | ||||
BOLD数据库 | GenBank数据库 | GenBank | GenBank数据库 | GenBank | |||
序列相似度 | 序列相似度 | 收录序列号 | 序列相似度 | 收录序列号 | |||
1 | 三斑海马 (H. trimaculatus) | H. Trimaculatus (99.85%) | H. Trimaculatus (99.85%) | OP113680 | H. Trimaculatus (100%) | OP108818 | |
2 | 棘海马 (H. spinosissimus) | H. spinosissimus (100%) | H. spinosissimus (100%) | OP113681 | H. spinosissimus (99.83%) | OP108820 | |
3 | 膨腹海马 (H. abdominalis) | H. abdominalis (100%) | H. abdominalis (99.70%) | OP113792 | H. abdominalis (99.83%) | OP108848 | |
4 | 浅黄海马 (H. guttulatus) | H. guttulatus (100%) | H. guttulatus (100%) | OP113791 | — | OP113131 | |
5 | 克氏海马 (H. kelloggi) | H. kelloggi (100%) | H. kelloggi (99.70%) | OP113793 | H. kelloggi (99.83%) | OP113530 | |
6 | 管海马 (H. kuda) | H. kuda (99.84%) | H. kuda (99.69%) | OP113687 | H. kuda (100%) | OP108825 | |
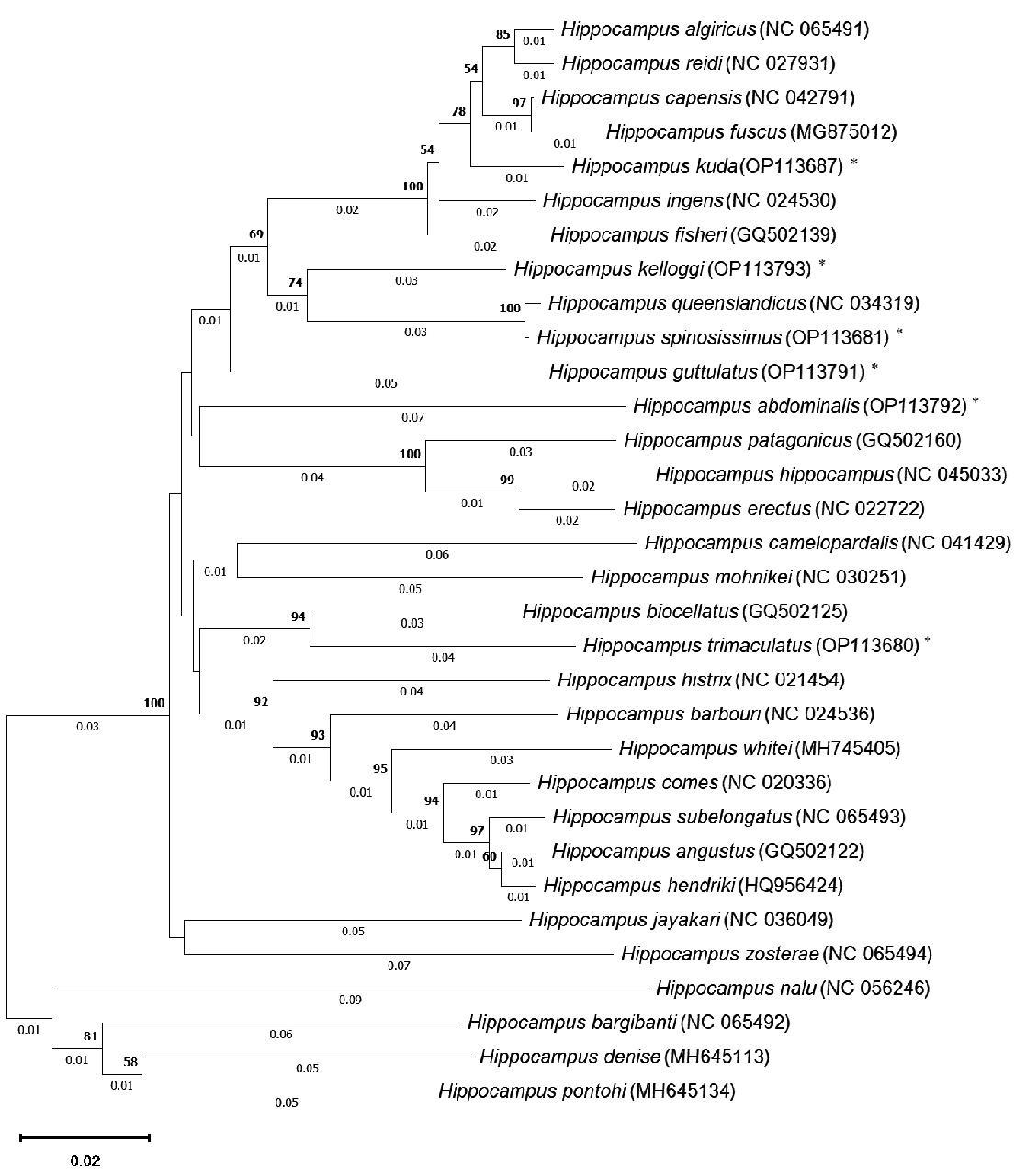
*为本研究测定的基因序列
图2 基于COⅠ基因的海马系统发育树
Fig.2 Phylogenetic tree of hippocampus based on COⅠ gene
表2 32种海马COⅠ基因的遗传距离
Table 2 Genetic distance of the COⅠ gene in 32 hippocampus species

*为本研究测定的基因序列
图3 基于16S rRNA基因的海马系统发育树
Fig.3 Phylogenetic tree of hippocampus based on 16S rRNA gene
表3 30种海马16S rRNA基因的遗传距离
Table 3 Genetic distance of the 16S rRNA gene in 30 hippocampus species







