CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
转基因大豆生信分析平台的构建及口岸初步应用
作者:杜鹃 刘誉 刘振宇 马鑫淼 李浩辰 马毅 董志珍
杜鹃 刘誉 刘振宇 马鑫淼 李浩辰 马毅 董志珍
Abstract Besed on the characteristics and practical needs of customs operations, this research uses technologies such as computational storage server integration and data mining applications, studies genetically modified (GM) soybean databases and data analysis methods, and establishes a visually-operated GM soybean bioinformatics analysis platform. Based on the long-term collection and accumulation of the genetically modified plant genome database, a pan-genome reference gene of soybean is constructed. Combining the standard samples of various soybean strains, the sequencing and sequence analysis of soybean samples are performed. After screening and assembling the suspicious sequences, the statistical threshold parameters are used to determine whether the samples are genetically modified varieties/strains, thereby achieving efficient identification of GM soybeans through gene sequencing data. Relying on distributed cloud computing technology to build a GM soybean analysis platform, it forms a gene sequencing data analysis method, and improves the speed and accuracy of GM soybean identification. A GM soybean bioinformatics analysis platform with advantages of visualization, simple operation, powerful functions, strong data security and information confidentiality has been built. This analysis platform simplifies the operation of GM soybean data analysis, with visually arranged process facilitating the data analysis operation of detection technicians. With only one-click, GM soybean data analysis can be realized, and illegal sequences of GM soybeans can be quickly and accurately identified.
Keywords genetically modified soybean; analysis platform; bioinformatics analysis; genome database
生信分析是对生物学数据进行统计分析和模式识别的过程,可帮助科研工作者更好地理解生物学数据,提高科研效率。转基因大豆生信分析是通过筛选测序序列与大豆泛基因组的相似性得到转基因序列,并通过对比转基因序列与合法转基因序列的一致性来判断转基因序列的来源。
近年来,国外商业化种植的转基因大豆品系众多,与我国审批允许进境的转基因品系数量差距很大。海关作为国门生物安全的第一道防线,在口岸转基因业务的安全监管和技术保障中面临着越来越大的压力,需要构建适合自身业务特点和口岸实际需求的生物信息学数据分析平台及相关数据库[1]。目前,日常工作中调取的数据资源多来自互联网开源数据库,生信分析也基本在线上完成。但上述开源数据库和在线分析网站大多由国外有关机构开发和维护,易成为国际信息技术壁垒的关键因素。加之大数据时代,数据安全已成为总体国家安全观的重要组成部分,对开源数据库和线上分析网站的依赖也容易构成数据信息安全隐患。再者,海关业务中涉及的生物信息数据量庞大且保密性要求高,其分析、储存、追踪、管理以及结果分析的审核查询等需要基于一个完整、系统且海关自有的基因组数据库和智能化数据分析平台[2-3]。因此,海关建设自己的高通量测序及数据分析平台意义重大,将提高相关业务领域的工作效能和业务水平。
本研究基于海关业务数据探索开发建立有针对性的算法和模型,构建海关专有的基因组信息数据库,旨在实现对多源基因组数据的收集和统一管理,为海关鉴别非目录下的转基因大豆及防控非法转基因贸易提供有力的研判依据和技术支撑。
1 材料与方法
1.1 平台架构设计
转基因大豆分析平台是基于系统资源管理(system resource manage,SRM)分布式云计算技术栈搭建的。CentOS7作为底层操作系统提供基础的运行环境,使用Docker[4]容器技术搭建Web应用服务的运行环境,使用PostgreSQL[5]对象-关系型数据库进行数据存储;前端以Vue.js[6]为展现框架,逻辑处理以Go语言[7]作为技术栈进行实现。基因组检索系统主要基于Vue.js和Go技术搭建,高通量数据分析流程以国际通用的WDL[8]语言为基础进行编排和搭建,调用GeneScan[9]等软件完成对转基因标签序列的识别判定。数据库总体架构设计包括应用层、数据层、底层存储等,如图1所示。
应用层采用业界成熟的Vue.JS+SpringBoot框架以微服务的形式进行产品模块构建,支持用户在Web端进行可视化操作,Web端设计的功能模块主要包含转基因标签合法性分析资源调度模块、操作动态显示模块、综合应用管理模块、流程分析编辑器等;数据层通过ETL数据清洗以及独有的分词检索技术,保证前端检索结果准确度;底层存储采用HDFS文件系统提高数据吞吐量,加速应用层的数据检索请求。
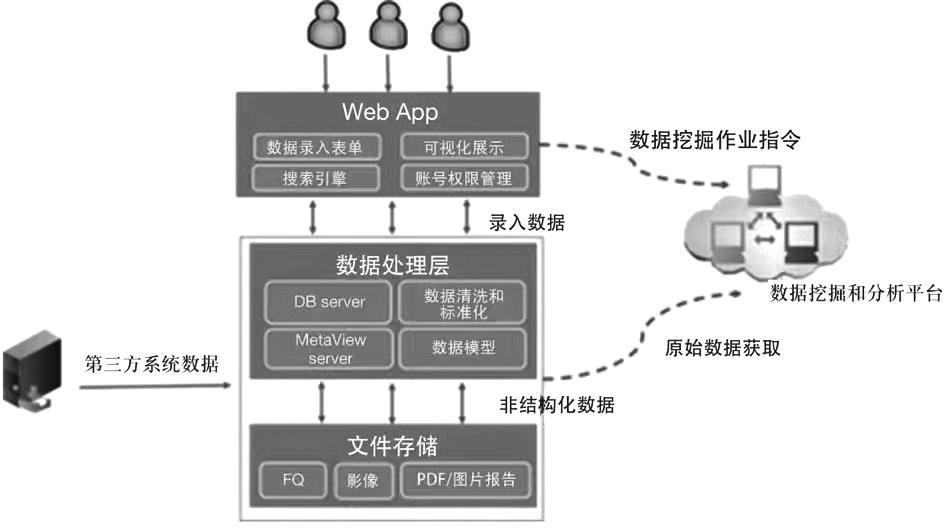
图1 分析平台数据流架构设计图
Fig.1 Design diagram of data flow architecture of the analysis platform
以转基因物种鉴定为例,使用人员可通过Web浏览器登录转基因大豆生信分析平台,导入待鉴定样本测序数据,勾选数据分析流程,对数据进行“一键式”组装及比对分析,最终获取分析报告以供鉴定参考,如图2所示。

图2 基于大豆泛基因组构建非靶向转基因大豆数据
分析流程
Fig.2 Construction of a non-targeted transgenic soybean data analysis process based on soybean
1.2 软硬件环境
基于植物泛基因组构建非靶向转基因鉴别应用算法,开发生物信息分析流程及计算资源优化与重构、应用卷积神经网络算法等人工智能技术、数据存储和聚合查询技术、基因组数据可视化技术、生物信息计算分析引擎等关键技术,按照数字化、模型化、软件化的技术思路,建立转基因大豆标签数据库需要的软硬件环境,见表1和表2。
表1 软件环境
Table 1 Software environment
系统管理软件/平台名称 | 版本 | 主要功能 |
PostgreSQL | 9.6 | 分析平台数据库 |
分析平台系统Web端 | V3.0 | 分析平台系统Web端 |
分析平台系统服务端 | V3.0 | 分析平台系统服务端 |
Archlous调度系统 | V5.0 | 作业资源调度及监控 |
Zookeeper | V3.4 | 重要组件 |
Ldap | 2.4.39 | 用户统一管理 |
Srm-dns | 1.0 | 提供集群服务解析 |
docker | 1.13.1 | 提供基础服务运行支撑 |
表2 硬件环境
Table 2 Hardware environment
设备名称 | CPU | 内存 (GB) | 数量 (个) |
应用服务器 | Intel(R) Xeon(R) CPU E5-2630 v3 @ 2.40GHz 32核 | 64 | 3 |
计算服务器 | Intel(R) Xeon(R) CPU E5-2630 v3 @ 2.40GHz 64核 | 128 | 3 |
存储服务器 | Intel(R) Xeon(R) CPU E5-2630 v3 @ 2.40GHz 32核 | 128 | 3 |
1.3 平台可视化功能设计
转基因大豆分析平台系统功能模块主要包括:资源调度管理模块、操作动态显示模块、综合管理模块及流程分析编辑器等。分析平台采用容器作为调度和管理的基本单位,将不同分析工具或同一分析工具的不同版本封装到各自独立且完整的自包含容器中,彻底解耦合了计算设备的运行环境和各自分析工具的运行环境。
操作动态显示模块用于显示所有用户工作区的近期活动状态。综合应用管理功能模块包括首页功能模块、数据资源模块、用户登录、作业调度等内容。流程分析编辑器支持用户对WDL等分析流程的上传、镜像工具的绑定,分析流程及相关流程参数的查询,生信分析作业的提交、查询、删除、取消,分析作业结果目录的查询与下载等。该分析平台简化了不同角色的用户在应用环境中执行生物信息分析的操作流程,同时支持用户在Web端进行可视化操作。
分析平台系统架构设计如下。1)应用层:采用业界成熟的Vue.Js+SpringBoot框架[10]以微服务的形式进行产品模块构建,保证系统在松耦合的基础之上增强系统的可靠性和稳定性;2)数据层:通过ETL数据清洗以及独有的分词检索技术,保证前端检索结果准确度;数据库部分针对结构化和非结构化数据,分别采用关系型数据库(PostgreSQL)+分布式数据库(mongodb)的设计思路,通过redis缓存数据库技术,提升前端检索效率[11];3)权限控制层:采用Spring security[12]安全框架;4)数据安全层:均采用SSL加密传输技术,微服务模块之间也采用加密认证的方式进行通讯,数据库安全部分,在不造成性能损耗的基础上,采用表空间级别的加密技术,保证密钥独立、权控独立、算法独立(RSA\SM2);5)底层存储:采用HDFS[13]文件系统提高数据吞吐量,加速应用层的数据检索请求。
1.4 数据来源及处理
通过梳理现有公开的常见贸易转基因大豆品系相关资料,根据目前截获与合法贸易情况,确定本研究涉及的进出境转基因大豆品系。从全球开源数据库美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)[14]中收集野生型大豆的基因组数据及转基因大豆各个转基因品系外源基因序列,对转基因大豆序列编号、序列名称、转录本、序列长度、序列描述、数据来源、分类标签等数据信息进行统一的标准化、结构化,运用计算存储服务器进行整合和挖掘应用,最后将整合后的数据录入到数据库中。
根据转基因标签数据库数据内容、数据属性及数据之间的逻辑关系设计并构建转基因标签数据库总体架构,包括应用层、数据层、权限控制层、数据安全层、底层存储等;根据生物作业的并行特征抽象成了迭代、嵌套的多个模型;根据转基因标签合法性分析生物信息流程描述和作业定义输入,动态将预定义流程边执行边构造成一个有向图,从而根据该有向图调度转基因标签合法性分析作业,实现将转基因标签合法性分析作业。此外,转基因标签合法性分析平台引擎使用容器作为调度和管理的基本单位,将不同分析工具或同一分析工具的不同版本封装到各自独立而完整的自包含容器中,彻底解耦合了计算设备的运行环境和各自分析工具的运行环境,构建了主要包括泛基因组比对过滤、未知序列组装、基因预测、功能注释等转基因植物标签合法性生物信息学分析数据库。
1.5 数据库架构
根据转基因大豆基因组数据库数据内容、数据属性及数据之间的逻辑关系设计并建立数据库总体架构,包括应用层、数据层、权限控制层、数据安全层、底层存储等,基于数据存储和聚合查询等技术实现对数据的统一管理,实现在囊括海量数据的数据库中进行高效组织与索引,将转基因标签数据进行集中管理与共享,支持快速溯源某检索条件下对应的信息和数据路径,支持对转基因大豆数据进行分析与应用。
1.6 基因组检索系统
转基因大豆分析平台的高性能分布式多维检索系统主要基于Vue.js、Go、HDFS、HBase、JStrom、MapReduce[6,7,15-17]等技术构建,用户通过SQL API进行查询时,查询引擎将查询切分成子查询,并分配到不同的查询引擎,不同的查询引擎会将查询结果进行合并后再返回给用户。
基于站内已有数据资源,用户仅需输入相关基因名称、转录本名称、突变位点等关键词,即可根据搜索条件得到筛选结果,同时还可以在此基础上进行二次精确筛选。可对搜索结果进行自定义排序以展现不同角度的分析视野。对于部分赋予权限的用户,还可对搜索结果进行下载。对于结构化数据,当数据量达到百万级别时,搜索结果能够实现秒级响应;对于非结构化数据,当数据量达到PB级别时,单个文件的搜索结果可以实现秒级响应。
1.7 数据分析流程
数据分析流程主要利用Docker[4]容器技术,将转基因大豆分析流程分为若干模块进行封装,细化流程的颗粒度,提高开源分析模块算法更新的便捷度,使用WDL[8]等多种国际通用的工作流描述语言重构分析流程,将分析流程维护周期缩短。同时采用分布式调度方式,并行、弹性调用计算资源,大幅降低流程维护难度。基于基因组学可视化技术聚合智能缓存和对象循环等各种不同技术加速分析流程,使内存占用量降至最低,加载速度达到秒级,可以向用户显示基因组学数据相关信息,支持可视化工作流编辑与执行,快速处理海量基因数据。
2 结果与分析
2.1 转基因大豆可视化基因数据库及分析平台
可视化基因数据库及分析平台提供了序列库、基因库以及作业管理模块等,用户可通过该平台快速进行转基因大豆数据分析。平台所有功能均可通过Web界面进行可视化操作,无需配置Linux操作系统,也不必安装复杂的分析软件、更不用下载大量的数据库。工作人员只需在平台中选择相关分析流程,输入相应文件,设置计算参数,通过可视化Web界面提交作业,即可向计算集群投递生信分析计算任务,即可快速高效地完成分析任务,并实时获取数据分析结果与分析报告。
2.2 分析流程及结果注释
转基因大豆数据分析流程主要包括:泛基因组比对过滤、未知序列组装、基因预测、功能注释等。首先对大豆样品测序序列进行质控分析并过滤掉低质量碱基和序列;将高质量序列与大豆泛基因组进行比对,与泛基因组匹配的序列作为基质序列,未与泛基因组匹配的序列作为可疑序列;将上述筛选出的可疑序列进行组装,得到初步组装结果,组装结果根据统计阈值参数判断该样本是否为转基因大豆样本;对组装后的可疑转基因序列通过GeneScan[9]进行基因预测,得到基因编码区序列(CDS);将得到的基因编码序列(CDS)与非靶向合法转基因数据库进行比对,BLAST2.10[18]输出比对结果,鉴别合法转基因样本及候选转基因样本;将候选转基因样本序列与GeneBank[19]等数据库进行比对,查询序列来源和基因功能,根据序列来源、基因功能人工判断是否为非法转基因序列。
2.3 样品高通量测序数据测试分析实例应用
以大豆样品GTS_SYH_89788_87751A为例,对该样品高通量测序数据进行测试分析:
1)访问并登录转基因大豆分析平台Web端;
2)选择转基因大豆数据分析流程;
3)点击创建作业;
4)选择待分析样本:GTS_SYH_89788_87751A(包括样本名称:GTS_SYH_89788_87751A,测序样本fastq文件:GTS_SYH_89788_87751A_R1.fastq.gz,GTS_SYH_89788_87751A_R2.fastq.gz),设置流程参数,并选择其他输入文件,包括:野生型大豆参考基因组序列文件、转基因品系外源基因序列文件、转基因品系信息文件等;
5)点击提交作业;
6)作业运行结束后,点击下载分析结果及相应分析报告,GTS_SYH_89788_87751A样品分析报告显示:该大豆样本分析结果为GMO(转基因)、Illegal(不合法)序列,即该大豆样本序列为非法转基因大豆序列;
7)以抗除草剂草甘膦转基因(Co-CP4-EPSPS)标准品运行测序程序,获得7.5 G有效数据。数据镜像至服务器后,以大豆泛基因组数据库和转基因序列数据库进行分析,最终成功对待测样品中抗除草剂转基因序列(Co-CP4-EPSPS)进行了预测和注释(获得一条与Co-CP4-EPSPS相匹配序列,长度1173 bp,相似度99.9%)。
2.4 分析平台使用结论
该分析平台简化了转基因大豆数据分析操作,流程的可视化编排便利检测技术人员对数据的分析操作,“一键式”操作即可实现转基因大豆数据分析。
使用人员通过Web浏览器登录该分析平台,导入待鉴定大豆样本测序数据,可视化勾选转基因大豆分析流程,即可对数据进行“一键式”分析,并获取转基因大豆鉴定参考分析报告。
3 讨论
3.1 本研究已有成果
本研究依托动植物泛基因组构建非靶向转基因鉴别应用算法、开发生物信息分析流程及计算资源优化与重构、数据存储和聚合查询技术、基因组数据可视化技术、生物信息计算分析引擎等关键技术,聚焦口岸基因测序与智能化分析平台、生物数据计算存储平台、基因组学数据库系统等任务,按照数字化、模型化、软件化的技术思路,建立了达到国内领先水平的转基因大豆分析平台。
该分析平台可实现容器管理、流程管理、监控管理、资源池管理、用户管理、配置管理、镜像管理等功能,支持可视化流程编排与调试,可支持一体化、自动化转基因大豆数据分析、数据管理等服务,并可按需对平台提供计算、存储资源等支持,有效解决了转基因大豆数据分析鉴定的突破问题,有助于海关部门对转基因大豆序列来源和合法性鉴定、未知转基因品系发现能力的提升,实现口岸转基因物种高通量快速精准探测,全面覆盖生物安全风险防控需求。
随着我国外贸形势不断变化和转基因生物全球化,海关系统抵御转基因生物安全性风险的“国门卫士”作用日显重要[20]。因此,构建口岸基因测序与智能化分析平台,可一站式解决转基因植物等物种从测序、分析、鉴定到数据存储与管理等问题,实现精准、快速的转基因成分来源和合法性鉴定,以及未知转基因品系的及时发现,有利于保障国门生物安全。
3.2 尚未解决的问题
目前转基因大豆生信分析平台涵盖物种较为单一,尚只能专门针对转基因大豆进行数据分析,下一步需要扩展研究对象范围,开展对玉米、棉花、油菜等其他转基因物种数据分析研究,形成涵盖物种丰富的口岸基因测序与智能化分析平台。另外,转基因大豆生信分析平台尚未推广到系统内其他海关,因此平台数据来源相对不足,下一步需要加大推广,广泛收集口岸截获及合法贸易样本,将测序得到的数据信息添加至现有数据库,提升现有数据容量和多样性,并进一步扩大生信平台的服务对象,探索更广阔的应用场景。
3.2 应用前景
转基因大豆生信分析平台操作便捷,分析流程快速、智能,降低了生物信息分析师的学习成本,缩减了海关系统运营成本,将成为海关植物检疫工作者对转基因大豆序列进行分子溯源、合法性鉴定及发现未知转基因品系的有力工具,并且在平台的使用中可逐渐形成海关系统转基因物种生物信息分析的特色方法学,从源头上做好转基因物种等重大生物安全事项的监测以及防控,防止非法转基因产品进入我国消费市场,保障国门生物安全,为智慧海关建设减负增效。
3.3 优化与完善
转基因大豆生信分析平台后续有望在全国海关进行应用推广,广泛收集各地海关口岸截获及合法贸易样本,并将测序得到的数据信息收录至现有数据库,扩大数据规模,丰富数据维度,细化数据颗粒度。同时,以市场为导向进行定向开发和应用拓展,吸引更多优质资源投入,进而提升行业整体水平。同时,不断挖掘数据价值,探索更多的服务空间和业务创新模式,例如追踪国外转基因新品系研发、商业化进展,前瞻性地收集所有进入商业化种植的转基因植物品系基因组数据,完善海关系统转基因品系数据库。
参考文献
[1]常亮, 韩辉, 郭铮蕾, 等. 国门安全生物资源库建设的研究和启示[J]. 中国口岸科学技术, 2022, 4(5): 4.
[2]梁照文, 王美玲, 郑炜, 等. 完善国门生物安全防御体系建设工作的思考[J]. 植物检疫, 2017, 31(1): 4.
[3]王永刚, 于洋, 左天荣, 等. 国门生物安全风险防范中进出境动物检疫的经验和做法[J]. 口岸卫生控制, 2021, 26(2): 36-38+51.
[4] Merkel D. Docker: lightweight linux containers for consistent development and deployment[J]. Linux Journal, 2014, 239(2): 2.
[5] Group T. PostgreSQL: The World’s Most Advanced Open Source Database[DB/OL]. https://www.postgresql.org/., 2023-06-29.
[6]朱二华. 基于Vue.js的Web前端应用研究[J]. 科技与创新, 2017(20): 3.
[7] Fox C. The Go Programming Language[J]. Computing Reviews, 2016, 57(4): 225-226.
[8] Openwdl/wdl: Workflow Description Language[EB/OL]. https://github.com/openwdl/wdl. 2020.
[9] Erdman D D, Weinberg G A, Edwards K M, et al. GeneScan Reverse Transcription-PCR Assay for Detection of Six Common Respiratory Viruses in Young Children Hospitalized with Acute Respiratory Illness[J]. Journal of Clinical Microbiology, 2003, 41(9): 4298-4303.
[10]杨妍. 基于Spring Boot与Vue的系统管理模块开发探究[J]. 电声技术, 2019, 43(2): 32-34.
[11]朱亚兴, 余爱民, 王夷. 基于Redis+MySQL+MongoDB存储架构应用[J]. 微型机与应用, 2014, 33(13): 3-5+9.
[12] Mick Knutson, Robert Winch, Peter Mularien. Spring Security: Secure Your Web Applications, RESTful services, and microservice architectures[M]. Birmingham: Packt Publishing Ltd., 2017.
[13]翟永东. Hadoop分布式文件系统(HDFS)可靠性的研究与优化[D]. 武汉: 华中科技大学, 2011.
[14] Sayers E W, Beck J, Brister J R, et al. Database Resources of the National Center for Biotechnology Information[J]. Nucleic Acids Research, 2007, 35, Database issue: D5-D12.
[15]郝树魁. Hadoop HDFS和MapReduce架构浅析[J]. 邮电设计技术, 2012(7): 37-42.
[16] Taylor R C. An Overview of the Hadoop/MapReduce/HBase Framework and Its Current Applications in Bioinformatics[J]. Bmc Bioinformatics, 2010, 11(Suppl 12): 1-6.
[17] Wang D, Huang C, Ju Z. Performance Optimization of Distributed Real-Time Computing System JStorm[C]. 2017 4th International Conference on Information Science and Control Engineering (ICISCE). IEEE Computer Society, 2017.
[18] Jian Y, Scott M G, Madden T L. BLAST: Improvements for Better Sequence Analysis[J]. Nucleic Acids Research, 2006, 34(Web Server issue): W6-9.
[19] Sayers E W, Cavanaugh M, Clark K, et al. GenBank 2023 update[J]. Nucleic acids research, 2023, 51(D1): D141-D144.
[20]张明辉, 鞠永涛, 尹晓燕. 加快国门生物安全体系建设[J]. 中国海关, 2020(9): 82-83.
基金项目:海关总署科研项目(2022HK141)
第一作者:陆冠亚(1981—),男,汉族,江苏常熟人,硕士,高级兽医师,主要从事进出口动物检疫、动物源性成分及动物物种鉴定工作,E-mail: 2643551374@qq.com
通信作者:赵晓燕(1972—),女,汉族,山西运城人,硕士,高级兽医师,主要从事进出口动物检疫、动物源性成分及动物物种鉴定工作,E-mail: 107168686@qq.com
1. 南京海关动植物与食品检测中心 南京 210019
2. 中国海关科学技术研究中心 北京 100026
1. Animal, Plant and Food Inspection Center of Nanjing Customs District, Nanjing 210019
2. Science and Technology Research Center of China Customs, Beijing 100026








