CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
大连和丹东地区甲型肝炎病毒流行株基因型分析
作者:李长征 田卓 顾晗潇 麻丽丹
李长征 田卓 顾晗潇 麻丽丹
Abstract This study collected a total of 129 serum samples from acute hepatitis A patients in Dalian and Dandong from 2016 to 2020. After the RNA of hepatitis A virus (HAV) being extracted, the VP1 and VP3 region sequences were used as specific gene targets for Nest PCR amplification reaction. The positive PCR products were sequenced and compared with 26 reference sequences downloaded from Genbank to construct a phylogenetic tree. The homology of nucleotide and amino acid sequences in samples with different coding regions was analyzed for determining the genotype. The experimental results showed that 86.7% (104/120) of HAV-positive patients were over 30 years old, and the proportion of male patients (63.3%) were higher than that of female patients (36.7%). 72 VP1 region sequences and 84 VP3 region sequences were successfully obtained from serum samples, all of which had the highest nucleotide identity with the Japanese HAV sequence (VP1 region: 97.45%-99.79%; VP3 region: 97.27%-99.76%). The phylogenetic tree showed that the prevalent strains of hepatitis A virus all belonged to gene subtype IA. The obtained sequences have amino acid identity of 99.36% to 100% and 99.29% to 100% in the VP1 and VP3 regions, respectively, with no amino acid changes. The research results indicated that the HAV prevalent strains in Dalian and Dandong from 2016 to 2020 were genotype IA, which belonged to the same genotype as the prevalent strains of HAV in Japan. This study provides molecular epidemiological evidence for the prevention and control of the spread of hepatitis A in Dalian and Dandong, and lays the foundation for the tracing research of HAV in the region.
Keywords hepatitis A virus; epidemic strain; genotype
甲型肝炎病毒(Hepatitis A virus,HAV)是一种无衣壳的RNA病毒,可在海水、淡水、废水和土壤中存活[1]。HAV主要通过饮用或食用被污染的水源和贝类等,或与被感染的患者接触等方式进行传播,引起人类急性病毒性肝炎,其中污染水源或食物的摄入是造成疫情暴发流行的主要原因[2-3]。全球每年因感染HAV的病例人数超过150万,是全球范围内被广泛关注的公共卫生问题之一,给人类健康造成了巨大威胁[4-5]。HAV只有一种血清型和一个抗原系统,抗原结构相对稳定,抗原中和位点主要位于P1编码结构蛋白的VP1、VP2和VP3区,其中VP1和VP3能够在病毒表面形成单一且占据主导优势的抗原决定表位,以引起中和抗体的应答[6-9]。大连和丹东作为辽宁省主要沿海城市,甲型肝炎发病率一直处于较高水平[10-11]。本研究对大连和丹东地区2016—2020年收集的HAV流行株进行基因分型研究,为辽宁省HAV分子流行病学调查、溯源和防控提供有力的技术支持。
1 材料与方法
1.1 材料与仪器
辽宁省丹东市、大连市急性甲肝患者血清标本共计129份(标本采集时间为2016—2020年。样品由丹东市疾病预防控制中心、大连市疾病预防控制中心提供),所有血清均经化学发光微粒子免疫检测法检测,结果均为抗HAV IgM阳性,于-80℃保存。
病毒RNA提取试剂盒(QIAamp Viral RNA Mini Kit,QIAGEN),逆转录酶(Avian Myeloblas-tosis Virus Reverse Transcriptase,Promeg),脱氧核糖核酸聚合酶(Ex-Taq,Takara),纯化试剂盒(MinElute Gel Extraction kit,QIAGEN)。
1.2 实验方法
1.2.1 标本中HAV核酸提取
使用QIAGEN QIAamp Viral RNA Mini Kit病毒核酸抽取试剂盒提取血清中的病毒核酸,按照试剂盒说明书进行提取操作,最后洗脱于50 μL焦碳酸二乙酯(Diethyl Pyrocarbonate, DEPC)处理水中,立即进行逆转录或冻存于-80℃。
1.2.2 引物信息
HAV VP1和VP3区引物来源于参考文献[11-13],引物序列位置均参照HAV毒株HM175(GenBank号:M14707)。具体序列信息见表1,实验所需引物均由生工生物工程(上海)股份有限公司合成。
1.2.3 逆转录-聚合酶链反应
取HAV核酸阳性标本的病毒核酸10 μL,外侧下游引物(VP1/3 R1)1 μL,按照Avian Myeloblas-tosis Virus Reverse Transcriptase试剂说明书进行逆转录反应,反转录体积共计30 μL,反应参数为25℃ 15 min,42℃ 80 min,70℃ 20 min。
按照日本Takara公司的RR001A聚合酶试剂盒说明书配制并进行巢氏PCR反应,反应体积均为50 μL。
1)反应体系。第一轮PCR反应:浓度为10 mol/L的上下游引物(VP1/3 F1、VP1/3 R1)各1 μL,模板cDNA5 μL。第二轮PCR反应:浓度为10 mol/L的上下游引物(VP1/3 F2、VP1/3 R2)各1 μL,第一轮扩增产物2 μL。
2)反应条件。第一轮扩增反应:94℃ 5 min;94℃ 30 s,53℃ 30 s,72℃ 30 s,35个循环;72℃,7 min;4℃保存。第二轮扩增反应:94℃ 5 min;94℃ 30 s,56℃ 30 s,72℃ 30 s,35个循环;72℃,7 min;4℃保存。
扩增产物经1.0%琼脂糖凝胶电泳鉴定后,送至生工生物工程(上海)股份有限公司使用第二轮内侧上下游引物进行双向测序。
1.2.4 基因序列分析
使用Bioedit综合序列分析软件对测序结果进行拼接和校正。使用MEGA 7.0软件的双参数遗传距离(Kimura 2-parameter)核苷酸进化模型,Bootstrap验证1000次;基于邻接法(Neighbor Joining,NJ)分别构建HAV VP1、VP3区系统进化树,将所测序列与参考株序列进行核苷酸、氨基酸的一致性比对分析。
2 结果与分析
2.1 标本基本资料及特征
129例急性甲肝患者血清标本中,有9例标本的患者年龄及性别信息不详,其余120例标本中,患者年龄及性别分布见表2,30岁以上患者数量明显高于30岁以下患者,男性患者数量高于女性患者。
表2 标本分布信息
Table 2 Sample distribution information
项目 | 分类 | 数量 (例) | 比例 (%) |
年龄 | 30岁以上 | 104 | 86.7 (104/120) |
30岁以下 | 16 | 13.3 (16/120) | |
性别 | 男 | 76 | 63.3 (76/120) |
女 | 44 | 36.7 (44/120) | |
地区 | 大连 | 5 | 3.9 (5/129) |
丹东 | 124 | 96.1 (124/129) |
本研究对提取的129份标本甲肝病毒RNA进行核酸检测,其中20份标本HAV核酸阴性,109份标本核酸阳性,阳性率为84.5%。核酸阳性的109份标本分别进行VP1和VP3片段巢氏PCR扩增和扩增产物的测序,最终VP1测序成功标本数量为72份、VP3测序成功标本数量为84份。
2.2 VP1区核苷酸序列分析
本研究通过GenBank基因库下载HAV参考株序列共计26条,序列来源于中国、日本、美国、意大利、墨西哥等13个国家,基因亚型包括ⅠA和ⅠB型。通过美国国家生物技术信息中心(National center for biotechnology information,NCBI)官网(https://www.ncbi.nlm.nih.gov/),使用BLAST功能对获得序列进行核苷酸序列比对。
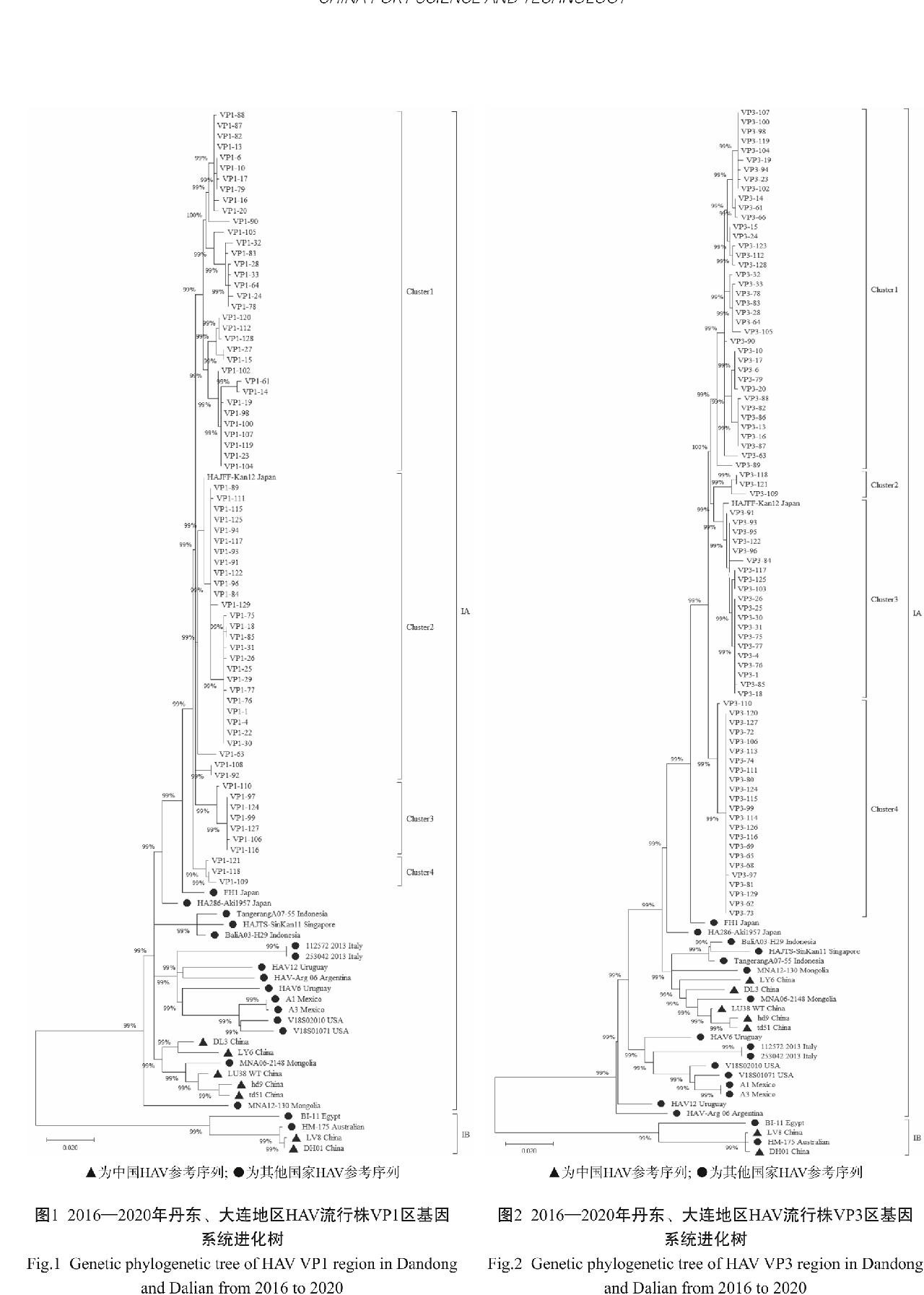
使用MEGA 7软件将72份HAV流行株与Genbank下载的HAV参考株序列根据VP1区的核苷酸序列构建系统发育树,结果如图1所示。所有HAV流行株样本核苷酸序列间一致性为96.92%~100%,与ⅠA亚型参考株LU38(GenBank no.AF357222.1/2003 China)的核苷酸一致性为94.37%~96.17%,与ⅠB亚型参考株HM175(GenBank no. M14707.1/1993 Australia)的核苷酸一致性为89.60%~90.73%。根据同一基因型间核苷酸差异小于15%[14],同一基因亚型核苷酸序列差异小于7.5%[15]的原则,表明72份HAV流行株属于ⅠA亚型。
72条HAV流行株序列位于进化树的不同分支,包括4个基因簇,分别为Cluster1、Cluster2、Cluster3和Cluster4(图1)。各基因簇中不同序列之间核苷酸一致性分别为96.92%~100%、99.04%~100%、99.36%~100%和99.47%~99.79%。
研究得到的序列与流行株HAJFF-Kan12(GenBank no. AB819870.1/2013 Japan)核酸序列最为接近,核苷酸序列一致性为97.45%~99.79%,其次为流行株FH1(GenBank no. AB020567.1/2001 Japan),核苷酸序列一致性为96.81%~98.51%。我国HAV序列中,最接近的为流行株DL3(GenBank no. AF512536.1/2004 China),核苷酸序列一致性为95.43%~96.59%。
2.3 VP3区核苷酸序列分析
根据VP1区的核苷酸序列,将本研究得到的84份HAV流行株与Genbank下载的HAV参考株序列构建系统发育树,结果如图2。所有HAV流行株样本核苷酸序列间一致性为96.44%~100%,与ⅠA亚型参考株LU38(GenBank no.AF357222.1/2003 China)的核苷酸一致性为94.55%~96.09%,与ⅠB亚型参考株HM175(GenBank no. M14707.1/1993 Australia)的核苷酸一致性为89.57%~91.32%,表明84份HAV流行株属于ⅠA亚型。
84条HAV流行株序列位于进化树的不同分支,包括4个基因簇,分别为Cluster1、Cluster2、Cluster3和Cluster4(图2)。各基因簇中不同序列之间核苷酸一致性分别为97.98%~100%、99.05%~100%、98.70%~100%和99.05%~100%。
研究得到的序列与流行株HAJFF-Kan12(GenBank no. AB819870.1/2013 Japan)核酸序列最为接近,核苷酸序列一致性为97.27%~99.76%,其次为流行株FH1(GenBank no. AB020567.1/2001 Japan),核苷酸序列一致性为96.45%~97.86%。我国HAV序列中,最接近的为流行株DL3(GenBank no.AF512536.1/2004 China),核苷酸序列一致性为94.31%~95.48%。
2.4 HAV流行株氨基酸序列一致性及主要中和抗原位点氨基酸序列分析
经MEGA 7软件分析,本研究中72份流行株序列与结构蛋白VP1区的氨基酸一致性为99.36%~100%;84份流行株序列与结构蛋白VP3区的氨基酸一致性为99.29%~100%。将所得序列分别与结构蛋白VP1区Ser102、Ser114、Val166、Val171、Lys105、Lys221、Asn104、Ala176、Trp170、Gln232,结构蛋白VP3区Ser71、Asp70、Pro65、Gln74等[16]主要参考序列氨基酸序列进行比对,发现研究所得序列均未发生氨基酸变化,表明本次研究样本在VP1、VP3区氨基酸序列均相对保守。
3 讨论
从连续4年收集的血清标本可以看出,近年来大连和丹东地区甲型肝炎患者的年龄趋于大龄化,86.7%的患者为年龄超过30岁的成人,与以往报道的全国甲型肝炎发病年龄主要集中在儿童和青壮年人群略有不同[16]。可能因为丹东和大连地区毗邻黄海海域,居民有生食或半生食海产品的习惯,并以青壮年食用此类海产品居多,大大增加了青壮年感染HAV的概率,并造成HAV的暴发和流行。
本研究以HAV中VP1和VP3区核苷酸序列为基础,对2016—2020年收集的急性甲肝患者血清进行基因分型分析,获得的72份流行株VP1区核苷酸序列间一致性为96.92%~100%,84份流行株VP3区核苷酸序列间一致性为96.44%~100%,得知此次收集的HAV流行株基因型为ⅠA亚型,为中国HAV流行株的主要亚型。不同编码区的进化树分支均分为多个基因簇,提示大连和丹东地区可能存在多个HAV传播链。研究表明HAV流行株进行基因分型时,采用结构蛋白VP3区分析效果好于采用结构蛋白VP1区,VP1区和VP3区的氨基酸一致性均较高,与主要中和抗原位点氨基酸序列比对也均未发生氨基酸变化,说明此次检测的HAV流行株VP1区和VP3区氨基酸序列非常保守。该研究对了解辽宁地区甲肝疫苗的应用效果具有重要意义。
通过进化树分析和GenBank比对后发现,大连和丹东地区的HAV流行株与分离自日本病人的HAV分离株(HAJFF-Kan12/GenBank no.AB819870.1)一致性最高,与我国DL3株(GenBank no. AF512536.1)的一致性相对较低。
为加强辽宁地区甲型肝炎疫情的防控工作,应加大地区宣传和监管力度,加强沿海地区居民的防范意识,并鼓励居民自愿接种甲肝疫苗,同时应进一步加强对毗邻国家进口海产品中HAV的检测和监管工作,同时对入境人员和货物加大检验监管力度,以保障人民群众生命健康安全。
参考文献
[1] Wang D, Cao J, Tian Z, et al. Development of a new concentration method for Hepatitis A virus detection (ISO 15216–2: 2019) in Manila clams (Ruditapes philippinarum)[J]. LWT-Food Science and Technology, 2022, 172: 114172.
[2] Vaughan G, Goncalves Rossi LM, Forbi JC, et al. Hepatitis A virus: host interactions, molecular epidemiology and evolution[J]. Infection Genetics And Evolution. 2014, 1(21): 227-243.
[3] 杨月, 孙博语, 周令, 等. 甲肝疫苗纳入扩大免疫规划前后大连市甲肝流行特征分析[J]. 国际病毒学杂志, 2019, 26(1): 63-65.
[4] Andani A, van Elten T M, Bunge E M, et al. Hepatitis A epidemiology in Latin American countries: A 2020 view from a systematic literature review[J]. Expert Review of Vaccines, 2020, 19(9): 795-805.
[5] 黄腾达. 基于二代测序和全基因组序列分析我国甲肝病毒流行株基因特征[D]. 北京: 中国疾病预防控制中心, 2018.
[6] Ma Q Q, Wang H J, Li J, et al. The infectivity and pathogenicity of hepatitis A virus live-attenuated vaccine strain H2 in type I interferon receptor-deficient mice[J]. Virologica Sinica, 2022, 37(5): 740-745.
[7] Shirasaki T, Feng H, Duyvesteyn H M E, et al. Nonlytic cellular release of hepatitis A virus requires dual capsid recruitment of the ESCRT-associated Bro1 domain proteins HD-PTP and ALIX[J]. PLoS Pathogens, 2022, 18(8): e1010543.
[8] Cao J, Bi S, Meng Q, et al. Genotyping of acute hepatitis a virus isolates from China, 2003–2008[J]. Journal of medical virology, 2011, 83(7): 1134-1141.
[9] 王昊. 我国甲肝病毒流行株全基因组序列分析及准种变异研究[D]. 北京: 中国疾病预防控制中心, 2014.
[10] 褚发军, 徐也晴, 闵霞, 等. 东港市2014年甲型肝炎病毒流行株基因分型研究[J]. 中国疫苗和免疫, 2014, 20(6): 536-537+561.
[11] 杨月, 朱琳, 杨芳, 等. 大连市2020年甲型肝炎病毒流行株基因特征[J]. 中国疫苗和免疫, 2023, 29(1): 47-52.
[12] Nainan O V, Xia G, Vaughan G, et al. Diagnosis of hepatitis A virus infection: a molecular approach[J]. Clinical microbiology reviews, 2006, 19(1): 63-79.
[13] 何晓兰, 黄腾达, 艾德尔艾力·阿有甫, 等. 新疆和田地区2016年甲型肝炎病毒流行株基因特征[J]. 中国疫苗和免疫, 2020, 26(2): 142-145.
[14] 郑惠惠. 中国部分甲肝病毒流行株结构蛋白VP3-VP1区基因特点分析及TaqMan Realtime PCR法定量检测甲型肝炎病毒核酸[D]. 北京: 中国疾病预防控制中心, 2011.
[15] 杜凤雪, 周文亭, 邱丰, 等. 16条甲型肝炎病毒流行株基因型及全基因组特征分析[J]. 病毒学报, 2022, 38(6): 1397-1404.
[16] 郝增平, 巴文生, 阿克忠,等. 青海省2004—2019年甲型肝炎流行病学特征[J]. 中国疫苗和免疫, 2021, 27(4): 446-449.
表1 引物核苷酸序列
Table 1 Nucleotide sequences of the primers
引物 | 序列 (5' ~3') | 位置 | 扩增产物片段大小 (bp) | |
名称 | 类型 | |||
VP1 F1 | 外侧 | GTTTTGCTCCTCTTTATCATGCTATG | 2167~2192 | 1106 |
VP1 R1 | CATCCATCTCAAGAGTCCACAC | 3384~3363 | ||
VP1 F2 | 内侧 | TATCATGCTATGGATGTTAC | 2181~2200 | |
VP1 R2 | TTCATTATTTCATGCTCCTCAG | 3286~3267 | ||
VP3 F1 | 外侧 | GCTAGGTTTACAGATTTGGAG | 1419~1439 | 948 |
VP3 R1 | TGTCTCAGGCACTTTCTTTGC | 2408~2388 | ||
VP3 F2 | 内侧 | ACTCCTCTTTCTACACAGATG | 1452~1472 | |
VP3 R2 | CACTTTCTTTGCTAAAACTGG | 2399~2379 | ||


▲为中国HAV参考序列; ●为其他国家HAV参考序列
图1 2016—2020年丹东、大连地区HAV流行株VP1区基因系统进化树
Fig.1 Genetic phylogenetic tree of HAV VP1 region in Dandong and Dalian from 2016 to 2020
▲为中国HAV参考序列; ●为其他国家HAV参考序列
图2 2016—2020年丹东、大连地区HAV流行株VP3区基因
系统进化树
Fig.2 Genetic phylogenetic tree of HAV VP3 region in Dandong and Dalian from 2016 to 2020
第一作者:鲍泽英(1974—),女性,汉族,浙江景宁人,本科,主治医师,主要从事口岸传染病流行病学研究,E-mail: 993719813@qq.com
通信作者:陈淑丹(1982—),女,汉族,浙江富阳人,博士研究生,副主任技师,主要从事微生物和传染病检测,E-mail: shudanjudy@163.com
1. 杭州国际旅行卫生保健中心(杭州海关口岸门诊部) 杭州 310012
2. 杭州机场海关 杭州 310003
1. Hangzhou International Travel Health Care Center (Hangzhou Customs Ports Outpatient Departments), Hangzhou 310012
2. Hangzhou Airport Customs, Hangzhou 310003