CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
沙门氏菌微滴式数字PCR定量检测方法的建立
作者:赵丽青 房保海 殷培军 刘云国 魏海燕 徐蕾蕊 马丹 焦洁
赵丽青 房保海 殷培军 刘云国 魏海燕 徐蕾蕊 马丹 焦洁
摘 要 本研究通过对肠炎沙门氏菌PCR特异性扩增引物序列及探针序列进行优化,建立了沙门氏菌微滴式数字PCR快速定量检测方法。实验结果显示,该方法可实现检测沙门氏菌菌悬液浓度范围为5.3×101~5.3×105 CFU/mL,可检测沙门氏菌有效基因组DNA浓度区间在2~20940拷贝/20 μL。采用该方法测得结果与菌落总数测试片相比,两者无显著性差异(p>0.05);与荧光定量聚合酶链式反应(Quantitative polymerase chain reaction,qPCR)方法相比,该方法可检测菌悬液浓度更低,且能实现准确快速定量。实验结果证明,该方法特异性强、灵敏度高、检测范围广、定量结果准确可靠,可满足公共卫生事件快速应急处理的需要,具有广泛的应用前景。
关键词 微滴式数字PCR(ddPCR);沙门氏菌;定量检测;拷贝数
Establishment of Droplet Digital PCR Assay for Quantitative Detection of Salmonella
ZHAO Li-Qing1 FANG Bao-Hai1 YIN Pei-Jun1* LIU Yun-Guo2
WEI Hai-Yan3 XU Lei-Rui3 MA Dan3 JIAO Jie4
Abstract In this study, a rapid quantitative detection method for Salmonella based on droplet digital PCR (ddPCR) was successfully established by designing synthetic Salmonella enteritidis specific PCR amplification primers and probe sequences. This method could detect Salmonella bacterial suspension concentrations ranging from 5.3×101 to 5.3×105 CFU/mL, and the effective genomic DNA concentration of Salmonella could be quantified in the range of 2 to 20940 copies/20 μL. The results obtained by this method were not significantly different from the total colony count test strip (p>0.05). Compared with the quantitative polymerase chain reaction (qPCR) method, this method could detect lower concentrations of bacterial suspension and can achieve accurate and rapid quantification. The experimental results demonstrate that this method has strong specificity, high sensitivity, broad detection range, and reliable quantitative results. These attributes make it well-suited for rapid emergency response to public health events and have the potential for further development.
Keywords droplet digital PCR (ddPCR); Salmonella; quantitative detection; copy number
基金项目:国家重点研发计划(2021YFF0602803)
第一作者:赵丽青(1980—),女,汉族,山西和顺人,硕士,高级兽医师,主要从事微生物检测工作,E-mail: zlq3022@163.com
通信作者:殷培军(1979—),男,汉族,山东青岛人,硕士,高级农艺师,主要从事植物检疫及微生物检测工作,E-mail: yinqaz309@126.com
1. 青岛海关技术中心 青岛 266109
2. 临沂大学生命科学学院 临沂 276000
3. 中国海关科学技术研究中心 北京 100011
4. 北京理工大学 北京 100081
1. Technology Center of Qingdao Customs District, Qingdao 266109
2. College of Life Science, Linyi University, Linyi 276000
3. Science and Technology Research Center of China Customs, Beijing 100011
4. Beijing Institute of Technology, Beijing 100081
沙门氏菌(Salmonella)属于肠杆菌科(Entero- bacteriaceae),广泛分布于自然界,是一类可引起人畜共患的危害严重的食源性致病菌[1]。其中,伤寒沙门氏菌只感染人类,可对肝、脾和骨髓造成损害;鼠伤寒沙门氏菌可感染一切地上走的或爬的活物,在人类身上造成的症状一般是食物中毒,通常会出现发热、腹泻、呕吐、腹痛等症状[2-3]。研究发现,部分肉制品感染沙门氏菌的概率较高[4] ,因此加强对沙门氏菌的检测尤为重要[5-6] 。
目前,检测沙门氏菌的方法有很多,例如酶标抗体法(Eenzyme Linked Immunosorbent Assay,ELISA)、同位素标记抗体法、荧光染色法[7]荧光定量聚合酶链式反应(Quantitative Polymerase Chain Reaction,qPCR)[8]等。与传统微生物培养法相比,虽然各有一定的优势[9-10]且方法本身也被证明是可靠的,但是仍存在灵敏度低、操作繁琐、有时不能反映样品的真实污染水平[11-12]等缺点。因此,针对沙门氏菌单独研究开发出一种既能实现快速准确定量,又能操作便捷的检测方法就显得非常必要。
微滴式数字PCR技术(droplet digital PCR,dPCR)可精确、快速实现DNA绝对定量[13-15],从核酸提取至完成检测最快能在2~3 h内完成[16-17]。它既能解决传统培养法周期长的问题,又能够对检测目标进行准确定量。本研究通过对鸡肉样品进行阳性添加[18-19],来确定建立的沙门氏菌ddPCR定量检测方法的可行性。
1 材料和方法
1.1 材料与仪器
试验所用菌株见表1。
试验主要仪器及材料:CF16RXI Ⅰ高速冷冻离心机(日本HITACHI公司);罗氏480实时荧光定量PCR系统(德国Roche公司);QX200微滴式数字PCR系统(美国Bio-Rad公司);U3010紫外可见分光光度计(日本HITACHI公司);Excella E25R恒温摇床(美国NBS公司);PetrifilmTM 菌落总数测试片(德国3M公司)。
表1 试验菌株
Table 1 Test strains
菌株编号 | 菌种名称 | 革兰氏属性 |
ATCC13076 | 肠炎沙门氏菌 | G- |
ATCC13311 | 鼠伤寒沙门氏菌 | G- |
ATCC14028 | 鼠伤寒沙门氏菌 | G- |
ATCC 25933 | 奇异变形杆菌 | G- |
ATCC43864 | 弗氏柠檬酸杆菌 | G- |
ATCC43162 | 布氏柠檬酸杆菌 | G- |
ATCC27156 | 柯氏枸橼酸杆菌 | G- |
ATCC 51755 | 大肠杆菌 | G- |
ATCC49132 | 普通变形杆菌 | G- |
NICPBP50205 | 阪崎肠杆菌 | G- |
ATCC700323 | 阴沟肠杆菌 | G- |
NICPBP51592 | 宋氏志贺氏菌 | G- |
ATCC15313 | 单核细胞增生李斯特氏菌 | G+ |
ATCC35967 | 格氏李斯特菌 | G+ |
ATCC19119 | 伊氏李斯特 | G+ |
ATCC 29213 | 沙门氏菌 | G+ |
CGMCC1.2429 | 表皮葡萄球菌 | G+ |
1.2 实验方法
1.2.1 引物、探针的设计
参考标准SN/T 1870—2016《出口食品中食源性致病菌检测方法 实时荧光PCR法》[20]合成引物、探针,见表2。
表2 沙门氏菌特异性PCR扩增引物和探针序列
Table 2 Primers and probe sequences for Salmonella-specific PCR amplification
引物/探针 | 序列 (5'-3' ) |
正向引物 | GCGGCGTTGGAGAGTGATA |
反向引物 | AGCAATGGAAAAAGCAGGATG |
探针 | CATTTCTTAAACGGCGGTGTCTTTCCCT |
1.2.2 菌株活化及DNA提取
取-80℃保存的菌株,在含0.6%酵母浸膏的胰酪胨大豆琼脂(TSA-YE)培养基上37℃培养16~24 h,然后挑取纯菌落至10 mL的营养肉汤中振摇培养24 h。将最终培养好的菌液用生理盐水稀释,依次梯度稀释至原浓度的10-1、10-2、10-3、10-4、10-5、10-6、10-7倍,做好标记备用 。采用3M测试片分别对不同浓度的菌悬液进行菌落总数计数,每个浓度设定3个平行。同时,分别取各梯度菌悬液3 mL,采用北京天根生化科技有限公司细菌基因组DNA提取试剂盒(货号:DP302)提取细菌DNA[21] 。
1.2.3 数字PCR试验
ddPCR反应体系:12.5 μL扩增预混液(ddPCRTM SuperMix for Probes),上、下游引物各1.0 μL(10 pmol/μL),0.5 μL探针(5 pmol/μL),2.5 μL DNA模板,用双蒸水补足体系总体积至25 μL。
将20 μL分装预混液加入到微滴生成卡的样品孔,70 μL的微滴生成油(Droplet generator oil)加入oil孔位,进行微滴生成。然后,将40 μL微滴缓慢地打入96孔板相应位置孔内,封膜后在30 min内进行PCR扩增反应。
反应条件:95℃预变性10 min;95℃变性30 s,60℃退火1 min,40个循环;98℃固化微滴10 min。
1.2.4 qPCR试验
参考标准SN/T 1870—2016中方法进行荧光定量PCR。
1.2.5 数字PCR方法特异性试验
按照优化方法,利用QX200微滴式数字PCR系统,对表1中17株菌株提取核酸,按照1.2.4试验要求,对上述核酸进行检测,以确定该方法的特异性。
1.2.6 数字PCR方法的灵敏度
将沙门氏菌DNA进行梯度稀释,按照1.2.4和1.2.5所述方法分别开展数字PCR试验和实时荧光PCR试验,每个梯度均重复3次,进行灵敏度研究。
1.2.7 Copy数和菌液浓度的线性关系
建立ddPCR的Copy数和菌液浓度的线性关系。数字PCR的Copy数与对应菌悬液浓度的换算关系公式见公式(1)[20]:
 (1)
(1)
式(1)中,C表示菌悬液浓度(CFU/mL)(根据ddPCR的Copy数换算);X表示ddPCR20 μL反应体系的Copy数(个/20 μL);V1表示DNA最终的定容体积,为150 μL,V2表示提取DNA的菌悬液体积,为3 mL,V3表示反应体系中模板体积,为2 μL。
1.2.8 人工污染样品检测
无菌操作称取25.0 g鸡肉样品,置于盛有225 mL缓冲蛋白胨水(BPW)的无菌均质袋中,用拍击式均质器制成鸡肉匀浆。采用人工染菌方式制备样品,样品污染浓度在10~107 CFU/mL,并设置阴性、阳性对照。按照1.2.3所述方法提取沙门氏菌DNA,进行ddPCR检测。
1.3 数据处理
每个试验重复3次,应用SPSS对数据作显著性及相关性分析,显著性水平为0.05。
2 结果与分析
2.1 数字PCR方法特异性验证结果
按照1.2.2所述方法提取表1中所列17种菌株的DNA,用1.2.3所述方法进行ddPCR扩增。试验结果如图1所示,3株沙门氏菌(ATCC13076、ATCC13311和ATCC14028)出现阳性微滴信号,而其他菌株及阴性对照均未出现扩增,表明1.2.1中的沙门氏菌引物探针特异性较强。
1: ATCC13076肠炎沙门氏菌; 2: TCC13311鼠伤寒沙门氏菌; 3: ATCC14028鼠伤寒沙门氏菌; 4: 阴性对照; 5: ATCC 25933奇异变形杆菌; 6: ATCC43864弗氏柠檬酸杆菌; 7: ATCC43162布氏柠檬酸杆菌; 8: ATCC27156柯氏枸橼酸杆菌; 9: ATCC 51755大肠杆菌; 10: ATCC49132普通变形杆菌; 11: NICPBP 50205阪崎肠杆菌; 12: ATCC700323阴沟肠杆菌; 13: NICPBP51592宋氏志贺氏菌; 14: ATCC 15313单核细胞增生李斯特氏菌; 15: ATCC35967格氏李斯特菌; 16: ATCC19119伊氏李斯特; 17: ATCC 29213沙门氏菌; 18: CGMCC1.2429表皮葡萄球菌
图1 数字PCR方法特异性验证结果
Fig.1 Specific verification results of ddPCR
2.2 数字PCR方法的灵敏度结果
沙门氏菌菌悬液初始浓度为5.3×107 CFU/mL,梯度稀释后,对不同浓度菌悬液提取DNA,然后进行ddPCR扩增,B05、C05、D05、E05、F05、G05、H05通道对应菌悬浓度(CFU/mL)分别为5.3×106 CFU/mL、5.3×105 CFU/mL、5.3×104 CFU/mL、5.3×103 CFU/mL、5.3×102 CFU/mL、5.3×101 CFU/mL、5.3×100 CFU/mL,如图2所示。本试验ddPCR能够检测到的沙门氏菌最低的菌悬液浓度为5.3×101 CFU/mL,检测得到的拷贝数为2.0 copies/20 μL,推测得模板的拷贝数为5.0×101 copies/mL,与菌悬液浓度不具有显著性差异(p>0.05)[15]。

图2 ddPCR灵敏度结果图
Fig.2 Sensitivity diagram of ddPCR
对同样浓度的沙门氏菌DNA模板进行荧光PCR,结果如图3所示。当浓度≥5.3×103 CFU/mL时,对应的Ct值≤35,检测结果为阳性;当浓度为5.3×102 CFU/mL时,35<Ct值≤40,结果需再次PCR确证;当浓度≤5.3×101 CFU/mL时,对应的Ct值≥40,检测结果为阴性。如表3所示,当菌液浓度为5.3×101 CFU/mL,qPCR结果为阴性,而ddPCR最低检出菌液浓度为50 CFU/mL,检测沙门氏菌灵敏度高、结果准确。
图3 荧光定量PCR灵敏度结果图
Fig.3 Sensitivity map of qPCR
以沙门氏菌菌悬液浓度为横坐标,以此浓度提取的DNA为模板进行ddPCR扩增,将所得Copy数为纵坐标,如图4所示,生成标准曲线线性关系良好(R2≥0.99),说明应用ddPCR技术对沙门氏菌开展定量检测是可行的,检测结果是可靠的,为进一步的实际应用提供数据支持。
2.3 重复性分析
通过3次重现实验得到ddPCR方法检测沙门氏菌的重现性,样品经过PCR扩增和微滴生成分析,生成的阳性微滴的拷贝数结果如图5所示。结果显示,标准偏差为0.71%,表明该方法重复性好。

图5 ddPCR检测拷贝数(n = 3)
Fig.5 Copy number detected by ddPCR (n = 3)
2.4 人工污染样品检测
对不同菌量人工污染的鸡肉样品,采用qPCR方法和ddPCR方法开展沙门氏菌检测,其中A09、A08、C08、D08、E08、F08、H08、G08通道对应菌悬浓度(CFU/mL)分别为5.3×107 CFU/mL、5.3×106 CFU/mL、5.3×105 CFU/mL、5.3×104 CFU/mL、5.3×103 CFU/mL、5.3×102 CFU/mL、5.3×101 CFU/mL、5.3×100 CFU/mL、D10阳性对照、C10阴性对照。从表4和图6可以看出,qPCR只能浓度为5.3×103 CFU/mL的沙门氏菌,ddPCR检测最低可检测菌悬液浓度为5.3×101 CFU/mL的沙门氏菌,并且ddPCR的检测相对标准偏差(relative standard deviation,RSD)均小于5.0%。
鸡肉中添加沙门氏菌,采用本研究建立的数字PCR进行检测,每个样品的检测结果均与1.2.3直接提取菌悬液DNA进行ddPCR扩增的结果相吻合。结果表明,采用该ddPCR技术对沙门氏菌进行实际样品的定量检测具有可行性。
表4 人工污染样本中沙门氏菌的检测结果
Table 4 Test results of Salmonella in artificially contaminated samples
序号 | (CFU/mL) | (Ct值) | (CFU/mL) |
S1 | 5.3×101 | 未检出 | 5.2×101 |
S2 | 5.3×102 | 未检出 | 5.3×102 |
S3 | 5.3×103 | 32.3 | 5.25×103 |
S4 | 5.3×104 | 30.6 | 5,26×104 |
S5 | 5.3×105 | 28.8 | 5,29×105 |
S6 | 5.3×106 | 26.5 | 5.30×106 |
S7 | 5.3×107 | 24.5 | 5.30×107 |
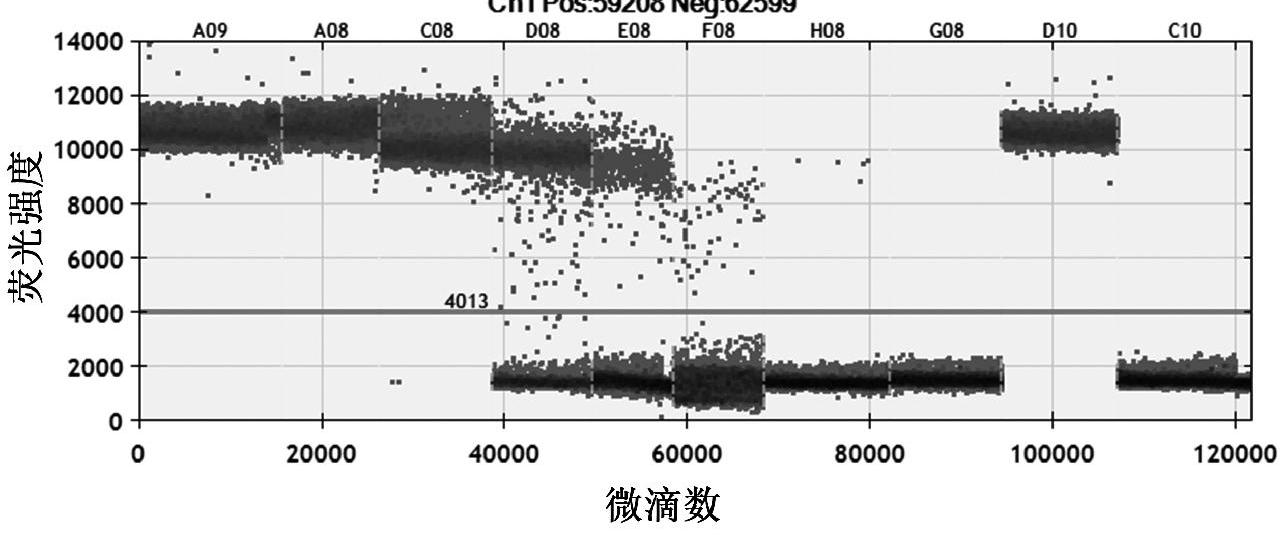
图6 微滴数字PCR方法实际样品检测结果
Fig.6 Actual sample detection results by ddPCR
3 结论
数字PCR技术以qPCR为基础,应用于特定基因及核酸的绝对定量检测,可直接计算出目标基因DNA分子的个数,是对起始样品的绝对定量。该方法适用于绝对定量或者qPCR中依赖Ct值不能很好分辨检测结果的情况[22-23]。
本试验结果显示,利用ddPCR技术开展沙门氏菌定量检测,可实现检测的有效基因组DNA浓度范围为2~20940拷贝/20 μL,检测的菌悬液浓度范围为5.3×101~5.3×105 CFU/mL。ddPCR检测方法与菌落总数测试片相比较,结果无显著性差异(p>0.05);当菌悬液浓度低至53 CFU/mL时,qPCR为阴性,而ddPCR最低检出菌液浓度为50 CFU/mL,ddPCR灵敏度更高且不依赖标准曲线和Ct值,可实现绝对定量[24]。
分析沙门氏菌的菌悬液浓度和ddPCR对应分析得到的Copy数,得到标准曲线R2≥0.99,表明线性关系良好。以人工添加沙门氏菌的鸡肉为实际样品进行ddPCR检测,样品定量结果与菌悬液定量结果基本一致,说明应用ddPCR技术对沙门氏菌开展定量检测具有可行性且具有一定的实际应用价值,可为沙门氏菌污染的快速定量检测提供技术支撑[25-26]。
参考文献
[1]朱恒文, 方艳红, 王元兰, 等. 肉鸡屠宰加工生产链中沙门氏菌的污染调查及ERIC-PCR溯源[J]. 食品科学, 2012, 33(17): 48-53.
[2]邵坤.山东省食源性沙门氏菌监测及分型研究[D]. 济南: 山东大学, 2011.
[3] Zhang H M, Shi L, Li L, et al. Identification and characterization of class 1 integron resistance gene cassettes among Salmonella strains isolated from healthy humans in China[J]. Microbiology and Immunology, 2004, 48(9): 639-645.
[4]黄玉柳. 食品中沙门氏菌的污染状况及预防措施[J]. 广东农业科学, 2010, 6: 225-226..
[5] 陈中婷, 杨松鑫, 张福鑫, 等. 基于金纳米球的肠炎沙门氏菌SERS免疫层析快速检测技术[J]. 畜牧与兽医, 2024, 56(5): 90-98.
[6]毕旺来, 赵巍薇, 马达, 等. 全基因组测序和real-time PCR法检测食源性沙门氏菌parC、gyrA基因突变特征[J]. 食品科学, 2022, 43(12): 296-302.
[7] Master C I, Shallcross J A, Mackey B M, et al. Effect of stress treatments on the detection of Listeria monocytogenes and enterotoxigenic Escherichia coli by the polymerase chain reaction[J]. Journal of Applied Bacteriology, 1994, 77(1): 73-79.
[8]杨纯佳, 张娟, 周臣清, 等. 数字PCR在食源性致病微生物检测中的应用研究进展[J]. 食品安全质量检测学报, 2016, 7(12): 4726-4730.
[8] Aricind A, Bhagwat A A. Simultaneous detection of Escherichia coli O157: H7, Listeria monocytogenes and Salmonella strains by real-time PCR[J]. International Journal of Food Microbiology, 2003(84): 217-224.
[10] Malorny B, Bunge C, Helmuth R. A real-time PCR for the detection of Salmonella enteritidis in poultry meat and consumption eggs[J]. Journal of Microbiological Methods, 2007, 70(2): 245-251.
[11] Seo K H, Valentin-Bon I E, Brackett R E. Detection and enumeration of Salmonella enteritidis in homemade ice cream associated with outbreak: comparison of conventional and real-time PCR methods[J]. Journal of Food Protection, 2006, 69(3): 639-643.
[12] Whyte P, Gill K M, Collins J, et al. The prevalence and PCR detection of Salmonella contamination in raw poultry[J]. Veterinary Microbiology, 2002, 89(1): 53-60.
[13]李春勇. 数字PCR技术原理及应用[J]. 生物技术世界, 2014,(3): 10-13.
[14] Cawthorn D M, Witthuhn R C. Selective PCR detection of viable Enterobacter sakazakii cells utilizing propidium monoazideor ethidium bromide monoazide[J]. Journal of Applied Microbiology, 2008, 105(4): 1178-1185.
[15]方佩佩, 赵丽青, 马云, 等. 副溶血性弧菌微滴数字PCR定量方法的建立[J]. 食品工业科技, 2018, 39(19): 252-256.
[16]禹思宇, 唐连飞, 陈雪琴, 等. 检测阴沟肠杆菌的数字PCR定量方法建立[J]. 畜牧与兽医, 2016, 48 (3): 1-4.
[17] Forghani F, Langaee T, Eskandari M, et al. Rapid detection of viable Bacillus cereus emetic and enterotoxic strains in food by coupling propidium monoazide and multiplex PCR(PMA-PCR)[J]. Food Control, 2015(55): 151-157.
[18] Wang L J, Ye C L, Xu H Y, et al. Development of an SD PMA-mPCR assay with internal amplification control for rapidand sensitive detection of viable Salmonella spp, Shigella spp. and Staphylococcus aureus in food products[J]. Food Control, 2015(57): 314-320.
[19] Wang L J, Li P, Zhang Z H, et al. Rapid and accurate detection of viable Escherichia coli O157: H7 in milk using acombined IMS, sodium deoxycholate, PMA and real-time quantitative PCR process[J]. Food Control, 2014, 36(1): 119-125.
[20] SN/T 1870—2016 出口食品中食源性致病菌检测方法实时荧光PCR法[S]. 北京: 中国标准出版社, 2016.
[21]赵丽青, 方佩佩, 唐静, 等. 数字PCR定量检测食品中单核细胞增生李斯特氏菌方法的研究[J]. 食品安全质量检测学报, 2017, 8(11): 4133-4138.
[22]董莲华, 张玲, 姜军, 等. 大肠杆菌O157: H7微滴数字PCR定量方法的建立[J]. 分析化学, 2015, 43: 319-324.
[23] GB 4789.4—2016 食品安全国家标准 食品微生物学检验 沙门氏菌检验[S]. 北京: 中国标准出版社, 2016
[24] Morisset D, Stebih D, Milavec M, et al. Quantitative analysis of food and feed samples with droplet digital PCR[J]. PLoS One, 2013, 8(5): 62583-62588.
[25] George D, Czech J, et al. Detection and quantification of chimerism by droplet digital PCR[J]. Chimerism, 2013, 4(3): 102-108.
[26] Beck J, Bierau S, Balzer S, et al. Digital droplet PCR forrapid quantification of donor DNA in the circulation of transplant recipients as a potential universal biomarker of graft injury[J]. Clinical Chemisty, 2013, 59(12): 1732.
表3 ddPCR方法定量检测梯度稀释的沙门氏菌纯菌液结果
Table 3 Quantitative detection of gradient diluted Salmonella by ddPCR
样品 | 3M纸片法 (CFU/mL) | ddPCR拷贝数/20 L | C-根据ddPCR拷贝数换算的菌悬液浓度 (CFU/mL) | 相对标准偏差 (RSD) | Ct值 |
1 | (5.3±0.1)×107 | / | / | 22.54 | |
2 | (5.3±0.1)×106 | / | / | 25.87 | |
3 | (5.3±0.1)×105 | 20940 | 523500 | 0.0410 | 28.85 |
4 | (5.3±0.1)×104 | 2100 | 52500 | 0.0067 | 30.10 |
5 | (5.3±0.1)×103 | 208 | 5200 | 0.013 | 32.30 |
6 | (5.3±0.1)×102 | 19.6 | 490 | 0.055 | 35.10 |
7 | (5.3±0.1)×101 | 2.0 | 50 | 0.077 | 40.03 |
8 | 5.0×100 | / | / | / | / |
图4 菌悬液浓度与ddPCR拷贝数的线性关系
Fig.4 Linear relationship between bacterial suspension concentration and ddPCR copy number







