CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
氯霉素高效降解菌分离鉴定及其特性研究
作者:秦富 李娟 吴良萍 麻晓莉 包昌健 苏华 汪文龙 罗兆飞
秦富 李娟 吴良萍 麻晓莉 包昌健 苏华 汪文龙 罗兆飞
摘 要 本研究筛选可高效降解氯霉素的微生物,并研究其降解特性。选用氯霉素高残留的花甲螺进行培养驯化,使用含氯霉素的营养琼脂平板对目标菌进行筛选分离;采用显微镜对目标菌进行形态学观察,使用全自动微生物分析系统对目标菌株进行生化鉴定,同时利用细菌16S rDNA序列测序的方法对目标菌株进行菌种鉴定;利用高效液相色谱-串联质谱法测定样品和反应物中氯霉素的含量,在不同反应时间、温度、pH、底物浓度、菌悬液含量等条件下,进行降解特性研究。最终,驯化分离一株具有降解氯霉素作用的菌株,代号SA-cd1,经形态特征、生理生化和16S rDNA序列比对结果鉴定为海藻希瓦氏菌(Shewanella algae)。SA-cd1菌降解氯霉素的温度为36~50℃,pH为6.0~9.0,0.1 mg/L氯霉素5 min的降解率为83.8%,降解率随菌悬液浓度增大而升高,随反应时间变长而升高;底物浓度提高,降解率有略微下降,10 mg/L氯霉素1 h内降解率达92.5%;除去菌体的上清液仍然具有较强的降解率,为47.3%。SA-cd1海藻希瓦氏菌分离培养容易,对氯霉素的降解效率高,适宜的温度和pH条件宽泛,可应用于环境中氯霉素的降解清除以及相关降解工程菌、降解酶的研发。
关键词 驯化筛选;海藻希瓦氏菌;生物降解;净化技术
Study on Isolation, Identification and Degradation Characteristics of Chloramphenicol Degrading Bacteria
QIN Fu1 LI Juan1 WU Liang-Ping1 MA Xiao-Li1
BAO Chang-Jian2 SU Hua1 WANG Wen-Long1 LUO Zhao-Fei1*
Abstract This study focuses on screening out the microorganisms that can degrade chloramphenicol efficiently and analyzing their degradation characteristics.The Paphia undulata with high chloramphenicol residue was cultured and domesticated. The target bacteria was screened and separated by nutrient agar plate containing chloramphenicol. In this research, a microscope was used to observe morphology of the target bacteria; a fully automatic microbial analysis system was adopted to conduct biochemical identification of the target strains; and bacterial 16S rDNA sequencing was used to identify the target strains. The content of chloramphenicol in samples or reactants was determined by high performance liquid chromatography tandem mass spectrometry. The degradation characteristics were studied under different reaction time, temperature, pH, substrate concentration and bacterial suspension content. A strain named SA-cd1, which can degrade chloramphenicol efficiently, was obtained by domestication and isolation. The bacteria was identified as Shewanella algae according to its morphological characteristics, physiological and biochemical characteristics and 16S rDNA sequencing. The optimal temperature and pH for chloramphenicol degradation of SA-cd1 was 36-50℃ and 6.0-9.0. The degradation rate of 0.1 mg/L chloramphenicol in 5 min reached 83.8%, and rose with the increase of the concentration of the bacterial suspension or the reaction time. The degradation rate did not decrease significantly with the increase of the substrate concentration, and the degradation rate of 10 mg/L chloramphenicol in 1 h was 92.5%. The supernatant after the removal of bacteria still had a strong degradation rate of 47.3%. SA-cd1 is easy to isolate and culture, with high degradation efficiency to chloramphenicol, as well as a wide range of suitable temperature and pH conditions, thus it can be applied to the degradation and removal of chloramphenicol in the environment, as well as the development of related degradation engineering bacteria and enzymes.
Keywords domestication and screening; Shewanella algae; biodegradation; purification technology
基金项目:海关总署科研项目(2020HK206)
第一作者:秦富(1987—),男,汉族,广西玉林人,硕士,工程师,主要从事食品安全检测与研究工作,E-mail: 514623946@qq.com
通信作者:罗兆飞(1978—),男,汉族,广西玉林人,硕士,高级兽医师,主要从事食品安全检测与研究工作,E-mail: 975558@qq.com
1. 南宁海关技术中心 南宁 530021
2. 广西医科大学 南宁 530021
1. Technical Center of Nanning Customs District, Nanning 530021
2. Guangxi Medical University, Nanning 530021
氯霉素属于氯代硝基芳香族类抗生素,1949年被人工合成后广泛应用于动物兽药和人类医学领域。氯霉素对人体健康具有较大影响,不仅会通过损坏造血功能引起再生障碍性贫血,而且可能具有致癌性和遗传毒性[1-2]。氯霉素滥用还会引起抗性基因的发生进而产生耐药细菌[3-4],对环境微生物生态系统造成严重影响。因此,氯霉素在2002年开始被禁止用于动物养殖过程,但是由于氯霉素的价格非常低廉,而且抗菌效果非常好,仍然存在违规使用的情况。据报道,我国东湖[5]、珠江和长江[6]、闽江[7]、博斯腾湖和乌伦古湖[8]等水域均发现有氯霉素类抗生素残留。其主要原因是工业[9]、医疗和养殖等行业的废物废水[10]中含有较高浓度的氯霉素未被吸附或降解处理而排放至环境和水源中。所以,如何采用有效的措施对废水中的氯霉素进行降解,是当前急需解决的水环境污染防治问题。
目前,废水中氯霉素的降解消除方法主要包括生物法、化学法以及物理法。过滤、吸附、絮凝和沉淀等传统的处理工艺实际上并不能有效地降解或去除废水中的氯霉素[11-12]。常见的高级氧化还原法,如电催化[13]、亚硫酸盐[14]和光催化[15],不仅需要使用大量的氧化还原试剂,而且需要额外投入能量。微生物燃料电池法[16]和臭氧技术[17]等新型技术去除氯霉素的效率较好,但是这些新技术方法仍然处于研究试验阶段。而特异性降解菌群法,不仅降解消除能力强,而且简单、环保。Zhao等[18]分离获得5株氯霉素降解菌株,55 mg/L氯霉素12 d内的降解率为84.0%。Ma等[19]从活性污泥中分离出一株氯霉素矿化菌群联合体,50 mg/L氯霉素5 d内矿化率为71.5%。Zhang等[20]分离的以氯霉素为唯一碳源和能量源的菌群联合体可在5 d内彻底降解120 mg/L氯霉素,矿化率达90%。其他氯霉素降解菌群还包括假单胞菌属[21-22]、金属地杆菌[23]和鞘氨醇单胞菌[24]等。然而,这些菌株降解氯霉素的过程需要依赖于菌体的生长,而且降解条件要求较高,难以实现广泛应用。为了筛选出降解效率高、降解条件简单的氯霉素降解菌,本研究利用含有高浓度氯霉素的花甲螺样品来进行驯化培养,对分离鉴定的菌株进行降解反应时间、温度、pH、底物浓度等特性分析,对菌体及其代谢产物(上清液)的降解能力进行分析,以期为废水等环境载体中氯霉素的降解和消除提供参考菌源。
1 材料与方法
1.1 试验材料
1.1.1 样品
花甲螺(含有4.3 mg/kg氯霉素)为实验室检测余样。
1.1.2 试剂
营养琼脂(Nutrient Agar,NA)和营养肉汤(Nutrient Broth,NB)购自北京陆桥公司;氯霉素标准品(99.2%)购自德国Dr.Ehrenstorfer公司;氯霉素-D5同位素内标(100 mg/L)购自天津阿尔塔科技公司;乙酸乙酯(色谱纯)购自德国CNW公司。
1.1.3 仪器
Agilent 1290- AB 4500高效液相色谱-串联质谱仪(美国Agilent公司和美国SCIEX公司);KL512氮吹仪(北京康林科技公司);VITEK 2 Compact全自动微生物分析系统(法国梅里埃公司);MIR-253恒温培养箱(日本三洋公司);ZQZY-BS8恒温振荡培养箱(上海知楚仪器公司);DM6000生物显微镜(德国Leica公司);S210 pH酸度计(上海梅特勒公司);TprofessionalTrio PCR仪(德国Biometra公司);EnduroTm Gel XL 电泳仪(美国Labnet公司);AlphaImager HP 凝胶成像系统(美国阿尔法公司)。
1.2 方法
1.2.1 样品处理
将花甲螺样本洗净去壳后,挑取螺肉进行打碎匀浆后备用。
1.2.2 氯霉素降解菌的驯化培养
取3 g花甲螺肉匀浆样品,测定样品中氯霉素的含量,然后再取10 g样品置于培养皿中,于36℃培养箱驯化培养,每隔24 h从中随机称取3 g用来检测氯霉素的含量。
1.2.3 氯霉素降解菌的筛选分离
首先,花甲螺肉匀浆样品经过48 h培养后,用接种环蘸取培养物的浸出液接种至添加有10 mg/L氯霉素的营养琼脂平板上,于36℃培养箱中培养24 h。其次,将菌落接种至新的添加有氯霉素的营养琼脂平板上,36℃培养24 h,按照相同步骤重复进行接种2次。最后,挑取优势单菌落接种至100 mL营养肉汤中继续培养24 h,将菌株用瓷珠保存,命名为SA-cd1菌株。
1.2.4 SA-cd1菌株生化鉴定
将SA-cd1菌株进行革兰氏染色,然后用显微镜对其形态进行观察。将SA-cd1菌株用0.45%氯化钠溶液稀释至0.5个麦氏浊度单位(McFarland,MCF),填充至GN卡,用全自动微生物分析系统进行生化鉴定。
1.2.5 SA-cd1菌株分子生物学鉴定
采用加热法提取细菌DNA:刮取一环菌落悬浮于200 μL无菌水中,涡旋振荡15 s,沸水浴5 min,冷却后,于10000 r/min离心5 min,吸取适量上清液用于PCR反应。参考赵洪涛等[25]的方法,使用16S rDNA通用引物5'-AGAGTTTGATCCTGGCTCAG-3'和5'-GGTTACCTTGTTACGACTT-3',制备25 μL的反应体系:10 μmol/L上游和下游引物各0.5 μL、10 x buffer 2.5 μL、2.5 μmol/LdNTP 2 μL、DNA聚合酶1 μL、DNA模板2 μL、ddH2O 16.5 μL。用PCR扩增仪进行扩增,扩增程序:于94℃条件预变性5 min,再于94℃条件变性30 s,然后于56℃条件退火30 s,接着于72℃条件延伸1 min,循环30次后,于72℃条件延伸10 min。PCR反应结束后,将反应产物用1.5%琼脂凝胶进行电泳检测,然后将样本送至广州艾基生物公司进行DNA测序,测试结果在GenBank数据库进行同源性比对。
1.2.6 不同反应时间氯霉素降解率分析
分别取10 mL菌悬液至6支50 mL无菌管中,再分别加入0.2 mL氯霉素(5 mg/L),于36℃恒温水浴中分别反应1 min、5 min、10 min、20 min、30 min、60 min,加入乙酸乙酯终止反应,然后取反应物检测氯霉素的含量。另取10 mL 营养肉汤,加入相同剂量的氯霉素作为试验对照组(Control,Ctrl)。
1.2.7 不同反应温度和pH氯霉素降解率分析
分别取10 mL菌悬液于7支50 mL无菌管中,再分别置于4℃、15℃、30℃、36℃、40℃、45℃、50℃、60℃、80℃冰箱冰浴或恒温水浴中适温5 min,分别加入0.2 mL 浓度为5 mg/L的氯霉素水溶液,继续培养10 min后加入终止液乙酸乙酯,取反应物检测氯霉素的含量。另取7支无菌管,每管加入10 mL菌悬液,分别用0.2 mol/L NaOH溶液或2 mol/L HCl溶液调节pH至4.0、5.0、6.0、7.0、8.0、9.0、10.0,加入等量氯霉素,于36℃恒温水浴中培养10 min,加入终止液乙酸乙酯,取反应物检测氯霉素的含量。每种处理都取10 mL营养肉汤,加入氯霉素作为对照组。
1.2.8 氯霉素底物浓度不同对氯霉素降解率的影响分析
取适量菌悬液,用弱酸小心调节pH至6.0,分装至7支无菌管中,每管10 mL。分别加入0.2 mL浓度分别是0 mg/L、0.5 mg/L、1 mg/L、2.5 mg/L、5 mg/L、50 mg/L、500 mg/L氯霉素,于50℃恒温水浴中培养60 min,加入终止液乙酸乙酯,取反应物检测氯霉素的含量。另取10 mL NB,加入0.2 mL氯霉素(5 mg/L)作为对照组。
1.2.9 菌悬液含量不同对氯霉素降解率的影响分析
分别取0 mL、0.25 mL、0.5 mL、1.0 mL、2.5 mL、5 mL、10 mL菌悬液,加入10 mL、9.75 mL、9.5 mL、9 mL、7.5 mL、5 mL、0 mL NB混匀,然后加入0.2 mL氯霉素(5 mg/L),于36℃培养10 min后加入乙酸乙酯终止反应,测定氯霉素含量。另取10 mL营养肉汤,加入等量氯霉素作为对照组。
1.2.10 除去菌体后的上清液降解氯霉素效果分析
取10 mL菌悬液作为菌悬液组;再取10 mL菌悬液,高速离心10 min,移取上清液当作上清液组;离心后的沉淀(菌体)用无菌水冲洗2次后,加入10 mL营养肉汤混匀的菌体组;取10 mL营养肉汤作为对照组。各组均加入0.2 mL氯霉素(5 mg/L),于36℃恒温水浴中培养10 min,加入终止液乙酸乙酯,然后取反应物检测氯霉素的含量。
1.2.11 氯霉素含量测定及降解率计算
螺肉中氯霉素的残留量参照文献方法[26]进行测定。降解反应物中氯霉素测定:每次反应后均加入10 mL乙酸乙酯终止反应,然后加入100 ng氯霉素-D5同位素内标,加入无水硫酸钠盐析分层,离心后取0.5 mL上清液于45℃水浴条件下进行氮气,氮吹干后加入1 mL超纯水进行复溶,过0.22 μm滤膜,供液相色谱-串联质谱测定,以Ctrl组为对照进行同位素内标法定量,并计算氯霉素降解率,各组降解率= 100%-(各组反应物中氯霉素含量/Ctrl组中氯霉素含量×100%)。
1.3 统计分析
试验数据采用统计软件SPSS 17.0进行统计分析,两两比较用t检验,多组比较用方差分析,以p<0.05为差异有显著性。
2 结果与分析
2.1 驯化培养前后螺肉中氯霉素的降解情况
螺肉样本培养驯化48 h后,螺肉中氯霉素的含量由原来的4.3 mg/kg下降至0.0045 mg/kg,降解了99.9%,提示样本中驯化出了能够降解氯霉素的细菌。
2.2 菌株的分离和鉴定结果
本试验分离筛选获得2株菌落形态不同的菌株。其中,1株不能降解氯霉素;另1株(SA-cd1)具有快速降解氯霉素的能力,如图1所示,细胞形态为杆状,革兰氏染色呈阴性,属于革兰氏阴性杆菌。经全自动微生物鉴定系统鉴定,结果提示为海藻希瓦氏菌(Shewanella algae),其具体生化试验结果见表1。PCR扩增序列(1407 bp)于GenBank数据库上进行比对,结果提示该菌与Shewanella algae(Sequence ID: KC210850.1)的同源性为100%。

图1 SA-cd1革兰氏染色结果
Fig.1 The result of Gram staining of SA-cd1
2.3 SA-cd1降解氯霉素的效率
本研究首先通过改变降解反应时间来分析降解菌的降解效率。结果如图2所示,降解菌的降解率随着反应时间延长而逐渐升高,5 min降解率达83.8%。

图2 反应时间对降解率的影响
Fig.2 The effect of reaction time on degradation rate
2.4 不同温度和pH条件下,SA-cd1的降解效果
为了进一步分析研究SA-cd1降解氯霉素反应的特性,我们分析了在不同温度和pH条件下,0.1 mg·L-1氯霉素降解率的变化。结果如图3所示,降解率随着温度升高而升高,在36~50℃范围内均较高,超过50℃后开始下降,80℃时,降解率为1.5%;在pH为6.0~9.0的范围内,氯霉素的降解率均较高(大于85.7%),pH继续降低或升高,其降解率均明显下降(图4)。结果表明SA-cd1降解氯霉素的反应需要适合的温度和pH条件,并且反应时间短,反应效率高,属于典型的酶促反应。当温度过低时酶的活性降低,导致氯霉素降解率显著下降;过高的温度或者强酸强碱使酶失去活性,同样可以导致氯霉素降解率显著下降。结果提示SA-cd1降解氯霉素的机制是通过产生酶类物质来起作用的。

图3 温度对降解率的影响
Fig.3 The effect of temperature on degradation rate

图4 pH对降解率的影响
Fig.4 The effect of pH on degradation rate
2.5 氯霉素底物浓度对降解率的影响
通过选择在最适pH(6.0)、最适温度(50℃)和反应时间为60 min的条件下进行试验,测定SA-cd1对不同浓度氯霉素底物的降解率,以分析SA-cd1的降解能力。结果显示,随着底物浓度逐渐升高,降解率略有下降,但在0.01~10 mg·L-1范围内均维持在较高水平,均大于92.5%,如图5所示。

图5 底物浓度对降解率的影响
Fig.5 The effect of substrate concentration on degradation rate
2.6 菌悬液含量对降解率的影响
为了后续将菌悬液应用于实际样品(如水样、粪肥、土壤等)中氯霉素的快速降解净化,本试验选择在氯霉素底物浓度为0.1 mg/L,不调节菌悬液pH(约为7.0),反应温度为36℃,反应时间为10 min条件下,分析不同含量的SA-cd1菌悬液对氯霉素的降解效果。结果如图6所示,降解率随着菌悬液含量增加而升高,含量添至10%时,降解率为91.9%,含量添至25%时,降解率达95.2%,超过25%后降解率不再升高。

图6 菌悬液含量对降解率的影响
Fig.6 The effect of bacterial suspension content on degradation rate
2.7 去除菌体的上清液降解氯霉素的效果
为进一步验证SA-cd1对氯霉素的降解反应属于酶促反应,不依赖于细菌的生长和繁殖,笔者通过高速离心把菌体和上清液分离后进行氯霉素降解试验。结果如图7所示,菌体组的降解率最低,为3.8%,上清液组的降解率为47.3%,低于菌悬液组。该结果进一步提示SA-cd1是通过生产释放酶类物质来达到降解氯霉素的作用。上清液组降解率下降,可能是由于菌体在无氯霉素刺激作用时产生释放的酶物质含量低,不足以将氯霉素降解完全。而菌体组降解率较低,原因是菌体产生释放的酶被清洗,加入培养基复溶后短时间内无法恢复活力生产释放酶来降解氯霉素。
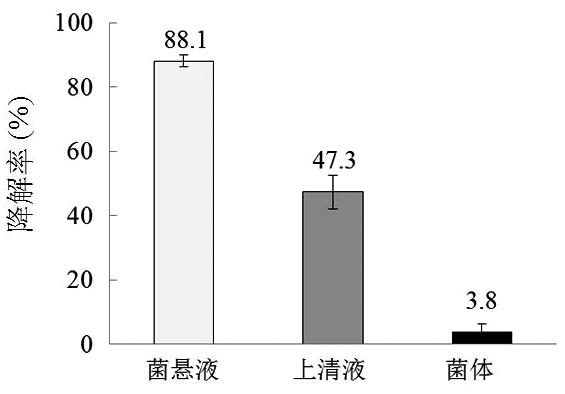
图7 上清液和菌体的降解率
Fig.7 Degradation ratio of supernatant and thallus
3 讨论
本课题组在对氯霉素进行日常检测过程中发现腐败的花甲螺可以将高含量的氯霉素进行快速降解,据此利用含有高浓度氯霉素的花甲螺样品,经过系统驯化、分离培养获得一株氯霉素降解菌SA-cd1,经鉴定为海藻希瓦氏菌。经查阅,此前文献并未见相关报道。SA-cd1氯霉素降解菌具有快速、灵敏、高效,温度、pH条件宽泛,可不依赖菌体的生长活力等特点,明显优于文献报道的降解菌[18-20]。更重要的是,SA-cd1的代谢物可独立降解氯霉素,提示代谢物中存在氯霉素降解酶参与氯霉素的降解。尽管在试验条件中SA-cd1的代谢产物(上清液)的降解率只有47.3%,但是后续研究若能将该酶类物质进行提取、纯化,其降解率将会大幅上升。已有研究表明细菌降解氯霉素的原理主要有两种途径,分别是氯霉素的酰胺键被水解产生对硝基苯丝氨酸和氯霉素的硝基被还原生成氨基氯霉素,其中涉及的两种关键酶分别为酰胺水解酶和硝基还原酶。据报道,大肠埃希氏菌[18, 27]和诺卡氏菌[28]能够产生酰胺水解酶将氯霉素转化为对硝基苯丝氨酸或硝基苯甲酸。流感嗜血杆菌[29-30]可以表达硝基还原酶,并将氯霉素还原成氨基氯霉素。另外,氯霉素乙酰转移酶(Chloramphenicol Acetyltransferase,CAT)将氯霉素的3-羟基乙酰化产生3-醋酸氯霉素,是细菌对氯霉素耐药的常见机制[31-32]。本研究仅对氯霉素高效降解菌SA-cd1的分离培养和降解特性进行了初步探讨,研究结果表明SA-cd1对氯霉素具有很高的降解效率,其降解方式属于酶促反应,但是具体由哪一种酶起降解作用尚不清楚。未来,本研究将继续对SA-cd1的全基因序列及其代谢产物进行深入研究,以分析其降解氯霉素的分子机制和原理,为SA-cd1应用于水体、土壤等环境载体中的氯霉素降解清除以及相关降解工程菌、降解酶的研发应用提供理论依据。
4 结论
本研究利用含有高浓度氯霉素的花甲螺样本进行培养、分离、驯化和鉴定后获得一株对氯霉素具有较高降解能力的海藻希瓦氏菌SA-cd1。该菌降解氯霉素的反应类似于酶促反应,降解反应灵敏、迅速、高效,所需温度和pH条件宽泛,其代谢物同样能够快速降解氯霉素,可以为环境中氯霉素降解清除以及相关降解工程菌、降解酶的研发提供理论依据。
参考文献
[1] Nguyen L M, Nguyen N T T, Nguyen T T T, et al. Occurrence, toxicity and adsorptive removal of the chloramphenicol antibiotic in water: a review[J]. Environmental Chemistry Letters, 2022(20): 1929-1963.
[2] Hanekamp J C, Bast A. Antibiotics exposure and health risks: Chloramphenicol[J]. Environmental Toxicology & Pharmacology, 2015, 39(1): 213-220.
[3] Zhang Y, Guo P, Wang M, et al. Mixture toxicity effects of chloramphenicol, thiamphenicol, florfenicol in Daphnia magna under different temperatures[J]. Ecotoxicology, 2021(30): 31-42.
[4] Aali R, Baragh S, Asgari E, et al. Tracking of chloramphenicol, erythromycin, and sulfamethoxazole antibiotic-resistant bacteria from untreated wastewater effluents to receiving river[J]. Environmental Health Engineering and Management Journal, 2019, 6(2): 89-96.
[5] 王璠,刘彬,郭丽.武汉市东湖抗生素污染现状及生态风险评估[J].环境监控与预警, 2022 (5): 176-182.
[6] 章强, 辛琦, 朱静敏,等.中国主要水域抗生素污染现状及其生态环境效应研究进展[J].环境化学, 2014, 7(33): 1075-1083.
[7] 刘四光,张乐,蒙黄智,等.闽江河口区水体抗生素污染特征及风险评价[J].渔业研究, 2022, 44(5): 426-443.
[8] Lu P, Fang Y, Barvor J B, et al. Review of antibiotic pollution in the seven watersheds in China[J]. Polish Journal of Environmental Studies, 2019, 28 (6): 4045-4055.
[9] 国彬, 姚丽贤, 冯志诚, 等. 广州市污水厂氯霉素类抗生素污染特征研究[J].杭州化工, 2011, 41(2): 15-17.
[10] 邱文倩, 林坚, 华永有. 水产养殖水环境氯霉素污染状况研究[J]. 中国公共卫生, 2019, 35(2): 246-249.
[11] 李书宁, 何德富, 陈瑞鹏, 等. 基于氨基化二氧化硅微球表面印迹聚合物高效去除水中氯霉素的研究[J]. 食品安全质量检测学报, 2022(9): 2840-2847.
[12] Li Y, Zhang J, Liu H. Removal of Chloramphenicol from Aqueous Solution Using Low-Cost Activated Carbon Prepared from Typha orientalis[J]. Water, 2018, 10(4): 351-367.
[13] 李靖宇, 王宪森, 王晓林, 等. 载铁活性炭颗粒电极降解氯霉素污染废水性能研究[J].江西化工, 2021, 37(5): 56-58.
[14] Chen X Y, Miao W, Yang Y L, et al. Aeration-assisted sulfite activation with ferrous for enhanced chloramphenicol degradation[J]. Chemosphere, 2019(238): 124599.1-124599.9.
[15] Karthik R, Vinoth Kumar J, Chen S M, et al. A study of electrocatalytic and photocatalytic activity of cerium molybdate nanocubes decorated graphene oxide for the sensing and degradation of antibiotic drug: chloramphenicol[J]. Acs Applied Materials & Interfaces, 2017, 9(7): 6547-6586.
[16] Zhang Q H, Zhang Y Y, Li D P. Cometabolic degradation of chloramphenicol via a meta-cleavage pathway in a microbial fuel cell and its microbial community[J]. Bioresource Technology, 2017(229): 104-110.
[17] 刘行浩, 张凤琳, 胡淑恒, 等. 臭氧降解水中氯霉素影响因素及机制研究[J]. 合肥工业大学学报 (自然科学版), 2022(3): 383-388.
[18] Zhao X, Tian F, Wang G, et al. Isolation, identification and characterization of human intestinal bacteria with the ability to utilize chloramphenicol as the sole source of carbon and energy[J]. Fems Microbiology Ecology, 2012, 82(3): 703-712.
[19] Ma X D, Qi M Y, Li Z L, et al. Characterization of an efficient chloramphenicol-mineralizing bacterial consortium[J]. Chemosphere, 2019 (222): 149-155.
[20] Zhang J Y, Zhao R X, Cao L J, et al. High-efficiency biodegradation of chloramphenicol by enriched bacterial consortia: Kinetics study and bacterial community characterization[J]. Journal of Hazardous Materials, 2020(384): 1-11.
[21] 吕玄文, 冯茜丹, 党志. 氯霉素降解菌的驯化、筛选及分离鉴定[J]. 农业环境科学学报, 2007, 26(4): 1477-1481.
[22] 向红, 刘欢, 李磊, 等. 氯霉素降解菌的筛选与降解性能[J]. 医学动物防制, 2017, 33(8): 811-814.
[23] Xu H D, Xiao L L, Zheng S L, et al. Reductive degradation of chloramphenicol by Geobacter metallireducens[J]. Science China-Technological Sciences, 2019, 62(10): 1688-1694 .
[24] Zhang J Y, Gan W H, Zhao R X, et al. Chloramphenicol biodegradation by enriched bacterial consortia and isolated strain Sphingomonas sp. CL5.1: The reconstruction of a novel biodegradation pathway[J]. Water Research, 2020(187): 1-12.
[25] 赵洪涛, 陈东奎, 陈香玲, 等. 沃柑溃疡病病原菌分离鉴定及防治药剂筛选[J]. 南方农业学报, 2019, 50 (12): 2703-2712.
[26] 汪文龙, 黄大新, 刘军义, 等. UPLC-MS/MS测定水产品中氯霉素残留量[J]. 中国卫生检验杂志, 2009, 19(8): 1767-1769.
[27] Tao W X, Lee M H, Wu J, et al. Inactivation of chloramphenicol and florfenicol by a novel chloramphenicol hydrolase[J]. Applied And Environmental Microbiology, 2012, 78 (17): 6295-6301.
[28] 赵梦君. 氯霉素降解菌株LMS-CY的分离鉴定、降解特性及其初步应用研究[D]. 南京:南京农业大学, 2017.
[29] Smith A L, Erwin A L, Kline T, et al. Chloramphenicol is a substrate for a novel nitroreductase pathway in Haemophilus influenzae[J]. Antimicrob. Agents Chemother, 2007, 51(8): 2820-2829.
[30] Crofts T S, Sontha P, King A O. Discovery and characterization of a nitroreductase capable of conferring bacterial resistance to chloramphenicol[J]. Cell Chemical Biology, 2019, 26 (4): 559-570.
[31] Mosher R H, Ranade N P, Schrempf H, et al. Chloramphenicol resistance in Streptomyces: cloning and characterization of a chloramphenicol hy-drolase gene from Streptomyces venezuelae[J]. Journal Of General And Applied Microbiology, 1990, 136(2): 293-301.
[32] Mosa A, Hutter M C, Zapp J, et al. Regioselective acetylation of C21 hydroxysteroids by the bacterial chloramphenicol acetyltransferase I[J]. Chembiochem, 2015, 16(11): 1670-1679.
表1 SA-cd1的生化试验结果
Table 1 The result of biochemical test of SA-cd1
孔号 | 生化指标 | 试验结果 | 孔号 | 生化指标 | 试验结果 |
2 | Ala-Phe-Pro-ARYLAMIDASE | + | 33 | 蔗糖 SACCHAROSE/SUCROSE | - |
3 | 侧金盏花醇 ADONITOL | - | 34 | D-塔格糖 D-TAGATOSE | - |
4 | 吡咯烷基芳胺酶 L-Pyrrolydonl-ARYLAMIDASE | + | 35 | D-海藻糖 D-TREHALOSE | - |
5 | L-阿拉伯醇 L-ARABITOL | - | 36 | 柠檬酸盐 (钠) CITRATE (SODIUM) | - |
7 | D-纤维二糖 D-CELLOBIOSE | - | 37 | 丙二酸盐 MALONATE | - |
9 | β-半乳糖苷酶 BETA-GALACTOSIDASE | - | 39 | 5-酮-葡萄糖苷 5-KETO-D-GLUCONATE | - |
10 | H2S产生 H2S PRODUCTION | + | 40 | L-乳酸产碱 L-LACTATE alkalinisation | - |
11 | BETA-N-ACETYL-GLUCOSAMINIDASE | + | 41 | α-葡萄糖 ALPHA-GLUCOSIDASE | - |
12 | 谷氨酰芳胺酶 pNAGIutamyl Arylamidase Pna | + | 42 | 琥珀酸盐产碱 SUCCINATE alkalinisation | - |
13 | D-葡萄糖 D-GLUCOSE | - | 43 | Beta-N-ACETYL-GALACTOSAMINIDASE | - |
14 | GAMMA-GLUTAMYL-TRANSFERASE | - | 44 | α-半乳糖苷酶 ALPHA-GLUCOSIDASE | - |
15 | 葡萄糖发酵 FERMENTATION/GLUCOSE | - | 45 | 磷酸酶 PHOSPHATASE | + |
17 | β-葡萄糖苷酶 BETA-GLUCOSIDASE | - | 46 | 氨基乙酸芳胺酶 Glycine ARYLAMIDASE | - |
18 | D-麦芽糖 D-MALTOSE | - | 47 | ORNITHINE DECARBOXYLASE | - |
19 | D-甘露醇 D-MANNITOL | - | 48 | 赖氨酸脱羧酶 DECARBOXYLASE BASE | - |
20 | D-甘露糖 D-MANNOSE | - | 53 | 组氨酸同化 L-HISTIDINE assimilation | - |
21 | β-木糖苷酶 BETA-XYLOSIDASE | - | 56 | 库马酸盐 COURMARATE | - |
22 | β-丙氨酸芳胺酶 pNABETA-Alanine arylamidase Pna | - | 57 | β-葡萄糖苷酸酶 BETA-GLUCORONIDASE | - |
23 | L-脯氨酸芳胺酶 L-Proline ARYLAMIDASE | + | 58 | RESISTANCE (comp.vibrio.) | - |
26 | 脂酶 LIPASE | - | 59 | Glu-Gly-Arg-ARYLAMIDASE | + |
27 | 古老糖 PALATINOSE | - | 61 | L-苹果酸盐同化 L-MALATE assimilation | - |
29 | 酪氨酸芳胺酶 Tyrosine ARYLAMIDASE | + | 62 | ELLMAN | - |
31 | 尿素酶 UREASE | + | 64 | L-乳酸盐同化 L-LACTATE assimilation | - |
32 | D-山梨醇 D-SORBITOL | - |
注: “+”表示生化反应阳性; “-”表示生化反应阴性







