CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
基于双靶点杂交链式反应的大肠杆菌核酸检测研究
作者:扈卓然 庄妙慧 杨 晨 翟若男 杨倩思茹 郑雅芳 胡善文
扈卓然 庄妙慧 杨 晨 翟若男 杨倩思茹 郑雅芳 胡善文
摘 要 本研究建立了一种以杂交链式反应为基础的双靶点检测大肠杆菌核酸的新方法。以大肠杆菌O157:H7的两段特异性核酸序列为靶标,设计了带有FAM荧光基团和猝灭基团标记的发卡探针,通过杂交链式反应扩增靶标序列,并优化了反应条件。结果表明,在0.15 mol/L Na+盐浓度和0.02 mol/L HEPES缓冲体系下,该方法具有良好的稳定性和选择性,幽门螺旋杆菌、志贺氏菌、金黄色葡萄球菌及沙门氏菌均不产生干扰信号。检测在大肠杆菌靶标核酸浓度为0.001~20 nmol/L范围内显示出良好的线性关系,检出限为0.001 nmol/L。在实际样本检测中,该方法能准确区分真实阳性样本,有望为大肠杆菌检测提供一种新技术。
关键词 大肠杆菌;杂交链式反应;双靶点检测
Research on Escherichia coli DNA Analysis Based on Dual-target Triggered Hybridization Chain Reaction
HU Zhuo-Ran1 ZHUANG Miao-Hui2 YANG Chen1 ZHAI Ruo-Nan1
YANG Qian-Si-Ru1 ZHENG Ya-Fang1 HU Shan-Wen1*
Abstract This study establishes a novel method for detecting Escherichia coli nucleic acids using a dual-target triggered hybridization chain reaction (HCR). By targeting two specific nucleic acid sequences from the gene of Escherichia coli O157:H7, hairpin probes labeled with FAM fluorescent group and quencher group were designed. The target sequences were amplified through HCR and reaction conditions were optimized. Results indicated that under the condition of 0.15 mol/L Na+ in 0.02 mol/L HEPES buffer, the method exhibited good stability and selectivity, with no interference signals from Helicobacter pylori, Shigella, Staphylococcus aureus, or Salmonella. The detection method showed a good linear relationship within the concentration range of 0.001 to 20 nmol/L, with a detection limit of 0.001 nmol/L. In actual sample testing, this method accurately distinguished true positive samples, offering a promising new approach for Escherichia coli detection.
Keyword Escherichia coli; hybridization chain reaction; dual-target detection
基金项目:福建医科大学公共卫生学院“大学生创新创业训练计划项目”(xy202410014)
第一作者:扈卓然(2005—),女,汉族,山东东营人,本科,主要从事卫生检验与检疫研究工作,Email: hzrydfp050403@163.com
通信作者:胡善文(1989—),男,汉族,山东济宁人,博士,副教授,主要从事核酸检测和卫检新方法研究工作,E-mail: shanwenhu@fjmu.edu.cn
1. 福建医科大学公共卫生学院 福州 350122
2. 厦门国际旅行卫生保健中心(厦门海关口岸门诊部) 厦门 361001
1. School of Public Health, Fujian Medical University, Fuzhou 350122
2. Xiamen International Travel Health Care Center (Xiamen Customs Port Outpatient Department), Xiamen 361001
大肠杆菌O157:H7(Escherichia coli O157:H7,E. coli O157:H7)是一种重要的肠道病原体,是肠出血性大肠杆菌的主要致病血清型,常引起腹泻、出血性肠炎,并易继发溶血性尿毒综合征和血栓性血小板减少性紫癜等严重并发症。据统计,每年全球约有200万人因大肠杆菌感染而死亡,约1.5亿人罹患膀胱炎、腹泻、败血症和脑膜炎等疾病[2]。在口岸卫生监督中,大肠杆菌是评价食品卫生质量的重要指标之一。因此,准确高效地检测大肠杆菌O157:H7具有重要意义。
我国现行检测标准GB 4789.36—2016《食品安全国家标准 食品微生物学检验 大肠埃希氏菌O157:H7/NM检验》主要通过增菌、分离、生化实验及血清学鉴定等步骤对食品中的大肠杆菌进行检测[3]。该方法检测准确性高,但操作烦琐且耗时较长。分子生物学方法可以提高检测效率,如聚合酶链式反应(Polymerase Chain Reaction,PCR)、环介导等温扩增(Loop-Mediated Isothermal Amplification,LAMP)、滚环扩增(Rolling Circle Amplification,RCA)杂交链式反应(Hybridization Chain Reaction,HCR)等分子检测技术已经广泛应用于大肠杆菌检测中[4]。
HCR是一种非常重要的体外核酸信号放大技术,其原理是通过设计两条互补的发夹状探针,当目标核酸序列存在时,这些探针会相继打开并形成一个长链聚合物,从而放大信号。由于HCR无酶参与,避免了常规酶促扩增方法中可能出现的非特异性扩增问题。此外,HCR反应条件温和(通常在室温下进行),背景干扰低,具有较好的特异性和稳定性,已成功应用于多种病原体的检测。例如,在金黄色葡萄球菌的检测中,通过使用特异性的发夹状探针,HCR能有效地识别并放大目标DNA序列,从而实现高灵敏度的检测[5]。而HCR在鼠伤寒沙门菌的检测中也展示了优于传统PCR方法的灵敏度[6]。钟心怡等[7]基于HCR,结合荧光检测方法,为志贺菌核酸的快速检测提供了一种新的途径。对于大肠杆菌检测,Dan Song等[8]开发了一种名为CHANCE的新方法,结合HCR和CRISPR/Cas12a两个阶段,利用靶标激活Cas12a的反式切割活性,用于快速超灵敏检测大肠杆菌O157:H7,并成功应用于实际环境水样。Miaolin Duan等[9]设计了基于接触淬灭和荧光共振能量转移的杂交链式反应方法,可通过淬灭FAM和从供体到受体破坏FRET而降低背景的CQ-FRET发夹,灵敏地检测大肠杆菌;Xiu Liu等[10]开发了基于ISD-HCR和LFS结合的方法对大肠杆菌O157:H7的16S rRNA进行可视化即时检测,是一种简单、灵敏、低成本的方法,无须精确控制温度,利用AuNPs与特定抗原相互作用以及侧向流动试纸条可以实现快速实时检测。
尽管大肠杆菌检测技术取得了一定发展,但现有方法在简便性、灵敏度、特异性和成本等方面仍存在不足,而且分子生物学方法在复杂样本背景中容易产生假阳性[11]。因此,本研究采用双靶点触发的杂交链式反应进行大肠杆菌核酸检测,通过荧光信号的强度来定量分析样品中的大肠杆菌,以提高检测的灵敏度和准确性。
1 材料与方法
1.1 仪器与试剂
天美FL970 Plus荧光分光光度计;HB120-S LED 数显加热金属浴。
氯化钠(NaCl)、过硫酸铵购自上海麦克林生化有限公司;10×PBS(磷酸盐缓冲溶液)、1×TBE(Tris-硼酸电泳缓冲液)、1×TE(Tris-EDTA缓冲溶液)、Tris-HCl缓冲溶液、HEPES(4-羟乙基哌嗪乙磺酸)缓冲溶液、30%丙烯酰胺、Gelgreen染液、N,N,N,N-四甲基乙二胺购自生工生物工程(上海)股份有限公司;20 bpDNA分子量标准购自宝日医生物技术(北京)有限公司。
以来源于National Center for Biotechnology Infor- mation(NCBI)数据库的大肠杆菌O157:H7 str. Sakai全基因组的两段特异性核酸序列(Genbank No: NC_002695.2)为靶标DNA,分别命名为靶标1(T1)和靶标2(T2),并设计发卡结构探针,分别命名为发卡探针1(H1)、发卡探针2(H2)发卡探针3(H3)和发卡探针4(H4)。幽门螺杆菌、金黄色葡萄球菌、志贺氏菌、沙门氏菌的靶标序列用于验证方法的选择性。所有靶标序列及探针均由生工生物工程(上海)股份有限公司人工合成,序列信息见表1。
1.2 实验方法
1.2.1 凝胶电泳表征
在丙烯酰胺凝胶中按照表2顺序加样,电压120 V条件下电泳1 h。电泳结束后,避光条件下GelGreen染液染色15 min并成像。
1.2.2 荧光检测条件优化
缓冲体系优化:配制含0.15 mol/L 的Na+的0.02M HEPES、1×PBS、1×TE、10mM Tris-HCl、1×TBE缓冲溶液,分别稀释H1、H2和大肠杆菌靶标DNA至10 μmol/L进行退火,孵育并测定荧光信号。
钠离子(Na+)浓度优化:配制含0.05 mol/L、0.1 mol/L、0.15 mol/L、0.2 mol/L、0.3 mol/LNa+的0.02 mol/L HEPES,并用其分别稀释H1、H2和大肠杆菌靶标DNA至10 μmol/L进行退火,孵育并测定荧光信号。
反应时间优化:用含有0.15 mol/L Na+的0.02 mol/L HEPES缓冲液稀释探针H1和H2至10 μmol/L,稀释靶标至10 nmol/L,进行退火。取靶标20 μL(10 nmol/L)与探针H1、H2各5 μL(10 μmol/L)混匀,立即测定荧光信号,重复扫描直至荧光信号稳定,记录反应时间。
荧光光谱仪参数:激发狭缝5 nm,发射狭缝5 nm,电压500 V,激发光波长495 nm,测定范围510~600 nm。
1.2.3 选择性和稳定性验证
准备大肠杆菌、幽门螺旋杆菌、志贺氏菌、金黄色葡萄球菌、沙门氏菌的DNA各200 μL,靶标DNA终浓度为1 nmol/L,H1、H2探针终浓度为250 nmol/L,待孵育后进行荧光检测。并对大肠杆菌DNA的平行样品进行测定,计算相对标准偏差评价方法的稳定性。 ,其中X代表荧光信号值;n代表样品数。
,其中X代表荧光信号值;n代表样品数。
1.2.4 工作曲线测定
取100 μmol/L的大肠杆菌靶标DNA、H1、H2,用0.02 mol/L HEPES溶液(含0.15 mol/L Na+)稀释至10 μmol/L退火;配制0.001~20 nmol/L大肠杆菌靶标DNA,H1、H2浓度为250 nmol/L,待孵育结束后进行荧光检测。
2 实验结果
2.1 杂交链式反应的凝胶电泳表征
用DNA凝胶电泳验证大肠杆菌靶标序列1与H1的结合,靶标序列2与H3的结合;靶标序列1在H1和H2的作用下发生级联反应,靶标序列2在H3和H4的作用下发生级联反应;分析方法具有可行性,电泳结果如图1所示。
2.2 荧光检测条件优化
缓冲体系、盐浓度、反应时间影响碱基互补配对,进一步影响发卡结构的形成,是核酸反应过程中的关键因素,故对荧光检测中的盐浓度、缓冲体系、反应时间作优化[12]。实验设计了5种缓冲体系(0.02M HEPES、1×PBS、1×TE、10mM Tris-HCl、1×TBE),分别进行核酸退火及杂交链式反应,检测荧光信号。如图2所示,荧光信号在0.02 mol/L的HEPES缓冲体系下呈现出最高荧光信号值,因而后续测试均选择在HEPES缓冲中进行。随后,我们配制了含不同浓度Na+的0.02 mol/L HEPES缓冲液,获得测试结果如图3所示,荧光信号在0.15 mol/L Na+处达到峰值,并以此浓度作为后续实验的最佳盐浓度。在加入靶标之后立刻扫描H1、H2与靶标T1反应过程的荧光信号,记录荧光信号随时间的变化,如图4所示,反应在15 min后荧光信号便处于稳定状态,说明本杂交链式反应在15 min时已充分完成。
2.3 检测方法的性能分析
在最佳测试条件下,本方法可对靶标序列产生响应。进一步对方法的稳定性、选择性和检测限等参数进行测试,结果如图5所示。在图5中,10份平行样品检测均值为19544,相对标准偏差(RSD)为0.11%。对于浓度相同的幽门螺旋杆菌、志贺氏菌、金黄色葡萄球菌、沙门氏菌的DNA,在相同条件下可见仅有大肠杆菌的目标序列有较强的荧光信号,其他微生物序列均未产生显著的信号(图6)。
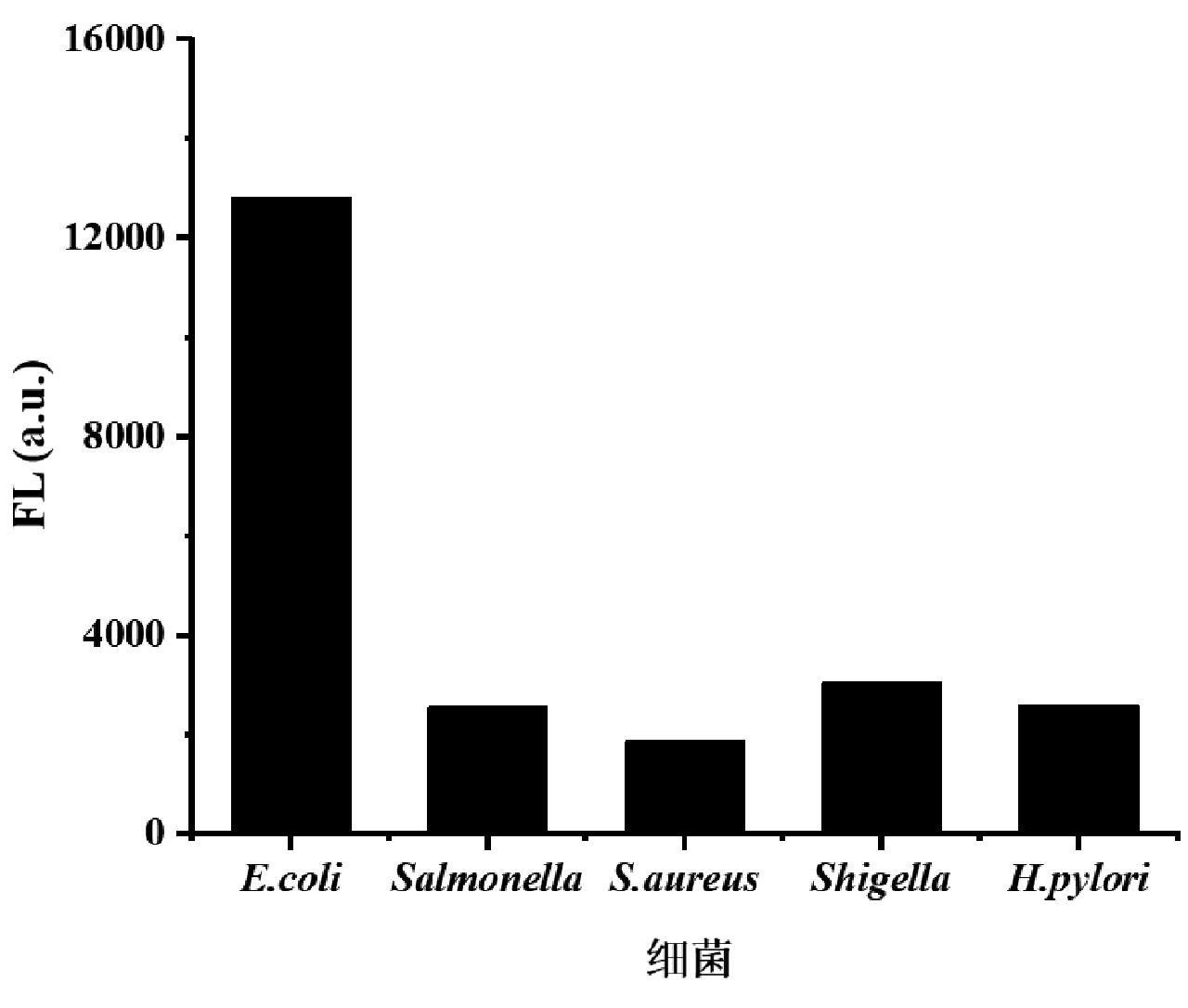
图6 荧光检测不同微生物靶标核酸的特异性表征结果
Fig.6 Specificity characterization results of different microbial target nucleic acids by fluorescence detection
为了提高本方法的应用性,我们进一步建立对目标序列的工作曲线。对0.001~20 nmol/L范围内的一系列不同大肠菌DNA浓度的标准溶液进行测定(平行样品检测取均值,n = 4),如图7(a)和(c)所示,荧光信号随着浓度的提高而提高,呈现正相关。为了提高拟合线性,我们对浓度区间以1 nmol/L为界限划分成了低浓度区间和高浓度区间。如图7b所示,在浓度1~20 nmol/L范围内,拟合线性曲线为y = 538.38x + 17389.6,R2 = 0.9979;而1 nmol/L以下的浓度区间内,拟合线性曲线y = 12815x + 4266.4,R2 = 0.9706,如图7d所示。 据此判定方法的检出限即为0.001 nmol/L。
2.4 样品检测
为验证双靶标检测的效果,设置4份人工加标样品,样品1为大肠杆菌核酸,样品2、样品3分别加入大肠杆菌靶标2和大肠杆菌靶标1,样品4加入幽门螺旋杆菌核酸,使用本方法进行测试,结果如图8所示。阳性样品(样品1)对2组探针均有响应,呈现高荧光信号值,为双阳性;加入大肠杆菌靶标1和靶标2的样品(样品3和样品2)仅产生单阳性信号,而阴性质控品(样品4)对2组探针信号响应程度均未出现信号。

图8 不同样品的荧光信号值
Fig.8 Fluorescence signal values of different samples
随后,使用本方法对一系列加入了不同浓度大肠杆菌的环境水样进行了检测,以荧光强度高于4000为阳性,并使用标准PCR方法进行对比以检测方法的有效性。从表3结果可见,使用本方法的检测结果与PCR方法有较好的一致性。
表3 环境水样大肠杆菌的HCR检测和PCR检测结果
Table 3 Detection results of E. coli in environmental water sample via HCR and PCR
检测方法 | 加标浓度 (cfu/mL) | |||||||
10.0 | 5.0 | 1.0 | 0.5 | 0.1 | 0.05 | 0.02 | 0.01 | |
HCR | + | + | + | + | + | + | + | + |
PCR | + | + | + | + | + | + | + | - |
注: “ +”代表阳性, “-”代表阴性; HCR检测中, 以荧光读数高于4000为阳性, 低于4000为阴性; PCR检测中, Ct值低于35为阳性, 高于35为阴性.
3 讨论
3.1 实验原理
本研究针对大肠杆菌的特异性靶标DNA序列T1,设计出能够与其碱基互补配对的发卡结构核酸H1,以及能够与H1发生反应并修饰有荧光基团6-FAM和猝灭基团BHQ-1的发卡结构核酸H2。当目标核酸序列不存在时,H1和H2均保持完整的发卡结构形式,两端的修饰基团由于空间距离较近而发生共振能量转移,导致荧光猝灭;当大肠杆菌靶标存在时,靶标T1捕获H1并打开其发卡结构,打开的H1可以进一步打开H2,使H2的荧光基团远离猝灭基团,从而恢复荧光信号。与此同时,打开的H2因其环部序列恢复了碱基互补活性,能够再次打开另一个发卡结构的H1。通过这种方式,两个发卡结构之间发生级联反应,实现了灵敏的检测[13-14]。靶标1和靶标2独立触发级联反应,提供双重验证,避免了单靶点检测中由于非特异性开环或其他因素引发的级联反应,提高了检测的准确性并降低了假阳性率。
DNA凝胶电泳验证结果显示,泳道4形成了迁移率低于靶标T1和发卡探针H1的条带,且靶标条带消失,证明H1可成功捕获靶标T1与其发生碱基互补配对且反应充分。泳道5和6的条带显示,H2无法单独与H1或T1发生反应。泳道7由于探针成功捕获靶标发生级联反应,发卡结构依次打开,碱基数逐级增加,出现杂交链式反应电泳的典型特征(逐级拖尾)。而另一个靶点触发的反应也显示了完全一致的级联过程。
3.2 方法检测性能
本研究在优化条件的基础上,通过平行检测验证评估了该检测方法的性能,结果显示该检测方法具有良好的稳定性,一方面是因为荧光检测具有稳定性,另一方面级联反应的动力学重现性较好,在一定时间内能呈现相似的反应进程,而这一特性优于PCR检测方法。
HCR反应是典型的靶标触发式反应,其反应动力来自于靶标与发卡完成碱基互补配对后释放的化学能,是一个熵增过程。因此,这一反应高度依赖于靶标和发卡的结合力,对于其他种类的微生物核酸,无法完成这一碱基互补配对过程,因而不能触发级联反应,从而使本方法表现出高度的特异性。
该检测体系响应速度快,从15 min起荧光信号即稳定且不随时间变化,符合无酶参与的杂交链式反应中分子反应迅速的特点,有望应用于快速检测中。大肠杆菌靶标DNA浓度与荧光信号强度呈现显著的正相关,在低浓度区间内,靶标分子可以更大程度地触发发卡探针反应,在发卡浓度保持一致的情况下,随着靶标浓度的增加,单个靶标分子触发反应的级联程度会出现下降。因此,为了增加工作曲线的实用性,本研究以1 nmol/L为界限,分成两段进行了线性拟合,提高了荧光信号与靶标浓度的线性相关性,可以看到高浓度区间的工作曲线斜率低于低浓度区间。本方法的检测限可达0.001 nmol/L,这一性能与文献报道的HCR反应检测限相当,可满足实际样本检测需求[7-11]。
3.3 双靶点检测降低假阳性
在核酸检测中,非特异性结合引发的假阳性信号是一个很普遍的问题。例如,在PCR检测中,引物与非模板链产生的缠绕、吸附等非碱基互补作用也可能触发扩增反应进行[15]。对于HCR反应,可能产生假阳性信号的来源主要是发卡自身的非特异性打开,这会导致在没有靶点序列的时候依然有一定程度的反应被触发[16]。而双靶点检测相当于对单靶点的检测结果再进行一次独立的确证实验,仅有在双重阳性时才判定为检出,这就大大降低了误判的可能性。
4 结论
本研究建立了一种基于双靶点杂交链式反应的大肠杆菌核酸检测新方法。通过设计针对大肠杆菌O157:H7特异性核酸序列的发卡探针,并优化检测条件,实现了对靶标核酸的灵敏性、稳定性和特异性的检测。该方法不仅表现出优异的稳定性和选择性,还在较宽的靶标浓度范围内保持良好的线性响应,检测限达0.001 nmol/L。在实际样本检测中,该方法能够精准区分出真实阳性样本。
该检测方法具有高灵敏度和特异性,能够有效地筛查和检测大肠杆菌,反应时间短,双靶点的设计也提高了检测的准确性,降低了误判风险。这不仅有助于海关应对口岸突发公共卫生事件,还能提高进出口货物的检验效率。总之,该方法为大肠杆菌的精准检测与控制提供了有力的技术支持,在口岸卫生检疫工作中具有广阔的应用前景。
参考文献
[1]李文, 王彩喆, 牟林云, 等. 大肠杆菌GalP-GlK途径功能分析[J]. 天津科技大学学报, 2024: 1-8.
[2]白小莲, 爱军. 生物传感器在食源性致病菌大肠杆菌O157:H7检测中的应用进展[J]. 生物技术进展, 2021, 11, 269-278.
[3] GB 4789.36—2016 食品安全国家标准 食品微生物学检验 大肠埃希氏菌O157:H7/NM检验[S].北京: 中国标准出版社, 2016.
[4] L Zhuang, J Gong, Y Zhao, et al. Progress in methods for the detection of viable Escherichia coli[J]. Analyst, 2024, 149, 1022-1049.
[5] X. Wang, X.Wang, S. Wei. Target-Triggered hybridization chain reaction self-assembly of G-quadruplex DNAzyme for colorimetric detection of Staphylococcus aureus in milk[J]. Microchemical Journal, 2023, 193, 109180.
[6] S. Dou, S. Zhou, H. Wang, et al. UIO66 low background signal and fluorescence synergism strategy for highly sensitive detection of Salmonella typhimurium [J]. Talanta, 2024, 274: 126013.
[7]钟心怡, 庄妙慧, 杨琪玲, 等. 基于杂交链式反应的志贺菌核酸快速检测研究[J]. 中国国境卫生检疫杂志, 2023, 46(1): 1-5.
[8] D. Song, X. Han, W. Xu, et al. Target nucleic acid amplification-free detection of Escherichia coli O157:H7 by CRISPR/Cas12a and hybridization chain reaction based on an evanescent wave fluorescence biosensor [J]. Sensors and Actuators: B. Chemical, 2023, 376: 133005.
[9] M. Duan, X. Xiao, Y. Huang, et al. Immuno-HCR based on contact quenching and fluorescence resonance energy transfer for sensitive and low background detection of Escherichia coli O157:H7[J]. Food Chemistry, 2021, 334: 127568-127568.
[10] X. Liu, S. Bu, H. Wei, et al. Visual assay of Escherichia coli O157:H7 based on an isothermal strand displacement and hybrid chain reaction amplification strategy[J]. Analytical Methods, 2021, 13(30): 3379-3385.
[11]杨婵, 王瑞嘉, 何婧琳, 等. 杂交链式反应在生物传感探针设计方面的应用[J]. 化学传感器, 2017, 37(3): 8-16.
[12] X. Zhong, Y. Deng, Q. Yang, et al. An extracellular electron transfer enhanced ECL aptasensor for Escherichia coli analysis[J]. Analyst, 2023, 148, 4414-4420.
[13] R. Guo, S. Hu, Z. Wang. A portable electrochemiluminescence bipolar electrode array for the visualized sensing of Cas9 activity[J]. Analyst, 2020, 145, 3569-3574.
[14] Y. Li, H. Liu, H. Huang, et al. A sensitive electrochemical strategy via multiple amplification reactions for the detection of E. coli O157:H7[J]. Biosensors and Bioelectronics, 2020, 147, 111752.
[15] G. Mak, D. Hung. Non-specific signals in real-time RT-PCR for detecting respiratory viruses[J]. Diagnostic Microbiology and Infectious Disease, 2024, 108, 116136.
[16] Y. Bian, M. Li, Z. Wu, et al. A natural biomaterial promotes hybridization chain reaction for ultra-sensitive detection of miRNA-155[J]. Talanta, 2024, 266, 125117.
表1 人工核酸序列信息表
Table 1 List of artificial DNA sequences used in this study
细菌/探针 | 序列 (5'~3' ) |
大肠杆菌靶标1 (T1) | CGGTAAAGCGACTGTTAGTGCGACA |
H1 | GGTAAAGCG ACTTGTAGCCTGAACTGA TGTCGCACTAACA GTCGCTTTACCG |
H2 | TCAGTTCAGGCTACAAGT TGAA CGGTAAAGCGACTGTTAG AGCCTGAACTG |
大肠杆菌靶标2 (T2) | GTGGTGATGGTACATATTCATGGTA |
H3 | TGGTGATGG TATCCAGATCCGTACAGT TACCATGAATATG TACCATCACCAC |
H4 | ACTGTACGGATCTGGATA TTA GTGGTGATGGTACATATT AGATCCGTACAG |
金黄色葡萄球菌 | CGGACGAGAAGCTTGCTTCTCT |
幽门螺杆菌 | ATTCCTCCTACCCGCGGCAAGACGGAAA |
志贺氏菌 | CCGCAGCACGTTCTTGCTTATGCAT |
沙门氏菌 | GGCGGCGGGCGTCGCGCACG |
表2 DNA凝胶电泳加样表
Table 2 Sample information in PAGE assay
泳道 (a) | 1 | 2 | 3 | 4 | 5 | 6 | 7 |
样品 | T1 | H1 | H2 | T1+H1 | T1+H2 | H1+H2 | T1+H1+H2 |
泳道 (b) | 1 | 2 | 3 | 4 | 5 | 6 | 7 |
样品 | T2 | H3 | H4 | T2+H3 | T2+H4 | H3+H4 | T2+H3+H4 |

a: T1引发的HCR; b: T2引发的HCR
图1 杂交链式反应可行性验证DNA凝胶电泳结果图
Fig.1 Feasibility verification of HCR
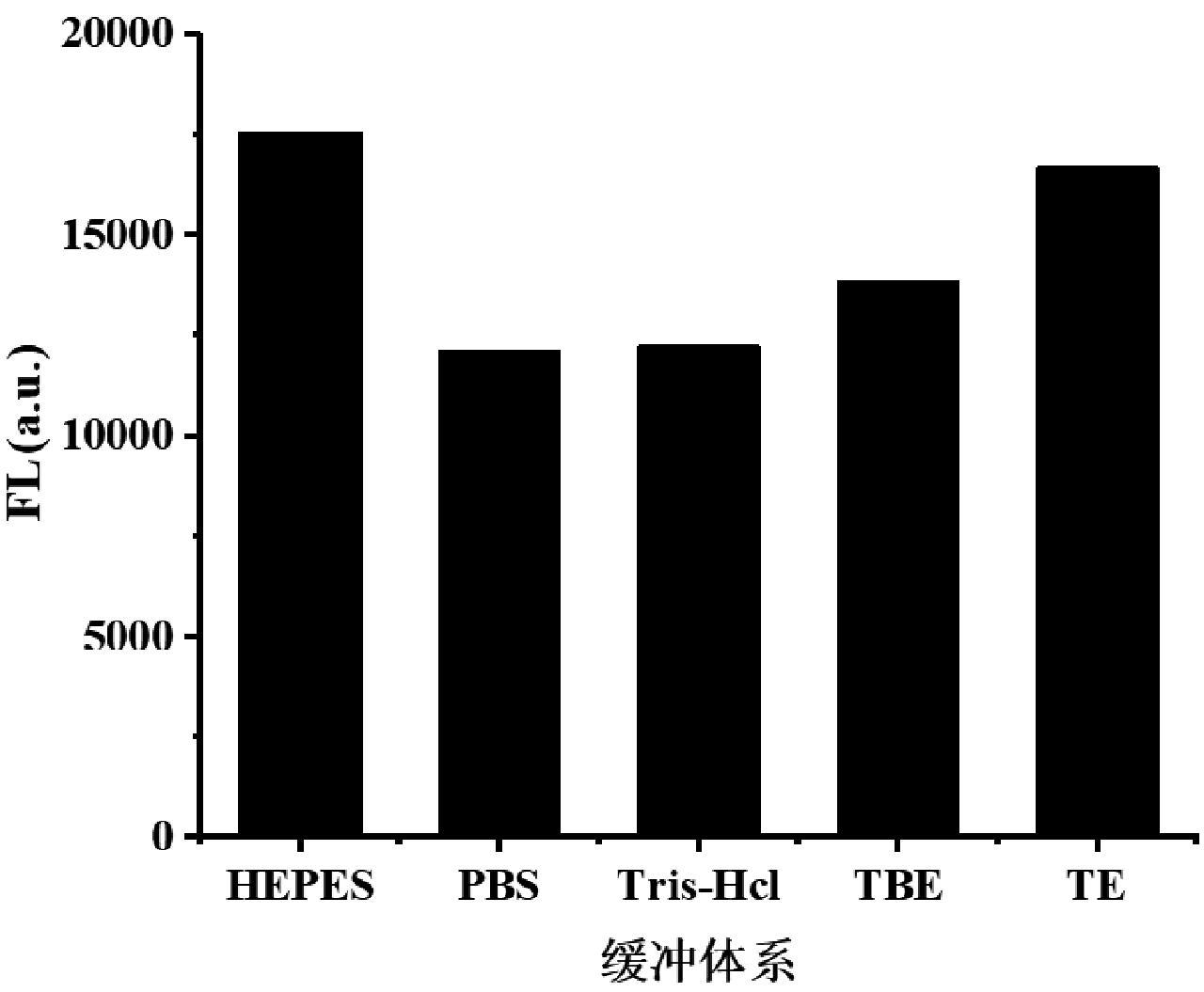
图2 不同缓冲体系下的荧光信号
Fig.2 Fluorescence signals under different buffer systems

图3 荧光信号随Na+浓度的变化
Fig.3 Fluorescence signal changes with Na+ concentration
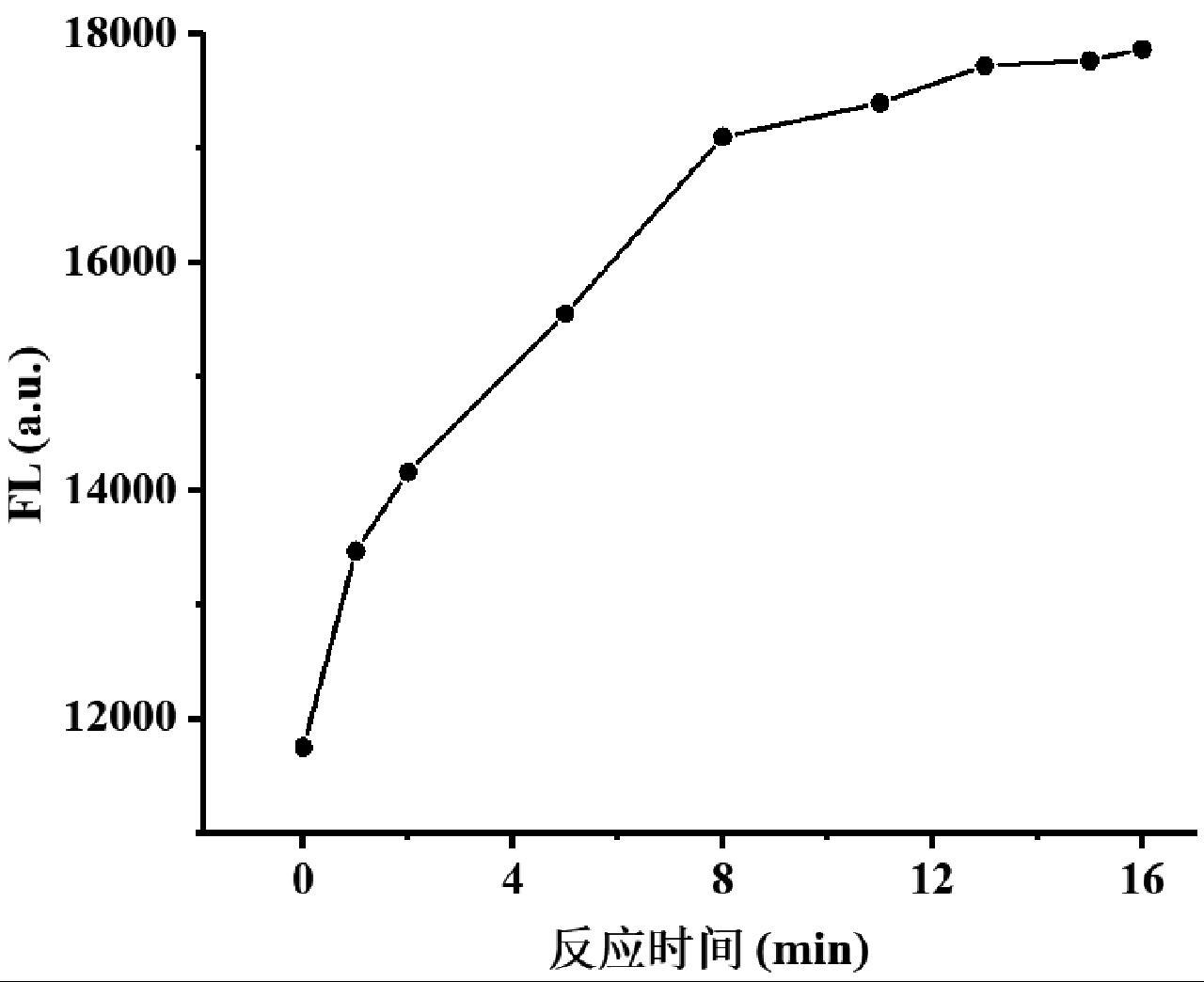
图4 荧光信号随反应时间的变化
Fig.4 Fluorescence signal changes with reaction time
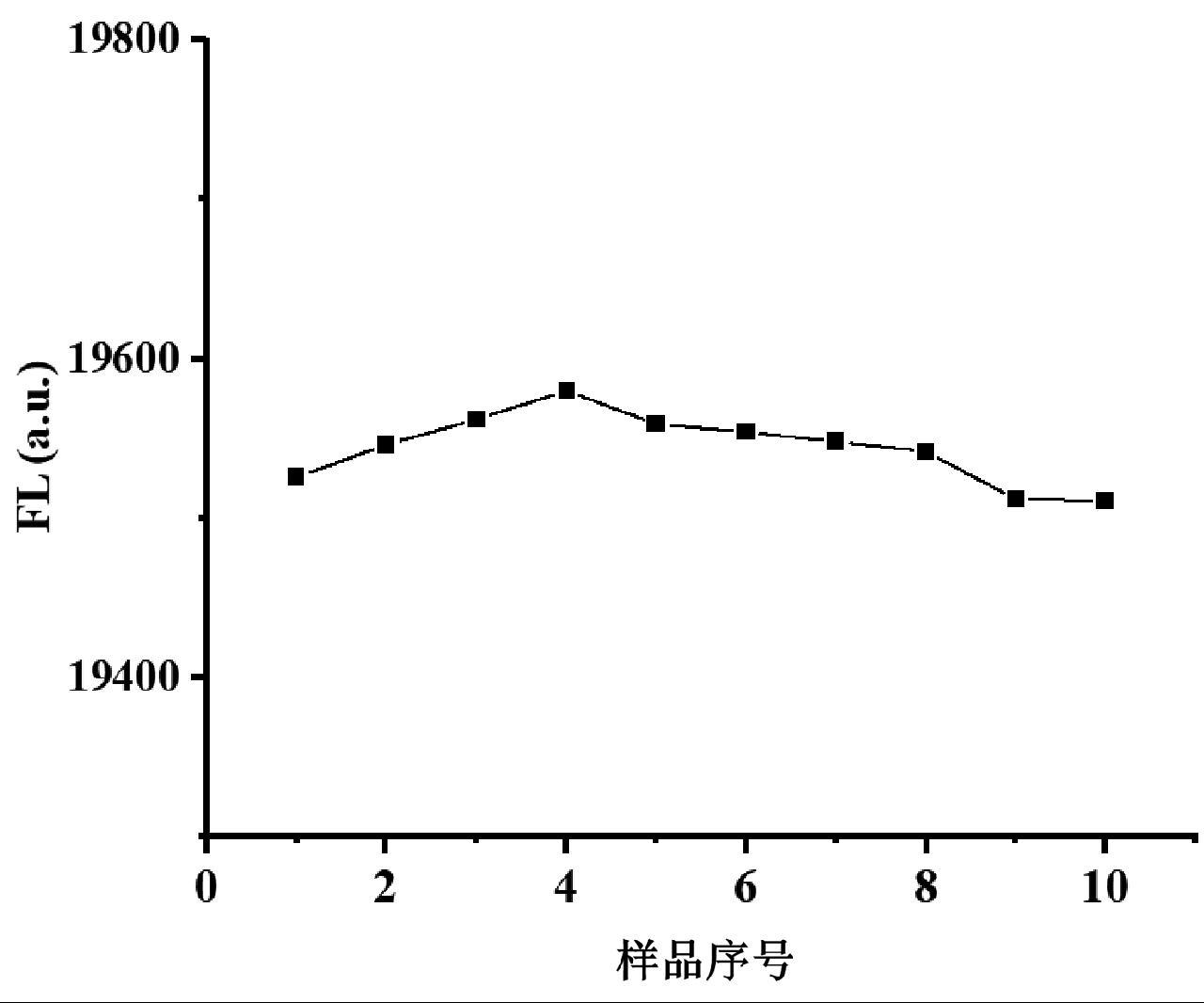
图5 荧光检测平行样品的稳定性表征结果
Fig.5 Stability characterization results of parallel samples by fluorescence detection

图7 不同浓度靶标DNA所测荧光谱线(a)、(c)及其对应检测工作曲线(b)、(d)
Fig.7 Fluorescence spectra of HCR with different concentrations of target DNA (a, c) and the corresponding working curves (b, d)







