CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
多重PCR-MALDI-TOF MS技术用于鉴别奥密克戎EG.5和BA.2.86变异株
作者:张晓龙 魏海燕 郭悠然 袁芳 祁科元 郭惠民 宋悦谦
张晓龙 魏海燕 郭悠然 袁芳 祁科元 郭惠民 宋悦谦
摘 要 鉴于奥密克戎EG.5和BA.2.86变异株的强传播能力,本研究将基质辅助激光解吸电离飞行时间质谱与多重PCR技术联用,针对其刺突(S)蛋白上的关键突变位点进行扩增和延伸引物设计,同时引入用于鉴别的D614G位点和用于SARS-CoV-2鉴定的ORF1ab及N基因检测位点,可一次性检测10个特征位点。结果表明,该方法可在同一反应体系中同时检测2种重点关注变异株EG.5和BA.2.86,检测结果准确,灵敏度低至20 copies/反应。该方法适合海关及其他疫情监测部门对重点关注变异株进行大量样本的动态筛查工作,是荧光定量PCR和高通量测序方法很好的技术补充,可为病毒溯源和疫情监测提供技术支撑。
关键词 奥密克戎变异株;多重PCR;基质辅助激光解吸电离飞行时间质谱;分型筛查
The Application of Multiplex PCR-MALDI-TOF MS Technique for Identification of Omicron EG.5 and BA.2.86 Variants
ZHANG Xiao-Long1 WEI Hai-Yan1 GUO You-Ran2 YUAN Fang1
QI Ke-Yuan2 GUO Hui-Min2 SONG Yue-Qian1*
Abstract In light of the highly transmissible nature of the Omicron EG.5 and BA.2.86 variants, this study integrates Matrix-Assisted Laser Desorption Ionization Time-of-Flight Mass Spectrometry (MALDI-TOF MS) with multiplex PCR technology. Primers were designed to amplify and extend key mutation sites on the spike (S) protein of these variants. Additionally, the D614G mutation site was included for identification, along with the ORF1ab and N gene detection sites for the identification of SARS-CoV-2, enabling the simultaneous detection of 10 characteristic sites. The results demonstrate that this method can detect both the EG.5 and BA.2.86 variants in the same reaction system with high accuracy and a sensitivity as low as 20 copies per reaction. This method is well-suited for dynamic screening of a large number of samples by customs and other epidemic monitoring departments, focusing on variants of concern. It serves as a valuable technical complement to quantitative fluorescent PCR and high-throughput sequencing methods, providing technical support for virus tracing and epidemic surveillance.
Keywords Omicron variants; multiplex PCR; Matrix-Assisted Laser Desorption Ionization Time-of-Flight Mass Spectrometry (MALDI-TOF MS); typing screening
基金项目:海关总署科研项目(2022HK149 )
第一作者:张晓龙(1972—),男,汉族,重庆江津人,博士,研究员,主要从事病原生物学领域相关工作,E-mail: zhzhsh2001@126.com
通信作者:宋悦谦(1972—),男,汉族,北京人,硕士,主要从事卫生检疫领域相关工作,E-mail: songyueq@263.net
1. 中国海关科学技术研究中心 北京 100026
2. 浙江迪谱诊断技术有限公司 杭州 311100
1. Science and Technology Research Center of China Customs, Beijing 100026
2. Zhejiang Digena Diagnostics Technology Co., Ltd., Hangzhou 311100
中国口岸科学技术
SARS-CoV-2为单股正链RNA病毒,其高突变率导致了奥密克戎(Omicron)的分支极多,如BA.1、BA.2、BA.5系列和 BQ.1、重组体XXB等。2023年7月发现的BA.2.86变异株引起了研究人员的广泛关注[2]。与参考株(ACCESSION No. NC_045512.2)相比,BA.2.86在S蛋白上有60多处突变,与其他Omicron毒株(BA.2、BA.5、XBB.1.5和EG.5.1等)相比,其特有的突变位点也多达26处[3]。2023年底,Omicron变异株EG.5仍占比较高[1]。EG.5是XBB.1.9.2的后代谱系,具有与XBB.1.5相同的刺突氨基酸谱,但携带额外的F456L突变。研究表明,EG.5及其亚分支更高的传播能力及更强的免疫逃逸能力与S蛋白上的F456L、Q52H等特有的突变位点有关[1]。
鉴于EG.5和BA.2.86变异株的强免疫逃逸能力和传播能力[4],开展Omicron关注变异株分型检测,可深入了解病毒的进化历程和基因变异规律,为病毒溯源和疫情监测提供科学依据。了解EG.5和BA.2.86变异株在不同地区和人群中的分布和传播情况,有助于评估其在全球传播的趋势,为公共卫生决策提供数据支持。
目前国内外在SARS-CoV-2分型方面的检测方法主要依赖于测序手段[5-6]。虽然测序技术具有高敏感性和特异性,但其成本较高,建库流程复杂,耗时较长且需要专业的分析软件或生信分析人员对数据进行处理,不利于对已知的重点关注变异株进行动态批量地分型筛查。基于定量PCR的SARS-CoV-2变异分析是一种快速、经济的方法。然而,由于受到仪器平台荧光检测通道数量的限制,这种方法在一次反应中只能检测到3~4个突变位点的靶点,因此,需要通过多突变位点组合以满足鉴定变异株系的需求[7-8]。
多重PCR-基质辅助激光解吸电离飞行时间质谱,集合了多重PCR的高灵敏度、单碱基延伸的特异性及基质辅助激光解吸电离飞行时间质谱(Matrix-Assisted Laser Desorption Ionization Time-of-Flight Mass Spectrometry,MALDI-TOF MS)的中通量等优点,已在SNP分型、耐药突变检测和病原微生物鉴定等方向得到越来越广泛的应用[9-12]。近期的研究进展展现了核酸质谱法在精准识别病毒变异株方面的潜力。该方法通过设计特定的位点靶标,能够在两步流程中高效地鉴别出多种变异类型。首先,在一个测试管中,通过对25个关键位点的分析,能够迅速筛检出Alpha、Beta、Delta以及Omicron共4种具有全球公共卫生意义的变体。一旦样本被确认为Omicron变体,研究者则会进一步采用第二管测试,包含28个额外的位点,以实现对Omicron亚型BA.1.1、BA.1.1.529和BA.2的精确区分。然而,这一检测策略目前尚未涵盖所有新兴的值得关注的变异株。例如,最近出现的EG.5和BA.2.86等变异株,由于其独特的遗传特征,当前的检测方案可能无法对其进行准确分类[13]。
本研究将MALDI-TOF MS技术与多重PCR技术联用,针对EG.5和BA.2.86的刺突(S)蛋白上的关键突变位点进行扩增和延伸引物设计,并增加用于SARS-CoV-2通用型鉴定的ORF1ab、N基因和D614G检测位点。通过一次实验,同时对10个特征位点进行检测,根据检测结果对两种关注变异株进行分型筛查。
1 材料与方法
1.1 材料
1.1.1 材料与试剂
病毒 DNA/RNA提取试剂盒 4.0(西安天隆科技有限公司);虾碱性磷酸酶(Shrimp Alkaline Phosphatase,SAP)、延伸反应试剂和核酸质谱芯片(浙江迪谱诊断有限公司)。
1.1.2 仪器与设备
DP-TOF核酸质谱仪(浙江迪谱诊断有限公司);ProFlex PCR仪(赛默飞世尔科技公司);NP968-C全自动核酸提取仪(西安天隆科技有限公司)。
1.1.3 样本来源
新型冠状病毒 EG.5株、BA.2.86株、Alpha株、Delta株、XBB.1.16等其他Omicron变异株、甲型流感病毒、乙型流感病毒、腺病毒、人冠状病毒OC43及229E亚型为实验室呼吸道病原检测项目收集到的临床样本;样本亚型信息均经过纳米孔测序方法验证。
1.1.4 引物设计
从国家生物信息中心(https://ngdc.cncb.ac.cn/ncov/)查询EG.5和BA.2.86相关的S蛋白突变位点信息以及用于SARS-CoV-2通用鉴别的ORF1ab和N基因的保守区信息,使用Typer-Assay Designer软件和Fast PCR在线工具对多重扩增引物体系和多重延伸引物体系进行设计,共包含7组用于分型鉴别的S蛋白突变位点的引物组和3组用于鉴别SARS-CoV-2阴阳性的引物组。由于飞行时间质谱系统检测范围为4000~10000 Da,在正反向引物的5'端引入人工序列ACGTTGGATG,使所有扩增引物的分子量>10000 Da,与延伸产物的分子量进行区分。设计完成后使用Primer-BLAST、MFE primer 3.1等在线软件对所设计的引物体系分别进行特异性、引物潜在二级结构的评估。引物序列、质谱检测时的Mass值等信息见表1。 所有引物均为PAGE纯化,由湖南艾科瑞生物工程有限公司合成。
1.2 方法
1.2.1 病毒RNA的提取
按照病毒 DNA/RNA提取试剂盒 4.0说明书要求对病毒RNA进行提取纯化,并于-20℃保存备用。
1.2.2 反应体系的建立
检测体系主要包含靶片段多重RT-PCR扩增、 SAP消化、单碱基延伸反应、树脂纯化和质谱检测。
多重RT-PCR扩增:将2.5 μL RT-PCR缓冲液、0.3 μL RT-PCR 酶,0.2 μL 多重PCR引物混合液(每种引物浓度2.5 μmoL/L )和2 μL提取的模板混合,反应程序为50℃ 10 min;95℃ 5 min;95℃ 15 s, 60℃ 30 s,进行45个循环;72℃ 5 min。
SAP 消化反应:将扩增产物加入 2 μL SAP 酶混合液,反应程序为 37℃ 40 min, 85℃ 5 min。
单碱基延伸反应:将消化产物加入0.94 μL延伸引物混合液,0.2 μL延伸酶混合液和0.86 μL延伸反应缓冲液,反应程序为94℃ 30 s;94℃ 5 s,(52℃ 5 s,80℃ 5 s,5个循环),40个循环;72℃延伸5 min。
将上述单碱基延伸产物转移至芯片待检点位对应的384微孔板中。
产物纯化及MALDI-TOF MS检测:将384微孔板和芯片放入DP-TOF 飞行时间质谱仪,编辑待检板和孔位信息,启动检测程序,仪器将自动对各微孔中的产物进行脱盐纯化,点样,产物基质共结晶,质谱飞行,结束后使用Typer 4.0软件对各延伸引物和延伸产物的分子量进行分析。
结果判定:查看各反应孔的质谱峰,按表2所示的规则判断不同质量峰对应的分型结果。
1.2.3 特异性测试
为确定反应体系特异性是否良好,将人冠状病毒-229E、人冠状病毒-OC43、甲型流感病毒、乙型流感病毒、腺病毒和新型冠状病毒(Alpha株、Delta株)的核酸作为扩增模板,使用 EG.5和BA.2.86作为阳性对照,无RNase去离子水为阴性对照,采用1.2.2确定好的反应体系和扩增程序,对方法进行特异性实验验证。
1.2.4 灵敏度测试
为确定多重反应体系的灵敏度,将已知浓度的阳性分离株EG.5和BA.2.86的核酸进行N基因拷贝数的数字PCR定量,针对10~105 copies/μL的10倍梯度稀释液,采用1.2.2的反应体系和程序,测定其临界检测限。
1.2.5 混合样本的检测
为确定体系对混合感染样本的分型检测能力,人工模拟制备EG.5和BA.2.86混合感染样本,以EG.5和BA.286单一感染样本为阳性对照,以无RNase去离子水为阴性对照,采用1.2.2的反应体系和程序,观察其杂合峰特征。
1.2.6 真实样本的检测
为确定本方法检测结果的可靠性,对真实临床样本同时进行了新型冠状病毒荧光定量PCR对N基因的检测和病毒全长纳米孔测序。比较荧光定量PCR Ct值与核酸质谱延伸准确率的关系。
2 试验结果
2.1 方法的特异性
对于非SARS-CoV-2的样本,所有样本均未检测到特异的延伸峰,仅有各位点对应的延伸引物峰,对于Alpha株和Delta株的样本,除ORF1ab和N基因的保守位点有延伸峰出现,其余位点延伸峰对应为非EG.5和BA.2.86,根据检测质谱图1、图2结果可判读分别为SARS-CoV-2阴性和SARS-CoV-2其他亚型。
2.2 方法的敏感性
对于10~105 copies/μL的10倍梯度稀释的阳性样本,EG.5和BA.2.86均可稳定检出。如图3、图4所示在10 copies/μL的浓度下,用于鉴别分型的个别位点延伸率未达到100%,此时有延伸峰出现但延伸引物没有完全消耗尽,说明该浓度已到达了此位点的检测下限附近。
2.3 方法的混合检出能力
本方法的V213EG、V445AD和P681HR位点可通过延伸位点的碱基不同进行EG.5和BA.2.86的区分,因此制备模拟混合样本,可评估各位点的延伸情况。图5为当EG.5和BA.2.86样本1∶1混合时质谱检测结果。如图5所示,以V213EG和V445AD为例,此种位点的延伸引物已完全消耗,延伸产物出现杂合峰,可判断为混合感染。
2.4 真实样本检测情况
对收集到的待测样本分别进行核酸质谱、qPCR及纳米孔全长测序的检测。当用于N基因的qPCR检测Ct值<30时,核酸质谱与纳米孔测序均可通过检测结果识别变异类型。在核酸质谱测试过程中,可发现当用于EG.5亚型特异性的鉴别位点F456L位点对应的延伸碱基非A,而Q52H位点对应的延伸碱基非T时,可明确该样本非EG.5亚型。当属于BA.2.86独有的突变类型——16MPLE Call值非G时,可指示该样本非BA.2.86亚型。又可根据D614G、Uni-N、Uni-ORF1ab 3个位点的延伸结果,判定此3个样本为非BA.2.86和EG.5的其他变异株系,如图6、图7、图8所示。使用3代测序手段对样本进行测序验证,证明了其分别为与EG.5亲缘关系较近的XBB.1.16、XBB.1.5和FL.16。
3 讨论
2023年5月5日,世界卫生组织宣布,新冠疫情不再构成“国际关注的突发公共卫生事件”[14],但为了监测新的变异株,国内外研究人员都在寻找介于荧光定量PCR(低通量)和测序(高通量)之间的中通量的突变位点检测手段。本研究尝试了一种多重PCR和MALDI-TOF MS联用的技术手段,弥补了荧光定量PCR检测通量低以及高通量测序流程烦琐、数据分析复杂的缺点,且操作流程简便,搭配DP-TOF核酸质谱平台单次运行最多可以检测384个样本,适用于对大批样本进行特定关注变异株的分型筛查。根据公开信息平台公布的SNP突变位点信息,可在原有体系中增加新的变异位点引物组或设计新的检测组合,快速响应新变异株鉴定的需求。
本方法中每个突变位点共需设计3条引物,即2条用于特异片段扩增的PCR引物和1条用于突变位点检测的延伸引物。在扩增阶段病毒的特异片段首先被富集,经SAP消化掉多余的dNTP原料后,引入延伸引物及ddNTP原料,在延伸酶的催化下进行单碱基延伸反应。3条引物为体系提供了双重特异性保障。在对常见的人呼吸道病原进行检测时,检测结果均为阴性,且对于EG.5和BA.2.86的单一感染样本检测结果准确。
当对阳性样本稀释至10 copies/μL时,部分靶标的延伸率未达到100%,这与其他研究中的结论相似,说明在多重PCR体系中,各靶标的扩增效率不能达到完全一致,导致在单碱基延伸时,延伸引物的消耗量也有所不同[15]。由于本研究对扩增反应体系的优化及引物浓度的调整,因此各靶标的临界检测限不完全一致,但仍在同一量级范围内,在20 copies/反应情况下,各靶标均可检出。
在模拟混合感染情况时,本研究假设了一位患者同时感染2株病毒的情况,但是并未对2株病毒浓度高低的多种情况进行模拟,评估2种延伸产物的比例对结果判读的影响。考虑到本方法适用于对已知关注突变株的初筛,不涉及定量检测,因此如果检测结果为2种突变类型同时存在,则应考虑配合其他检测方法进一步确认是否为新的突变亚型。
在对真实样本的检测方法比较中,本文作者观察到一些Omicron变异株在V213EG、V445AD以及P681HR这三个特定遗传位点上展现出与EG.5亚型一致的延伸产物特征。然而,值得注意的是,对于特异于EG.5亚型的标志性突变(F456L与Q52H)并未在这些样本中出现。这一发现明确排除了样本属于EG.5亚型的可能性。后续的测序数据进一步证实了上述初步判断,即这些样本并不属于本次检测所重点关注的EG.5亚型分类。
本研究基于多重PCR-MALDI-TOF MS法,对重要的突变位点进行扩增引物和延伸引物设计,可在同一反应体系中同时检测2种重点关注变异株EG.5和BA.2.86,检测结果准确,灵敏度低至20 copies/反应,适合海关及其他疫情监测部门对重点关注变异株进行大量样本的动态筛查工作,是荧光定量PCR和高通量测序方法很好的技术补充。
参考文献
[1] Rasmussen M, Møller F T, Gunalan V, et al. First cases of SARS-CoV-2 BA. 2.86 in Denmark, 2023[J]. Eurosurveillance, 2023, 28(36): 2300460-2300465.
[2] Lassaunière R, Polacek C, Utko M, et al. Virus isolation and neutralisation of SARS-CoV-2 variants BA. 2.86 and EG. 5.1[J]. The Lancet Infectious Diseases, 2023, 23(12): 509-510.
[3] Scarpa F, Pascarella S, Ciccozzi A, et al. Genetic and structural analyses reveal the low potential of the SARS‐CoV‐2 EG. 5 variant[J]. Journal of Medical Virology, 2023, 95(9): e29075.
[4] Rahimi F, Darvishi M, Bezmin Abadi A T. ‘The end’–or is it? Emergence of SARS-CoV-2 EG. 5 and BA. 2.86 subvariants[J]. Future Virology, 2023, 18(13): 823-825.
[5] Bull R A, Adikari T N, Ferguson J M, et al. Analytical validity of nanopore sequencing for rapid SARS-CoV-2 genome analysis[J]. Nature Communications, 2020, 11(1): 6272-6280.
[6] Bal A, Destras G, Gaymard A, et al. Two-step strategy for the identification of SARS-CoV-2 variant of concern 202012/01 and other variants with spike deletion H69–V70, France, August to December 2020[J]. Eurosurveillance, 2021, 26(3): 2100008-2100013.
[7] Corbisier P, Petrillo M, Marchini A, et al. A qualitative RT-PCR assay for the specific identification of the SARS-CoV-2 B. 1.1. 529 (Omicron) Variant of Concern[J]. Journal of Clinical Virology, 2022, 152: 105191-105197.
[8] Holland S C, Holland L R A, Smith M F, et al. Digital PCR discriminates between SARS-CoV-2 Omicron variants and immune escape mutations[J]. Microbiology Spectrum, 2023, 11(4): 52-58.
[9] 梁建琴, 吴雪琼, 安慧茹. 核酸基质辅助激光解吸电离飞行时间质谱技术在结核病和非结核分枝杆菌病诊断中的临床应用专家共识[J]. 中国防痨杂志, 2023, 45(6): 543-558.
[10]张晓峰, 宋士琦, 曾若雪, 等. 六种猪呼吸道RNA病毒MALDI-TOF MS同步检测体系的构建[J]. 微生物学报, 2023, 63(9): 3653-3666.
[11] Qian X, Agyekum E A, Zhao L, et al. A comparison of DP-TOF Mass Spectroscopy (MS) and Next Generation Sequencing (NGS) methods for detecting molecular mutations in thyroid nodules fine needle aspiration biopsies[J]. Frontiers in Endocrinology, 2022, 13: 928788-928796.
[12] Jang K S, Kim Y H. Rapid and robust MALDI-TOF MS techniques for microbial identification: a brief overview of their diverse applications[J]. Journal of Microbiology, 2018, 56: 209-216.
[13] Wacharapluesadee S, Hirunpatrawong P, Petcharat S, et al. Simultaneous detection of Omicron and other SARS-CoV-2 variants by multiplex PCR Mass ARRAY technology[J]. Scientific Reports, 2023, 13(1): 2089-2100.
[14] WHO. Statement on the fifteenth meeting of the IHR (2005) Emergency Committee on the COVID-19 pandemic [EB/OL].(2023-05-30) [2023-09-24]. https://www.who.int/news/item/05-05-2023-statement-on-the-fifteenth-meeting-of-the-international-health-regulations-(2005)-emergency-committeeregarding-the-coronavirus-disease-(covid-19)-pandemic.
[15] Zhao F, Lu J, Lu B, et al. A novel strategy for the detection of SARS-CoV-2 variants based on multiplex PCR-mass spectrometry minisequencing technology[J]. Microbiology Spectrum, 2021, 9(3): e0126721.
表1 扩增及延伸引物序列信息
Table 1 Sequences of amplification and extension primers
目标位点 | 正向扩增引物 (5' →3' ) | 反向扩增引物 (5' →3' ) | 延伸引物 (5' →3' ) | 延伸引物质量 (Da) |
Q52H | TAATAAACACCACG | TAGTCTCTAGTCAG | AATCCTCAGTTTTACATTCAACTCA | 7535.0 |
16MPLE | TAATAAACACCACG | TAGTCTCTAGTCAG | GAGATTAAACAGCGGCATGA | 6199.0 |
V483del | ATCATATGGTTTCC | GCTGGTGCATGTAG | GGGTAACAAACCTTGTAATGGTGT | 7431.8 |
V213EG | CTTTCCATCCAAC | GAATTTCAATTTTG | TGAGGGAAATCACGC | 4626.0 |
F456L | ATTATAATTACCAC | TTTTACAGGCTGCG | AATTATAATTACCTGTATAGATTGTT | 7973.0 |
V445AD | ATTATAATTACCAC | TTTTACAGGCTGCG | CAAGCTTGATTCTAAGC | 5169.4 |
P681HR | CTACACTACGTGC | GGTGCAGGTATATG | TGACTAGCTACACTACGTGCCCGCCGA | 8205.4 |
D614G | CAGGTTGCTGTTC | GAATAGCAACAGGG | TTGGGACTTCTGTGCAGTTAACA | 7069.6 |
Uni-N | AAATCAGCGAAAT | TACTGCCAGTTGAATCTG | ACCCCGCATTACGTTTGGTGGACC | 7594.0 |
Uni-ORF1ab | GAATCCTTTGGTGG | ATCCTTTAGGATTTGGA | CATCGTGTTGTCTGTACTGCCGTTGCC | 7622.0 |
表2 多重PCR-MALDI-TOF MS检测结果的判读
Table 2 Interpretation of multiplex PCR-MALDI-TOF MS detection results
目标位点 | 测得的Mass值 (Da) | 对应的延伸碱基 | 判读结果 |
F456La | 7973 | 无延伸 | SARS-CoV-2阴性 |
8300.4 | T | 非EG.5的其他亚型 | |
8244.4 | A | EG.5 | |
Q52Ha | 7535 | 无延伸 | SARS-CoV-2阴性 |
7822.2 | G | 非EG.5的其他亚型 | |
7862.2 | T | EG.5 | |
16MPLEb | 6199 | 无延伸 | BA.2.86或SARS-CoV-2阴性 |
6446.2 | G | 非BA.2.86的其他亚型 | |
V483delb | 7431.8 | 无延伸 | BA.2.86或SARS-CoV-2阴性 |
7759 | T | 非BA.2.86的其他亚型 | |
V213EGc | 4626 | 无延伸 | SARS-CoV-2阴性 |
4953.2 | A | EG.5 | |
4873.2 | G | BA.2.86 | |
4897.2 | T | 非EG.5和BA.2.86的其他亚型 | |
V445ADc | 5169.4 | 无延伸 | 非EG.5和BA.2.86的其他亚型或SARS-CoV-2阴性 |
5416.6 | C | EG.5 | |
5440.6 | A | BA.2.86 | |
P681HRc | 8205.4 | 无延伸 | SARS-CoV-2阴性 |
8492.6 | C | 非EG.5和BA.2.86的其他亚型 | |
8532.6 | A | EG.5 | |
8452.6 | G | BA.2.86 | |
D614Gd | 7069.6 | 无延伸 | SARS-CoV-2阴性 |
7396.7 | A | SARS-CoV-2阳性 | |
7316.8 | G | ||
Uni-Nd | 7594 | 无延伸 | SARS-CoV-2阴性 |
7841.2 | C | SARS-CoV-2阳性 | |
Uni-ORF1abd | 7622 | 无延伸 | SARS-CoV-2阴性 |
7893.2 | A | SARS-CoV-2阳性 |
注: a 为用于EG.5的特异性鉴别的位点; b 为用于BA.2.86的特异性鉴别位点; c 为用于通用鉴别EG.5和BA.2.86的位点; d 为用于通用鉴别SARS-CoV-2的位点.
表2(续)

图1 HCoV-229E株质谱产物峰
Fig.1 Mass spectrum product peak of the HCoV-229E strain

图2 SARS-CoV-2 Alpha株质谱产物峰
Fig.2 Mass spectrum product peak of the SARS-CoV-2 Alpha strain
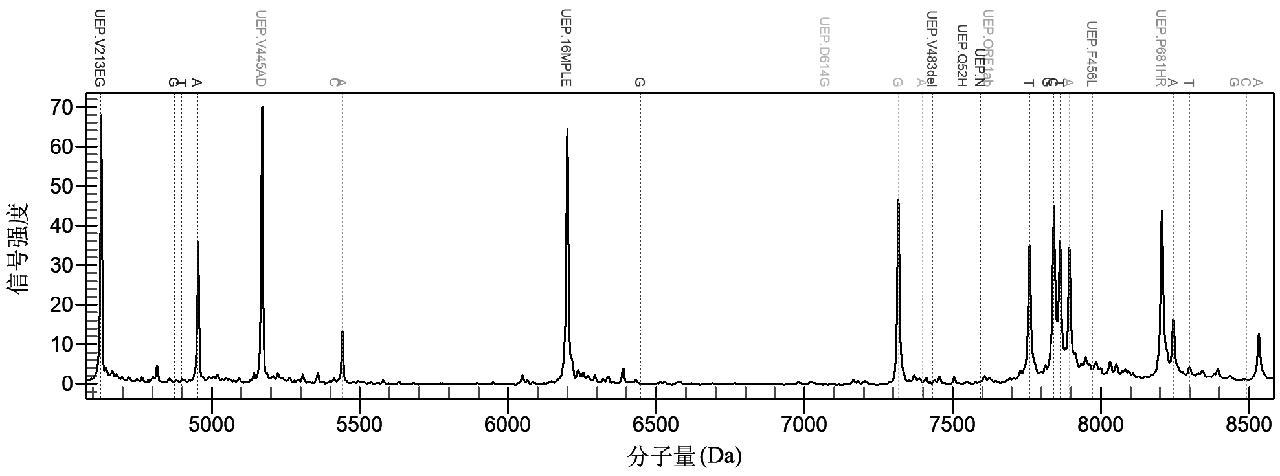
图3 EG.5临界检测限样本
Fig.3 Critical detection limit sample for EG.5

图4 BA.2.86临界检测限样本
Fig.4 Critical detection limit sample for BA.2.86

图5 模拟混合感染样本检测结果
Fig.5 Detection results of simulated mixed infection samples

图6 Omicron XBB.1.16株质谱产物峰
Fig.6 Mass spectrum product peak of the Omicron XBB.1.16 strain

图7 Omicron XBB.1.5株质谱产物峰
Fig.7 Mass spectrum product peak of the Omicron XBB.1.5 strain

图8 Omicron FL.16株质谱产物峰
Fig.8 Mass spectrum product peak of the Omicron FL.16 strain







