CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
基于催化发夹自组装的多种病原微生物阵列检测
作者:黄敏芳 庄妙慧 张习梅 陈岚岚
黄敏芳 庄妙慧 张习梅 陈岚岚
摘 要 本研究建立了一种利用催化发夹自组装反应阵列对多种微生物进行联合检测的新方法。该方法采用双靶点策略,第一个靶点为原核生物16S rRNA的一段保守序列,用于初步判定原核生物,第二个靶点分别为大肠埃希氏菌、单核增生李斯特菌、金黄色葡萄球菌的特异性序列,用于确证微生物种类。双靶点识别后,释放引发链触发催化发夹自组装反应,循环打开带有FAM荧光基团和猝灭基团标记的发夹探针,产生荧光信号。优化反应条件,评价信号的稳定性后,建立对靶标序列检测的工作曲线,通过荧光信号实现对多种微生物的鉴定和定量检测,并建立检测阵列。本方法在靶标浓度为3~50 nmol/L范围内具有良好的线性关系,检出限为0.001 nmol/L。在阵列检测中,本方法能够有效区分3种目标微生物,为多种病原微生物的同时检测提供了新的技术思路。
关键词 病原微生物;催化发夹自组装;阵列检测
Array Detection of Multiple Pathogenic Microorganisms Based on Catalytic Hairpin Assembly
HUANG Min-Fang 1 ZHUANG Miao-Hui 2 ZHANG Xi-Mei 1 CHEN Lan-Lan 1*
Abstract This study establishes a new method for the simultaneous detection of multiple microorganisms using a catalytic hairpin assembly reaction array. The method employs a dual-target strategy. The first target is a conserved sequence of prokaryotic 16S rRNA, used for the preliminary identification of prokaryotes. The second targets are specific sequences for Escherichia coli, Listeria monocytogenes, and Staphylococcus aureus, used for confirming the microbial species. Upon dual-target identification, the trigger chain is released to initiate the catalytic hairpin assembly reaction, cyclically opening hairpin probes labeled with FAM fluorescent groups and quenching groups, thereby generating a fluorescent signal. After optimizing reaction conditions and evaluating signal stability, a working curve of the target sequence detection was established. The method achieves the identification and quantitative detection of multiple microorganisms through the fluorescent signal and establishes a detection array. It demonstrates a good linear relationship within the target concentration range of 3-50 nmol/L and a detection limit of 0.001 nmol/L. In array detection, this method effectively distinguishes three target microorganisms, providing a new technical approach for the simultaneous detection of multiple pathogenic microorganisms.
Keyword pathogenic microorganisms; catalytic hairpin assembly; array detection
基金项目:国家自然科学基金面上项目(22176035)
第一作者:黄敏芳(1998—),女,壮族,广西钦州人,硕士,主要从事分析化学研究工作,E-mail: hmf121599@163.com
通信作者:陈岚岚(1989—),女,汉族,安徽马鞍山人,博士,副研究员,博士生导师,研究方向为生物传感、活体成像分析及纳米药物免疫治疗,E-mail: llchen@fzu.edu.cn
1. 福州大学化学学院 福州 350108
2. 厦门国际旅行卫生保健中心(厦门海关口岸门诊部) 厦门 361001
1. School of Chemistry, Fuzhou University, Fuzhou 350108
2. Xiamen International Travel Health Care Center (Xiamen Customs Port Outpatient Department), Xiamen 361001
病原微生物是指能够引起宿主(如人类、动物、植物等)感染和疾病的微生物,包括细菌、病毒、真菌和原生动物等。病原微生物通过不同的途径(如空气、水、食物、直接接触等)进入宿主体内,引发各种感染性疾病,常见的病原微生物包括大肠埃希氏菌、单核增生李斯特菌、金黄色葡萄球菌等[1]。大肠埃希氏菌(Escherichia coli,E. coli)是一种革兰氏阴性、兼性厌氧的杆状细菌[2],分为致病型和非致病型,在一定条件下可引起人与多种动物的胃肠道感染,症状包括腹部绞痛、腹泻、呕吐等[3]。GB 4789.38—2012《食品安全国家标准 食品微生物学检验 大肠埃希氏菌计数》主要通过采集后进行培养和筛选,进行生化实验后鉴定等步骤对大肠埃希氏菌进行检测[4]。单核增生李斯特菌(Listeria monocytogenes)是革兰氏阳性小杆菌,广泛分布于自然界,能在0~45℃的温度范围内生存[5]。感染单核增生李斯特菌易出现发热、恶心、呕吐症状,严重的可导致脑膜炎和败血症[6]。GB 4789.30—2016《食品安全国家标准 食品微生物学检验 单核细胞增生李斯特氏菌检验》通过增菌、分离、初筛和鉴定等步骤检测李斯特菌[7]。金黄色葡萄球菌(Staphylococcus aureus,S. aureus)是一种革兰氏阳性球菌,能够引起多种严重感染,如肺炎、菌血症、骨髓炎等[8]。GB 4789.10—2016《食品安全国家标准 食品微生物学检验 金黄色葡萄球菌检验》同样通过增菌、分离、初筛和鉴定等步骤检测金黄色葡萄球菌[9]。传统的病原微生物鉴定方法存在检测周期长、灵敏度不高、操作繁琐、成本较高等缺点。
随着新兴检测技术的发展,分子生物学技术通过对生物大分子的直接识别,可以更快速、更特异性地完成检测。例如,聚合酶链式反应(Polymerase Chain Reaction,PCR),通过扩增目标致病菌的DNA,实现快速检测[10];环介导等温扩增(Loop-Mediated Isothermal Amplification,LAMP)技术,无须PCR的严格控温,在等温条件下即可对核酸进行扩增[11]。催化发夹自组装(Catalytic Hybridization Assembly,CHA)是一种无酶介导、反应稳定、背景信号低的核酸信号放大技术[12],其原理是通过链置换反应识别目标核酸链之后进入循环反应,对信号进行指数级放大。Zou等[13]开发了一种基于级联CHA-HCR系统的荧光适配传感器,使用适配体来识别幽门螺杆菌,随后触发无酶信号放大,实现了对临床粪便样本中幽门螺杆菌的超灵敏检测,检出限低至5 CFU/mL。Wang等[14]建立了基于CHA的比率型SERS适配传感方法,用于快速可靠地测定大肠埃希氏菌O157:H7,可在约3 h内完成检测,检测限为2.44×102 CFU/mL。Ren等[15]建立了CHA结合PS微球的微孔电阻DNA检测方法,用于对单核增生李斯特菌的高灵敏度和准确检测[15]。
尽管使用CHA方法检测微生物已经取得了一定进展,但是仍有一些问题亟待解决,如假阳性率高和检测效率低等。本研究以催化发夹自组装为基础,通过双靶点模式提高检测准确性,以及通过矩阵提高检测效率,以建立一种面向多种病原微生物的联合阵列检测新方法。
1 材料与方法
1.1 仪器与试剂
HB120-S LED数显加热金属浴(大龙兴创实验仪器,中国);F-4500荧光光谱仪(HITACHI,日本);ChemiDocTMTouch凝胶成像仪(Bio-Rad,日本);Millipore纯水/超纯水一体化系统(Millipore, 美国)。
氯化钠(NaCl)、过硫酸铵(上海麦克林生化有限公司);1×PBS(磷酸盐缓冲溶液)、5×TBE(Tris-硼酸 电泳缓冲液)、30%丙烯酰胺、N,N,N,N-四甲基乙二胺(生工生物工程(上海)股份有限公司);上样缓冲液(Loading buffer)和20 bp DNA分子量标准(宝日医生物技术有限公司);Gelred(李记生物)。
大肠埃希氏菌、单核增生李斯特菌、金黄色葡萄球菌的16S rRNA中的保守序列和特异性序列分别来源于National Center for Biotechnology Information(NCBI)数据库的16S ribosomal RNA(Genbank:NC_002695.2;NR_044823.1;MN508958.1),分别命名为T1、T2。所有靶标序列及探针均由生工生物工程(上海)股份有限公司合成,序列信息见表1。
1.2 方法
1.2.1 凝胶电泳表征
在12%浓度的聚丙烯酰胺凝胶电泳(Polyacrylamide gel electrophoresis,PAGE),按图1顺序加样验证,每个DNA样品与2 µL的6×loading buffer混合均匀,在1×TBE缓冲液中进行电泳,电压100 V条件下电泳60 min。电泳完成后,使用GelRed对凝胶进行染色,并在ChemiDocTMTouch凝胶成像系统中进行成像。
1.2.2 荧光检测条件优化
钠离子(Na+)浓度优化:配制含0.05 mol/L、0.1 mol/L、0.5 mol/L、0.7 mol/L、0.9 mol/L Na+的1×PBS缓冲液,并用其分别稀释大肠埃希氏菌引发链C、探针H1和H2、占位链O1和O2、靶标链T1和T2,至10 μmol/L进行退火,37℃孵育2 h并测定荧光信号。取H1、H2各10 μL加入各盐浓度缓冲液至终体积为100 μL 37℃孵育2 h并测定荧光信号,作为方法的背景信号。以下荧光检测均以此作为测量背景信号。反应时间优化:用含有0.7 mol/L Na+的1×PBS缓冲液稀释大肠埃希氏菌探针H1和H2至5 μmol/L,稀释引发链C和靶标T1、T2和占位链O1、O2至1 μmol/ L,进行退火,取引发链C和靶标T1、T2和占位链O1、O21 μL与探针H1、H2各10 μL成终体积为100 μL的溶液,混匀立即测定荧光信号,记录反应时间。
荧光光谱仪参数:激发狭缝5 nm,发射狭缝5 nm,电压600 V,激发光波长495 nm,测定范围510~600 nm。
1.2.3 选择性和稳定性验证
幽门螺旋杆菌、志贺氏菌、金黄色葡萄球菌、单核增生李斯特菌、大肠埃希氏菌的靶标DNA各100 μL,引发链C和靶标T1和各细菌靶标T2和占位链O1、O2,终浓度为10 nmol/L,分别使用大肠埃希氏菌探针和单核增生李斯特菌探针和金黄色葡萄球菌探针H1、H2,各探针H1、H2终浓度为500 nmol/L。37℃孵育2 h后进行荧光检测。同时对大肠埃希氏菌DNA的平行样进行测定。依据公式RSD = 计算相对标准偏差评估方法的稳定性,其中,X代表荧光信号值,n代表样品数。
计算相对标准偏差评估方法的稳定性,其中,X代表荧光信号值,n代表样品数。
1.2.4 工作曲线测定
取100 μmol/L的大肠埃希氏菌引发链C,T1、T2、O1、O2,用1×PBS溶液(含0.7 mol/L Na+)稀释至1 μmol/L退火,探针H1、H2、稀释至5 μmol/ L退火;配制0.001~100 nmol/L的引发链,T1、T2、O1、O2;H1、H2浓度为500 nmol/L,37℃孵育2 h结束后进行荧光检测。
1.2.5 联合阵列检测
取未知水样在双盲情况下加标并命名为Sample 1—Sample 8,分别为未加任何靶标的(0, 0);只加了一个T1靶标的(T1, 0);只加3种未知靶标的(T2, 0)和同时加了T1和3种未知靶标的(T1, T2)。各靶标终浓度为10 nmol/L,探针1、2、3分别是大肠埃希氏菌、金黄色葡萄球菌、单核增生李斯特菌探针。各探针H1、H2终浓度为500 nmol/L。按照上述方法37℃孵育2 h结束后,对其进行双靶点阵列检测,每个样品均经历3次不同探针的CHA检测,测量荧光信号。
2 实验结果
2.1 催化发夹自组装反应的凝胶电泳表征
用DNA凝胶电泳验证CHA反应可行性。含有引发链C和占位链O1、O2的溶液中加入靶标T1、T2后,靶标T1、T2与占位链O1、O2互补配对从而被释放。释放的引发链C打开探针发夹H1,打开的H1能打开H2,引发链被置换参与下个循环。泳道5中,H1、H2经过退火后形成发夹,在靶标未加入时,不会引发CHA,发夹单独存在。泳道6中,H1与H2共退火即未形成发夹互补的反应条带即为CHA的反应条带,泳道7中在加入引发链和占位链时,并没有出现泳道6中的结果条带,证明引发链与占位链碱基互补配对成功,在加入引发链C、T1、T2、O1、O2后的泳道9也能观察到泳道6的反应条带,证明CHA反应的可行性。电泳结果如图1所示。
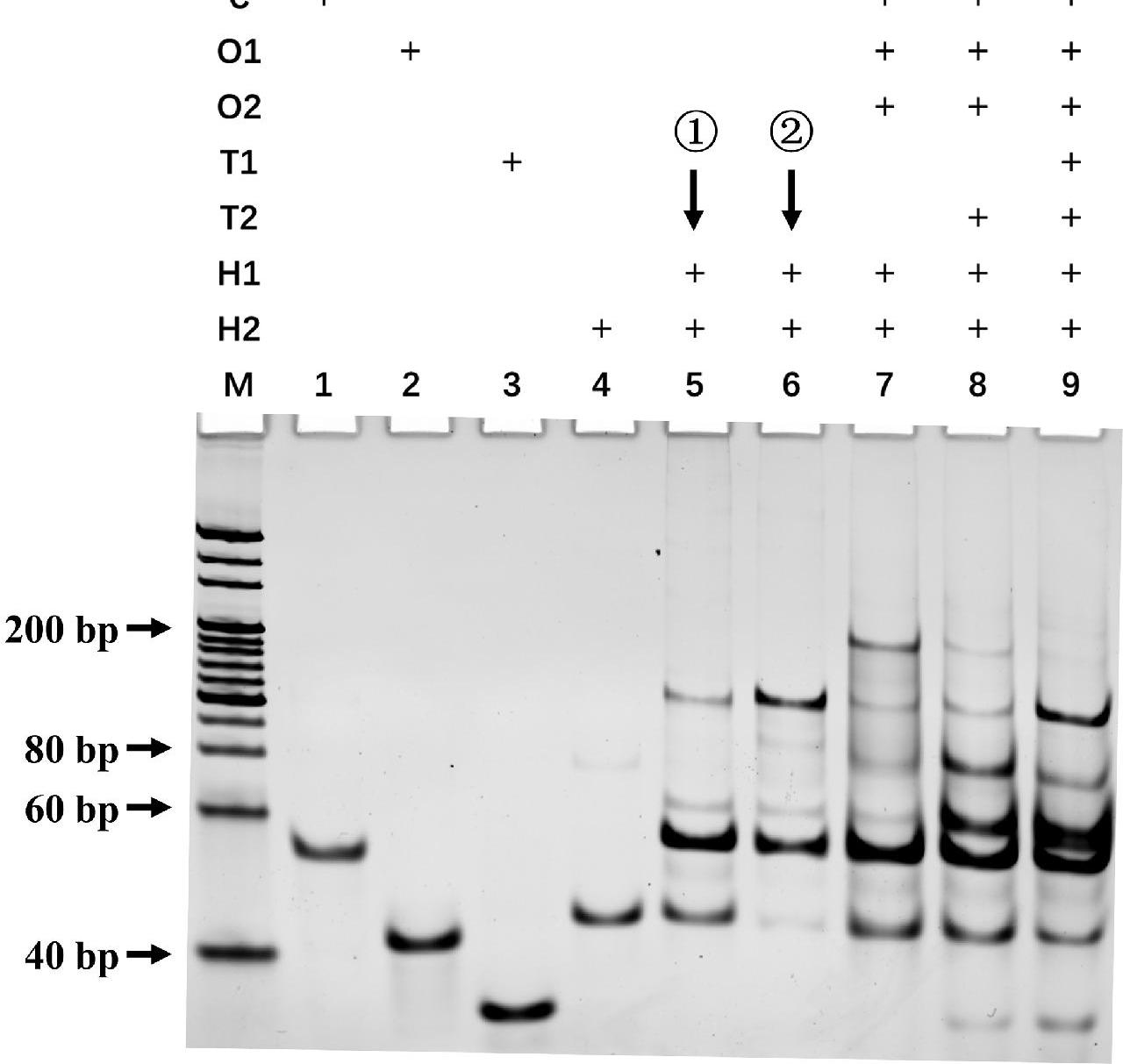
①为H1、H2分别退火形成发夹结构后混合; ②为H1、H2混合后直接退火
图1 催化发夹自组装验证DNA凝胶电泳结果图
Fig.1 Feasibility verification of CHA
2.2 荧光检测条件优化
发夹结构的形成受盐浓度、反应时间影响[16],会进一步影响后续的CHA反应。通过配制含不同浓度Na+的1×PBS缓冲体系,分别进行核酸退火至形成发夹结构后进行孵育,检测荧光信号。如图2所示,荧光信号在0.7 mol/LNa+达到峰值,故以此浓度为反应最佳盐浓度。扫描加入探针H1、H2后的反应过程的荧光信号变化,记录荧光信号随时间的变化。由于CHA是非级联反应且在反应过程中,发夹的空间构型和尺寸会影响与引发链C的相互作用[17],且为了提高探针发夹结构的稳定性和特异性,发夹结构中的茎部互补碱基对较多,导致CHA动力学缓慢。结果如图3所示,荧光信号一直平稳上升,证明链置换反应需要一定的时间,在120 min进入平稳反应时间。
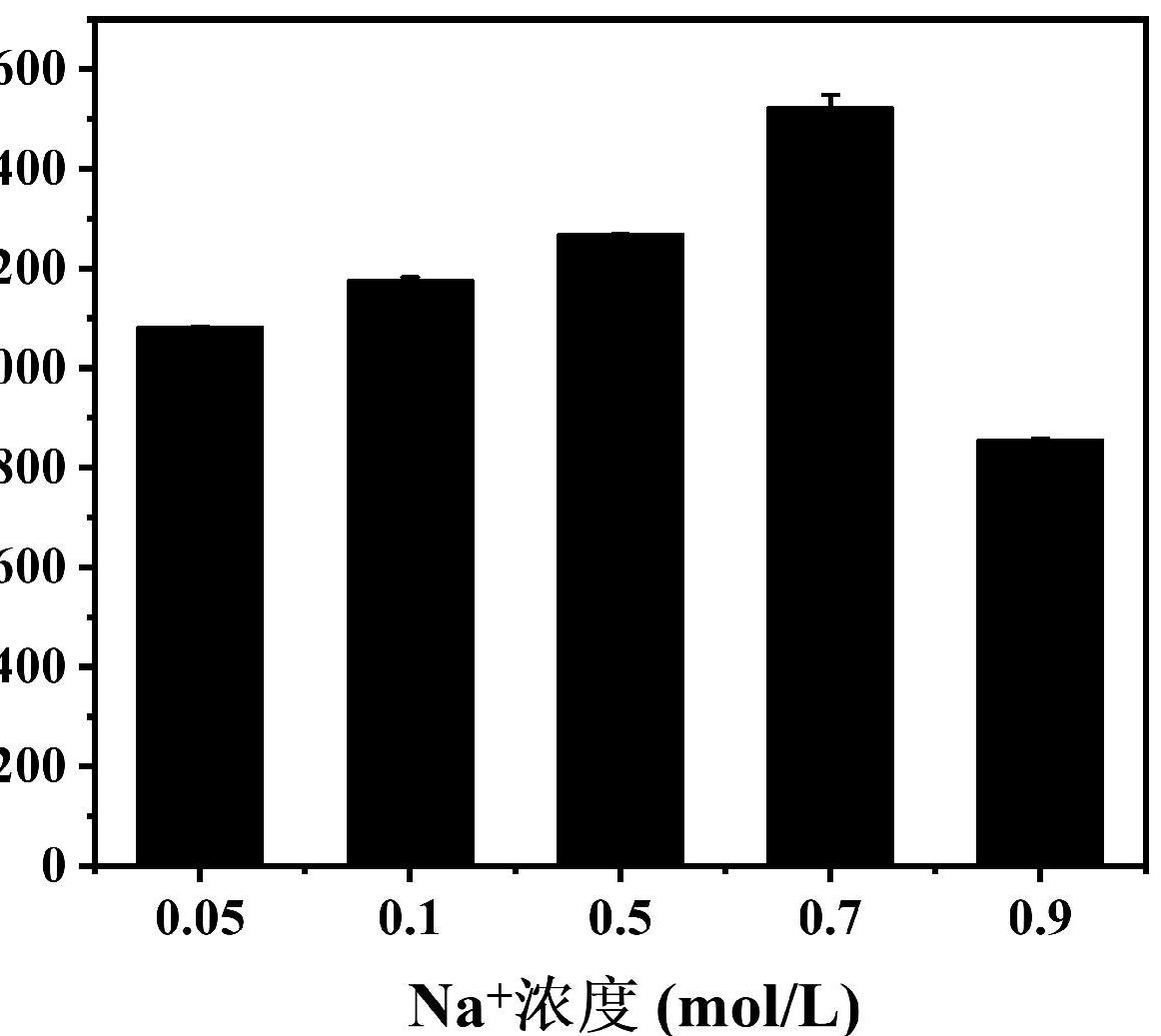
图2 荧光信号随Na+浓度的变化
Fig.2 Fluorescence signal changes with Na+ concentration
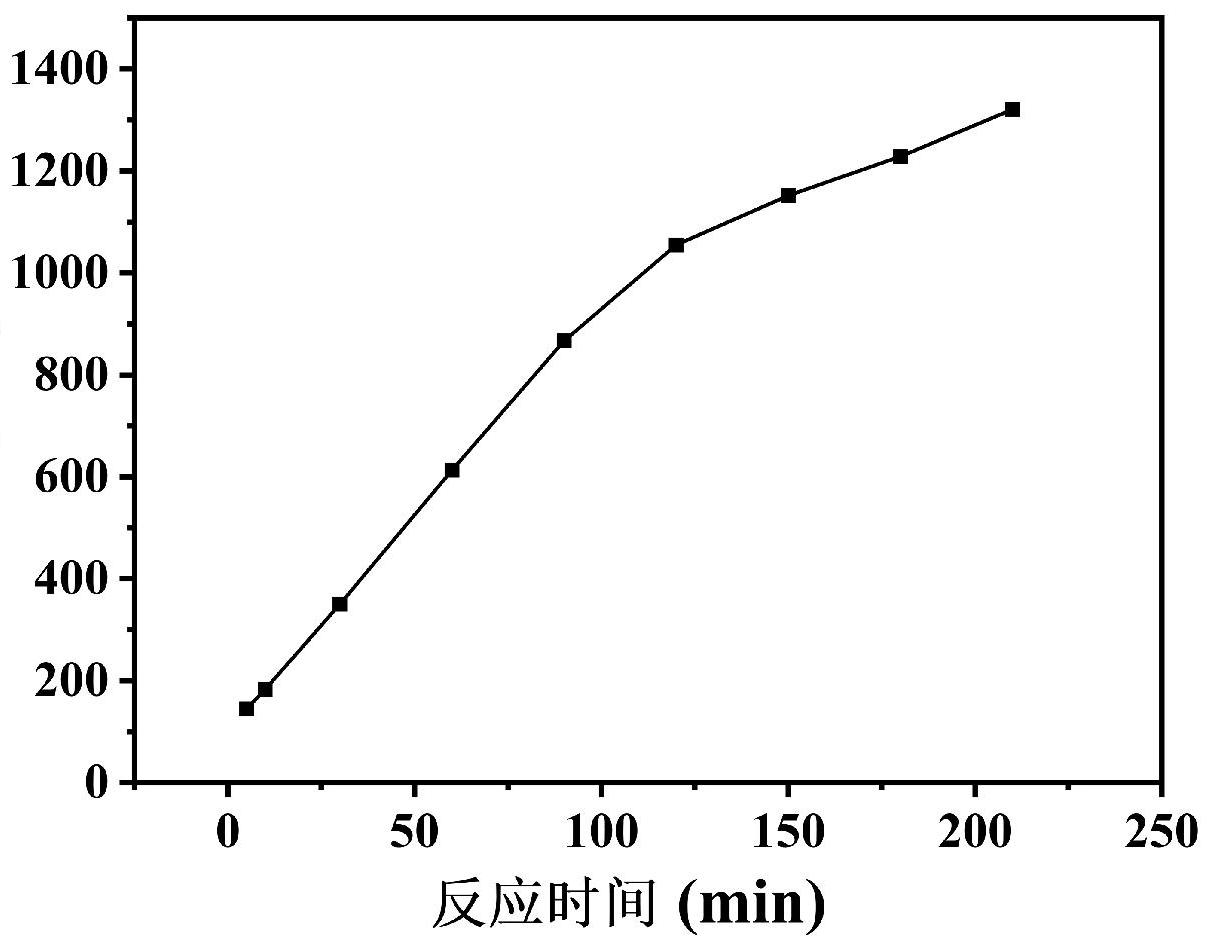
图3 荧光信号随反应时间的变化
Fig.3 Fluorescence signal changes with reaction time
2.3 检测方法的性能分析
按1.2.3所述方法,对含有一定浓度的H1、H2和靶标DNA的溶液进行荧光测定,对平行样品进行检测获得如图4a结果,8份平行样检测均值为1852,相对标准偏差(RSD)为0.03%,证明该反应有良好的稳定性。本研究以大肠埃希氏菌探针为代表,考察探针对于对应靶标的选择性,如图4b所示,对浓度相同的幽门螺旋杆菌、志贺氏菌、金黄色葡萄球菌、单核增生李斯特菌的DNA进行检测,在相同条件下仅有大肠埃希氏菌的靶标序列有较强的荧光信号输出,表明第一组探针对大肠埃希氏菌有特异性的识别能力而不会被其他靶标干扰。为了提高本方法的应用性,使用上述优化条件,在0.001~100 nmol/L 区间测定工作曲线。如图5所示,线性浓度区间为3~50 nmol/L,拟合线性曲线为y = 2634.8x - 940.5,R2 = 0.9999,该方法的检出限即为0.001 nmol/L。
图5 不同浓度靶标DNA所测荧光谱线(a)及其对应检测工作曲线(b)
Fig.5 Fluorescence spectra of CHA with different concentrations of target DNA (a) and the corresponding working curves (b)
2.4 样品检测
为了验证CHA对微生物进行阵列检测的效果,取8份未知样品,按上述方法进行检测,获得荧光信号强度如图6所示,根据图6判定检测结果见表2。
3 讨论
3.1 实验原理
16S rRNA(16S核糖体RNA)是原核微生物核糖体的一个重要组成部分,对于所有细菌和古菌都是普遍存在的,序列中有高度保守区间,在不同种属微生物中具有一致性,也有特异性区间,可用于鉴别微生物种属[18]。
本研究采用双靶点策略进行微生物种属鉴定。实验原理如图7所示,首先设计了由1条引发链和2条发卡探针组成的CHA循环体系,发卡探针的两端分别修饰了荧光基团6-FAM和猝灭基团BHQ-1,退火完成后发卡呈闭合状态,两端修饰的基团由于荧光共振能量转移而猝灭。引发链C与发卡链H1结合后,打开发卡,使荧光基团远离猝灭基团,荧光信号恢复,同时打开的H1发卡的环部序列恢复配对活性,从而可以与第二个发卡探针H2反应,打开H2,2个发卡结合后由于链取代作用而使之前结合的引发链离开H1,恢复自由状态,可以进入下一个循环,从而形成了循环反应,一经触发就产生高灵敏荧光信号[12]。然后,使用两条占位链锁住引发链C,仅当2条占位链被结合后,引发链才能恢复自由状态,进入CHA循环。而靶标T1和T2分别与占位链结合,仅当两者同时完成反应时引发链才可以被释放。实验选取了16S rRNA中的保守序列为T1,每个原核微生物的T1序列均一致,种属特异性序列为T2,每个病原微生物的T2序列不同,通过这种方式完成确证式检测,仅当两者均检出时才判定为检出,避免单靶点检测产生的假阳性。
DNA凝胶电泳验证结果表示,只有加入靶标T1、T2,之后占位链O1、O2与T1、T2互补配对释放出引发链与探针H1、H2发生CHA反应。以上结果表明检测体系的核酸反应可以按照设计路线进行。
3.2 方法检测性能
本研究在优化条件的基础上,通过平行样检测验证了该检测方法的性能,证明该方法具有良好的稳定性。一方面是因为荧光检测具有稳定性,另一方面是设计的催化发夹结构具有高度特异性和热力学稳定性,能够减少非特异性结合,重现性较好。
CHA反应是链置换的无酶核酸扩增反应,设计的高度特异性发夹结构只有在目标序列存在才能触发发夹的打开和后续反应,单靶点匹配或不匹配的微生物核酸无法引发CHA反应。该体系灵活的发夹结构设计,适应同时不同的检测目标,靶标DNA浓度在3~50 nmol/L荧光信号强度呈显著的正相关,在未知样的检测时,只有对应靶标存在时才有明显荧光信号输出,CHA与多重阵列检测相结合能够对多个样品进行分析,简化检测操作,显著提高检测效率,在实际应用中尤为重要。本方法的检测限可达0.001 nmol/L,能够满足实际样本检测需求。
3.3 阵列检测与双靶点确证
阵列检测可以使用多个传感器或检测单元对样品进行同时检测,阵列检测能够提供比单一检测更丰富的信息和数据[19]。在核酸检测中,非特异性结合会引发假阳性信号导致判断失误,例如,发夹之间的非特异性结合会导致没有靶标时仍出现信号输出[20]。本研究中依据CHA的特性,根据两种靶标设计的3种不同探针,能满足阵列检测多个检测器的要求。
所检测出的荧光信号阵列图如图6所示。结果判定如表2所示,大肠埃希氏菌探针对样品6有高荧光信号值输出,而对其他样品呈低荧光信号输出,证明只有当同时存在T1和该病原微生物的特异性序列T2时,才能对此病原微生物进行判定。仅加入T1或T2的低荧光信号证明该方法有良好的抗干扰性。同样地,金黄色葡萄球菌和单核增生李斯特菌的探针也仅在对应靶标存在的情况下才能产生强信号,表明3组探针均对于对应靶标有较好的选择性,这是阵列检测的理论基础。3种探针同时检测8份样品,即在较短时间内能够完成大量样品的检测,大大节省了实验时间,同时双靶点检测能够有效降低假阳性信号影响,降低误判的风险。
4 结论
综上所述,本研究以催化发夹自组装为基础,建立了对多种病原微生物进行阵列检测的检测方法,可广泛应用于海关口岸卫生检疫工作中,既有利于提高检测质量,又有助于简化操作,提高检测效率,提升口岸卫生检疫水平。这种阵列检测模式利用CHA无酶放大且能设计多个探针的优势,结合荧光检测,达到对多种病原微生物进行阵列检测的目的,而且有广阔的拓展空间,可以根据疫情风险评估和口岸的实际检疫需求增加目标微生物,因此具有良好的应用潜力。
参考文献
[1] 李珂, 张洪勋, 余志晟, 等. 中国水环境微生物污染研究进展[J]. 科技导报, 2021, 39(15): 110-116.
[2] Denamur E, Clermont O, Bonacorsi S. et al. The population genetics of pathogenic Escherichia coli[J]. Nature Reviews Microbiology, 2021, 19(1): 37-54.
[3] Croxen, M., Finlay, B. Molecular mechanisms of Escherichia coli pathogenicity [J]. Nature Reviews Microbiology, 2010, 8(1): 26-38.
[4] GB 4789.36—2016. 食品安全国家标准 食品微生物学检验 大肠埃希氏菌 O157:H7/NM检验[S].北京:中国标准出版社, 2016.
[5] Maury, M., Tsai, YH., Charlier, C.et al. Uncovering Listeria monocytogenes hypervirulence by harnessing its biodiversity[J]. Nature Genetics, 2016, 48, 308-313.
[6] Disson Olivier, Moura Alexandra, Lecuit Marc. Making Sense of the Biodiversity and Virulence of Listeria Monocytogenes[J]. Trends in Microbiology, 2021, 29(9): 811-822.
[7] GB 4789.30—2016. 食品安全国家标准 食品微生物学检验 单核细胞增生李斯特氏菌检验[S]. 北京:中国标准出版社, 2016.
[8] Keim Klara C, Horswill Alexander R. Staphylococcus aureus[J]. Trends in Microbiology, 2023, 31(12): 1300-1301.
[9] GB 4789.10—2016.食品安全国家标准 食品微生物学检验 金黄色葡萄球菌检验 [S]. 北京:中国标准出版社, 2016.
[10] McMahon Tanis, Clarke Sarah, Deschênes. et al. Real-time PCR primers and probes for the detection of Shiga toxin genes, including novel subtypes[J]. International Journal of Food Microbiology, 2024, 419: 110744.
[11] Feng Junl, Dai Zhiyuan, Tian Xiaolan, et al. Detection of Listeria monocytogenes based on combined aptamers magnetic capture and loop-mediated isothermal amplification[J]. Food Control, 2018, 85: 443-452.
[12] Yin Peng, Choi Harry M T, Calvert Colby R. et al. Programming biomolecular self-assembly pathways[J]. Nature, 2008, 451(7176): 318-322.
[13] Zou Haimin, Lin Chiliang, Zan Haocheng. et al. A novel fluorescent aptasensor for ultrasensitive detection of Helicobacter pylori in stool samples based on catalytic hairpin assembly cascade hybridization chain reaction[J]. Sensors and Actuators B: Chemical, 2022, 368: 132157.
[14] Wang Panxue, Liu Ying, Li Xiang. et al. A ratiometric SERS aptasensor based on catalytic hairpin self-assembly mediated cyclic signal amplification strategy for the reliable determination of E. coli O157:H7[J]. Microchimica Acta, 2024, 191(8): 441.
[15] Ren Liangqiong, Hong Feng, Chen Yiping. Enzyme-free catalytic hairpin assembly reaction-mediated micro-orifice resistance assay for the ultrasensitive and low-cost detection of Listeria monocytogenes[J]. Biosensors and Bioelectronics, 2022, 214: 114490.
[16] Deng Chunyan, Pi Xiaomei, Qian Pin. et al. High-Performance Ratiometric Electrochemical Method Based on the Combination of Signal Probe and Inner Reference Probe in One Hairpin-Structured DNA[J]. Analytical Chemistry, 2017, 89(1): 966-973.
[17] Mo Liuting, Liang Danlian, Qin Runhong. et al. Three-Dimensional CHA-HCR System Using DNA Nanospheres for Sensitive and Rapid Imaging of miRNA in Live Cells and Tissues[J]. Analytical Chemistry, 2023, 95(31): 11777-11784.
[18] Anagnostopoulos Dimitrios A, Syropoulou Faidra Parlapani Foteini F. et al. Microbiota profile of filleted gilthead seabream (Sparus aurata) during storage at various conditions by 16S rRNA metabarcoding analysis[J]. Food Research International, 2023, 164: 112312.
[19] Zhang Xiaorong, Qiu Hongzhao, Zhong Xinyi. et al. A CRISPR/Cas12a-assisted array for Helicobacter pylori DNA analysis in saliva[J]. Analytica Chimica Acta, 2023, 1239: 340736.
[20] Lv Zhaoyue, Huang Mengxue, Li Peiran. et al. Hybridization chain reaction-based DNA nanomaterials for biosensing, bioimaging and therapeutics[J]. Chinese Chemical Letters, 2024, 35(2): 108601.
表1 核酸序列表
Table 1 DNA sequences used in biosensing
细菌/探针 | 序列 (5'~3' ) |
占位链 (O1) | TTACCGCGGCTGGTCCTTGAGGCTGGCACGTAGTTA |
保守序列 (T1) | TAACTACGTGCCAGCAGCCGCGGTAA |
引发链 (CE. coli) | TAACTACGTTCTCAAGGACCAGGCCGCATCTCTACGTGGGAAAG |
占位链 (O2E. coli) | CTTTCCCACCAACGCTAGAGATGCGGGCTCAATTCC |
特异性 (T2E. coli) | GGAATTGAGCAGCGTTGGTGGGAAAG |
H 1(E. coli) | CGCATCTCTACGTGTAGACCTCGTCATCAGGTAGAGATGCGGCCTGG |
H2 (E. coli) | TCTACGCCTAGACCTCGTCATCCGCATCTCTACGTGGATGACGAGGTCTAGGCGTAGAGATGCC |
C (Listeria) | TAACTACGTTCTCAAGGACCAGGCCGCATCTCTACGCCAGCCGC |
T2 (Listeria) | AGGGTCACCTTTATGGAGCCAGCCGC |
O2 (Listeria) | GCGGCTGGCTCCATATAGAGATGCGGAGGTGACCCT |
H1 (Listeriar) | CGCATCTCTACGCCTAGACCTCGTCATCGGCGTAGAGATGCGGCCTGG |
H2 (Listeria) | TCTACGCCTAGACCTCGTCATCCGCATCTCTACGCCGATGACGAGGTCTAGGCGTAGAGATGCC |
C (S. aureus) | TAACTACGTTCTCAAGGACCAGGCCGCATCTCTACCAAGCAAAT |
T2 (S. aureus) | CAGCGAAACCGCGAGGTCAAGCAAAT |
O2 (S. aureus) | ATTTGCTTGACCTCGTAGAGATGCGGGGTTTCGCTG |
H1 (S. aureus) | CGCATCTCTACCAATAGACCTCGTCATCTTGGTAGAGATGCGGCCTGG |
H2 (S. aureus) | TCTACGCCTAGACCTCGTCATCCGCATCTCTACCAAGATGACGAGGTCTAGGCGTAGAGATGCC |
图4 荧光检测平行样品的稳定性表征结果(a)和荧光检测不同微生物靶标核酸的特异性表征结果(b)
Fig.4 Characterization of the stability of parallel samples by fluorescence detection (a), specificity characterization results of different microbial target nucleic acids by fluorescence detection (b)
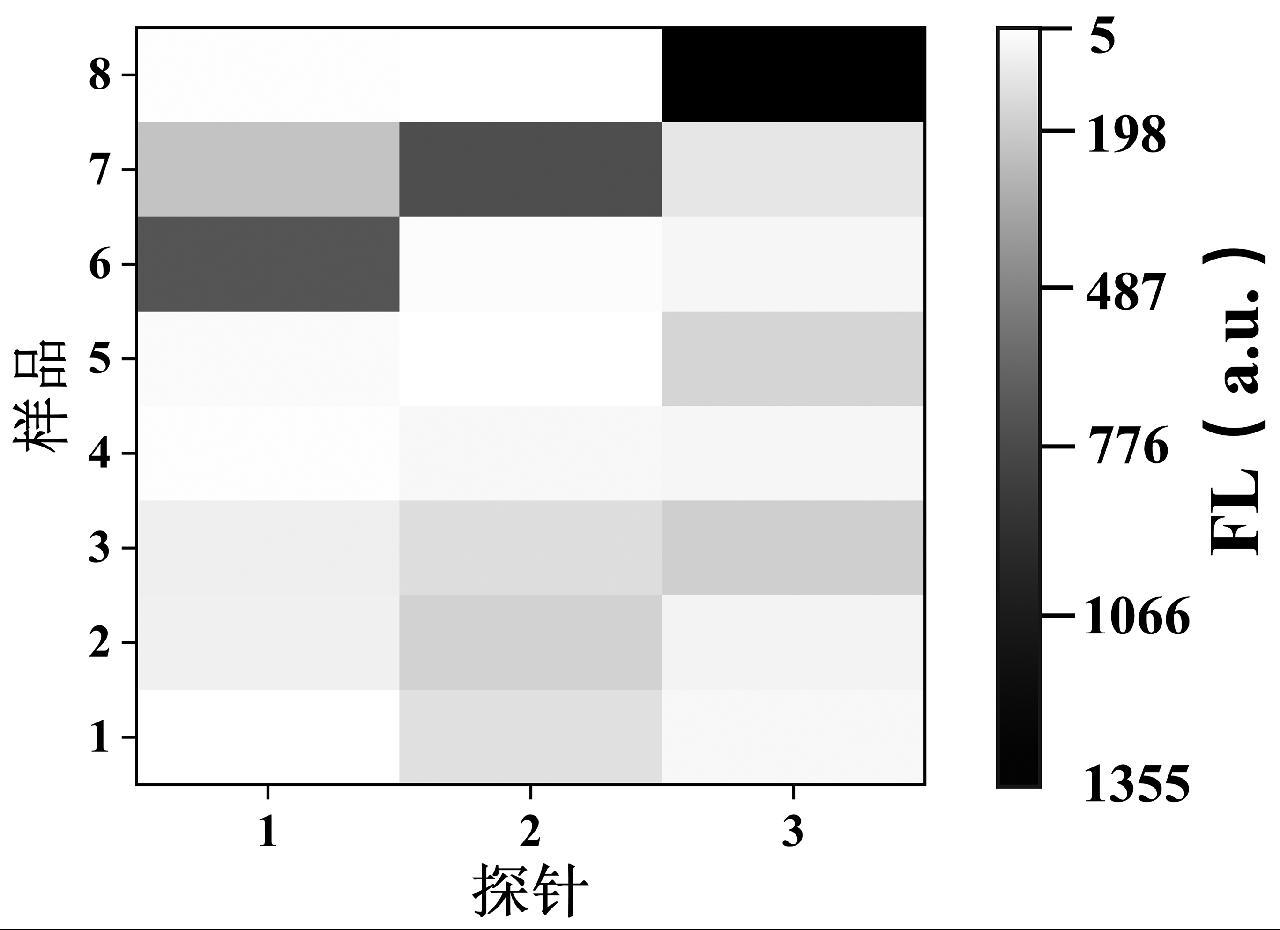
图6 3种探针检测未知样的荧光信号强度阵列图
Fig.6 Array diagram of fluorescence signal intensity of three probes detecting unknown samples
表2 3种探针检测未知样的加样和结果判定
Table 2 Sample addition and result determination of unknown samples using three probes
样品 | 加样 | 探针 | 探针 | 探针 |
1 | (0, 0) | off | off | off |
2 | (T1, 0) | off | off | off |
3 | (0, T2(未知)) | off | off | off |
4 | (0, T2(未知)) | off | off | off |
5 | (0, T2(未知)) | off | off | off |
6 | (T1, T2(未知)) | on | off | off |
7 | (T1, T2(未知)) | off | on | off |
8 | (T1, T2(未知)) | off | off | on |
注: “on”代表阳性; “off ”代表阴性; CHA检测中, 以荧光读数高于140为阳性, 低于140为阴性.
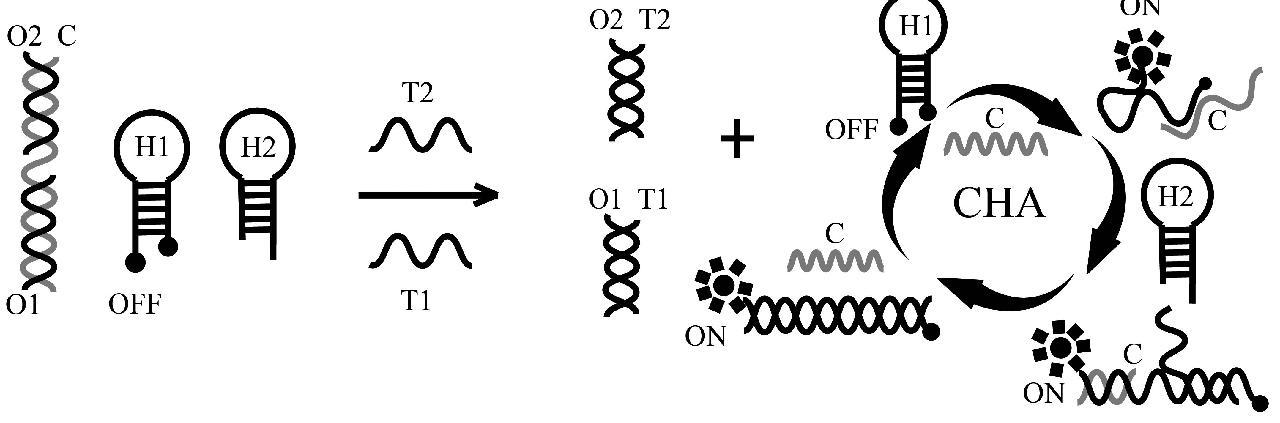
图7 CHA反应实验原理示意图
Fig.7 Schematic diagram of CHA reaction experimental principle







