CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
熊科动物COX2基因DNA条形码鉴定方法的建立
作者:邵建宏 蒋晓霞 张洪 刘云国 麻丽丹 莫秋华 蔡一村 罗宝正
邵建宏 蒋晓霞 张洪 刘云国 麻丽丹 莫秋华 蔡一村 罗宝正
摘 要 为有效鉴定熊科(Ursidae)物种,根据熊科动物线粒体细胞色素c氧化酶2(cytochrome c oxidase subunit 2,COX2)基因设计扩增片段为790 bp的通用引物,对9份熊科动物组织样本、4份熊科动物合成质粒和16份对照样本进行聚合酶链式反应(Polymerase Chain Reaction,PCR) 扩增、电泳和测序,计算物种遗传距离,并采用邻接法构建进化树。结果显示:16份对照样本均未见扩增条带;9份熊科动物组织DNA和4份熊科动物合成质粒的检测结果符合预期,且与美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)参考序列的一致性均>96%;8种熊科动物的最大种内遗传距离为0.014%,最小种间遗传距离为0.022%,存在显著条形码差距。结果表明,本研究所建立的COX2基因DNA条形码方法能够准确鉴定熊科物种,可为海关等执法部门打击违法犯罪行为、履行《濒危野生动植物种国际贸易公约》提供技术支撑。
关键词 熊;DNA条形码;细胞色素c氧化酶2;鉴定
Establishment of an Ursidae Identification Method through COX 2 Gene Base on DNA Barcoding
SHAO Jian-Hong1 JIANG Xiao-Xia1 ZHANG Hong1 LIU Yun-Guo2
MA Li-Dan1 MO Qiu-Hua3 CAI Yi-Cun4 LUO Bao-Zheng1*
Abstract To effectively identify the species of Ursidae, a universal primer with an amplification fragment of 790 bp was designed based on the mitochondrial cytochrome c oxidase 2 (COX2) gene of Ursidae, and nine Ursidae tissue samples, four Ursidae synthetic plasmids, and sixteen control samples were subjected to polymerase chain reaction(PCR) amplification, electrophoresis, and sequencing. Genetic distances of the species were calculated, and an evolutionary tree was constructed using the neighbor-joining method. The results showed that no amplified bands were observed in the eighteen control samples. In contrast, the nine Ursidae tissue DNAs and the four Ursidae synthetic plasmids exhibited expected detection results, and the consistency with the NCBI reference sequences was >96%. Moreover, the maximum intraspecies genetic distance of the eight species of Ursidae was 0.014%, while the minimum interspecies genetic distance was 0.022%, indicating a significant barcoding gap. The study demonstrates that this DNA barcoding method for the COX2 gene can accurately identify the species of Ursidae, which can provide technical support for Customs and other law enforcement departments to combat illegal and criminal behaviors and fulfill the Convention on International Trade in Endangered Species of Wild Fauna and Flora (CITES).
Keywords ursidae; DNA barcoding; cytochrome c oxidase subunit 2(COX2); identification
基因项目:海关总署科研项目(2022HK003)
第一作者:邵建宏(1983—),男,汉族,山东临朐人,本科,高级兽医师,主要从事动物传染病检测与动物源性成分鉴定研究工作,E-mail: shaojianhong@qq.com
通信作者:罗宝正(1975—),男,汉族,山东莱西人,博士,研究员,主要从事动物传染病检测与动物源性成分鉴定研究工作,E-mail: bzluo@163.com
1. 拱北海关技术中心 珠海 519001
2. 临沂大学生命科学学院 临沂 276012
3. 深圳市易瑞生物技术股份有限公司 深圳 518101
4. 上海海关动植物与食品检验检疫技术中心 上海 200135
1. Technical Center of Gongbei Customs, Zhuhai 519001
2. College of Life Sciences, Linyi University, Linyi 276012
3. Shenzhen Bioeasy Biotechnology Co., Ltd., Shenzhen 518101
4. Technical Center for Animal, Plant and Food Inspection and Quarantine, Shanghai Customs, Shanghai 200135
熊的生物分类学归类为脊索动物门、脊椎动物亚门、哺乳纲、食肉目、熊科,现存共5属8种,均被列入世界自然保护联盟(International Union for Conservation of Nature,IUCN)红色名录。熊属于大型哺乳动物,处于食物链顶端,其种群数量的稳定性对生态平衡的稳定性至关重要。IUCN红色名录公布的资料显示,除大熊猫和美洲黑熊的数量有所增加、棕熊的数量保持稳定外,其余5种熊科动物的数量仍然持续减少,提示当前的保护措施未能在总体上有效缓解熊科动物的濒危困境。另外,熊胆在传统中医文化中被认为具有特殊药用价值,最早可追溯至唐代甄权的《药性论》[1]。虽然我国已将境内分布的5种熊科动物全部纳入《国家重点保护野生动物名录》,严禁野外捕猎,部分学者也通过实验数据和临床观察结果证明了使用熊胆替代品的可行性,呼吁取缔活熊取胆,但非法猎杀黑熊、贩卖熊组织的情况仍然时有发生。传统的形态学鉴定法对目标样本的完整性和形态特征要求严格,鉴定过程主观性强,理化鉴定法受目标样本的均一性影响大,难以标准化、无法鉴定到种。因此,迫切需要确立一种准确、可靠且能够将鉴定流程标准化的分子生物学检测鉴定方法,精确鉴定熊科动物及其产品的物种来源,通过不同基因段对熊科动物的物种分类进行鉴定和比较,也能够进一步丰富熊科动物分化和进化的研究材料,为后续明确其物种发育关系提供研究基础。该方法还可以进一步打击猎杀和贩卖野生保护动物的违法犯罪行为,履行《濒危野生动植物种国际贸易公约》(Convention on International Trade in Endangered Species of Wild Fauna and Flora,CITES),助力维护生态安全。
本研究采用的DNA条形码技术利用一个或多个标准化、易于扩增且长度相对较短的DNA片段进行目标DNA扩增,而后将扩增产物的测序结果与DNA条形码参考库中序列进行比对,从而明确目标物种的归类[2],具有高效、准确、成本低廉等优势,成为应用最为广泛的物种鉴定技术手段,能够充分满足本研究的目的。
1 材料与方法
1.1 材料
拱北海关技术中心动物检疫实验室(以下简称“本实验室”)存留的熊科动物组织(目标样本)9份(表1);本实验室存留的陆生和水生动物组织(对照样本)16份,均使用本实验室自行设计,经中国合格评定国家认可委员会(China National Accreditation Service for Conformity Assessment,CNAS)认证的分子生物学鉴定方法确认为所列物种(表2);本实验室委托北京六合华大基因科技有限公司合成的熊科动物参考序列质粒(目标序列)4份(表3)。
1.2 样本DNA提取
为确保提取的DNA质量,使用无水乙醇均匀擦拭样本表面数次后风干,以清除附着在样本表面的异物,使用合适的无菌采样器具分别取样、剪碎,并于研磨钵辅以液氮研磨稀碎,称取约50 mg装入离心管待检。待检样本按照E.Z.N.A.® Tissue DNA Kit D3396-02核酸提取试剂盒(美国OMEGA公司)说明书提取DNA,并于-20℃保存备用。
1.3 PCR引物设计与合成
使用DNAMAN软件对从美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)获取的现存熊科物种线粒体基因序列进行多重比;使用Oligo软件在细胞色素c氧化酶2(cytochrome c oxidase subunit 2,COX2)基因的高度保守区域设计一对通用PCR引物,正向引物F-primer序列:5'-GGTGAAAATCCTTTATCTTTATGGC-3'、反向引物R-primer序列:5'-AGTTGTGGCATCTTCATT AAGGAG-3',扩增片段预期大小约790 bp。上述引物委托上海辉瑞生物科技有限公司合成。
1.4 PCR扩增
以目标样本和对照样本提取的DNA、目标序列质粒、阴性对照和空白对照为模板进行常规PCR扩增,反应体系:2×Taq PCR Mastermix 12.5 μL,上、下游引物(10 μmol/L)各1.0 μL,DNA模板2.0 μL,补水至总反应体积25.0 μL。反应程序:94℃预变性4 min;94℃变性30 s,58℃退火30 s,72℃延伸60 s,共35个循环;72℃完全延伸10 min。
1.5 电泳和测序
取5 μL扩增产物在质量浓度为1.5%的琼脂糖凝胶进行电泳,100 V 35 min,通过凝胶成像分析系统观察、分析,对与目标片段大小相符的条带进行切胶、纯化,由本实验室采用桑格测序法进行测序。
1.6 序列分析
通过NCBI的序列检索平台基本局部比对搜索工具(Basic Local Alignment Search Tool,BLAST)对PCR阳性产物相应测序序列进行比对,获取目标序列一致性等相关信息,鉴定物种来源。将4种共9条目标样本DNA序列和8种共16条熊科动物线粒体DNA序列(表4),共计25条序列进行两两比较,通过MEGA软件的两参数(Kimura-2-parameter,K2P)模型计算各物种的遗传距离,以最大复合似然法估算、碱基转换、1000次重采样和完全删除空链为基础参数条件构建邻接法(Neighbor-Joining Method,NJ)进化树。
表4 16条熊科动物线粒体DNA序列
Table 4 Sixteen mtDNA sequences of Ursidae
序列序号 | 物种 | 学名 | NCBI登录号 |
2 | 亚洲黑熊 | Ursus thibetanus | PQ182686.1:7898-8580 |
4 | 马来熊 | Helarctos malayanus | FM177765.1:7955-8637 |
6 | 眼镜熊 | Tremarctos ornatus | NC_009969.1:7899-8636 |
8 | 懒熊 | Melursus ursinus | MH910095.1:7514-8263 |
10 | 美洲黑熊 | Ursus americanus | OQ318937.1:7629-8309 |
12 | 棕熊 | Ursus arctos | OK512925.1:7617-8312 |
14 | 北极熊 | Ursus maritimus | OK001275.1:8198-8893 |
16 | 大熊猫 | Ailuropoda melanoleuca | EF196663.1:7876-8562 |
1.7 特异性与灵敏度试验
对4份目标样本提取的DNA和16份对照样本提取的DNA按照1.4的试验方法分别进行PCR扩增,以验证方法的特异性。将含有马来熊目标基因片段的质粒DNA进行10倍梯度稀释,共稀释为8个梯度,质粒DNA的浓度范围分别为4.3×108~4.3×101 拷贝/μL,对稀释后的质粒DNA按照1.4的试验方法分别进行PCR扩增,以验证方法的灵敏度。
2 结果与讨论
2.1 扩增产物电泳结果
编号1—9的熊科动物组织样本和序号1—4的熊科动物合成质粒在约790 bp处均呈现显著扩增条带,阴性对照和空白对照均未见扩增条带(图1、图2)。
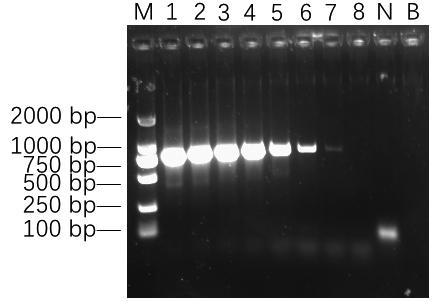
样本编号: 1—9; N: 阴性对照; B: 空白对照
图1 熊科动物组织样本DNA扩增产物电泳结果
Fig.1 Electrophoresis results of amplified products for tissue samples of Ursidae
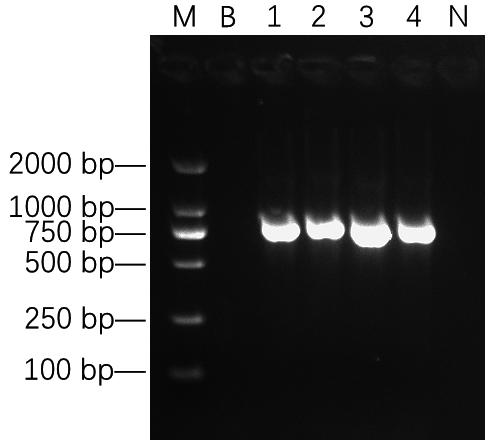
合成质粒序号: 1—4; N: 阴性对照; B: 空白对照
图2 熊科动物合成质粒扩增产物电泳结果
Fig.2 Electrophoresis results of amplified products for synthetic plasmids of Ursidae
2.2 序列比对结果
样本编号1—5为亚洲黑熊、样本编号6—7为棕熊、样本编号8为北极熊、样本编号9为大熊猫,所有序列比对结果的一致性均≥98%(表5);合成质粒序号1—4分别为马来熊、懒熊、眼镜熊和美洲黑熊,所有序列比对结果的一致性均≥96%(表6)。
表5 样本编号1—9序列BLAST结果
Table 5 BLAST results for the sequences of sample No. 1 to 9
样本编号 | 动物组织 | 学名 | 一致性 (%) |
1 | 熊胆 | Ursus thibetanus | 98.07~99.11 |
2 | 熊胆 | Ursus thibetanus | 99.41~99.56 |
3 | 熊爪 | Ursus thibetanus | 98.04~98.95 |
4 | 熊胆 | Ursus thibetanus | 98.00~99.50 |
5 | 熊肉 | Ursus thibetanus | 99.00~99.86 |
6 | 熊爪 | Ursus arctos | 99.59~99.73 |
7 | 熊毛 | Ursus arctos | 99.57~99.86 |
8 | 熊毛 | Ursus maritimus | 99.42~99.42 |
9 | 熊肝 | Ailuropoda melanoleuca | 99.69~100.0 |
表6 合成质粒序号1—4序列BLAST结果
Table 6 BLAST results for the sequences of synthetic plasmid No.1 to 4
合成质粒序号 | 学名 | 一致性 (%) |
1 | Helarctos malayanus | 98.17~99.72 |
2 | Melursus ursinus | 98.37~99.86 |
3 | Tremarctos ornatus | 99.31~99.86 |
4 | Ursus americanus | 96.96~100.0 |
2.3 遗传距离与进化树分析
在遗传距离方面,25条序列的两两比较结果显示序列间的最大种内遗传距离为0.014,出现于两条亚洲黑熊组织样本DNA序列;最小种间遗传距离为0.022,出现于一条北极熊组织样本DNA序列和一条基因库棕熊线粒体DNA序列;北极熊组织样本DNA序列与棕熊组织样本DNA序列的遗传距离为0.024,仅次于两个物种之间非单纯组织样本的遗传距离。上述结果提示,最小种间遗传距离大于最大种内遗传距离,表明不同熊科物种的DNA条形码序列之间存在显著差距(表7)。在进化关系方面,25条序列在NJ进化树中引出7个不同的进化分支,归集为8个熊科物种,其中眼镜熊和大熊猫与其他6个物种的进化关系相对较远,而棕熊与北极熊的进化关系相对较近,9份共4种熊科组织样本与相应物种的参考序列亦分别归为一支(图3)。
2.4 特异性与灵敏度
4份目标样本提取的DNA特异性扩增条带显著,16份对照样本提取的DNA未见扩增条带(图4),扩增条带亮度随质粒DNA浓度的降低逐渐下降,检出限为4.30×102 拷贝/μL(图5)。
2.5 讨论
非法捕杀珍稀野生动物的行为在全球各地屡禁不止,野生动物的生存和种群多样性面临巨大威胁,不法分子常通过瞒报、谎报、走私濒危野生动物等非法行径逃避监管和处罚,因此,准确鉴定形态相似的动物组织及其产品对于监管部门的执法具有重要意义。传统的形态学鉴定法是辨别组织样本固有特征的一种经济、高效的方法,鉴定专家能够通过数字图像信息作出鉴定结论,为海关等执法部门的后续工作提供重要参考信息,在某些执法案件中,该类线索可能成为唯一可供鉴定的野生动物相关犯罪证据[3]。但形态学鉴定工作不仅要求鉴定人员具备充分的专业知识和丰富的鉴定经验,对目标样本的完整性、清晰度、组织部位和形态特征也有较为严格的要求,不适用于已经失去原始形态的目标样本,如熊胆、熊胆粉、熊肉罐头等产品的鉴定。
针对熊科动物及其制品,既往实验室常通过理化方法进行鉴定,包括显微观察法[4]、荧光观察法[5-7]、薄层色谱法[7-8]、红外光谱法、高效和超高效液相色谱法[9-10]、氨基酸和微量元素测定法等[11-12]。上述方法的应用范围均围绕熊胆或熊胆粉,局限性较大,判定过程依赖于鉴定人员的经验、观感,主观性较强,判定结果的准确性受目标样本的均一性影响较大,难以标准化。随着PCR技术的推广和普及,基于PCR技术原理的各种分子生物学检测鉴定方法已逐渐成为分子鉴定方面的重要技术支撑。田娜等[13]在12S ribosomal RNA(12S rRNA)基因段和16S ribosomal RNA(16S rRNA)基因段设计特异性引物,根据猪、熊、鱼等9个物种的短串联重复序列(Short Tandem Repeat,STR)指纹图谱差异进行物种鉴定[13]。何永胜等[14]采用实时荧光PCR法以黑熊和棕熊为目标对象在线粒体细胞色素c氧化酶1(cytochrome c oxidase subunit 1,COX1)基因设计特异性引物和探针,并对方法的灵敏度和特异性进行检验。赵远等[15]采用常规PCR法,以猪、牛和熊为目标对象分别设计3套特异性引物,并通过测序对检测结果进行验证。上述研究的主要目的主要在于熊胆粉的鉴定,且特异性引物、探针序列基于黑熊、棕熊的相关基因序列,而无法明确其对熊科其他物种的适用性。
对于DNA条形码鉴定技术,选择恰当的目标基因片段是鉴定方法适用性和鉴定结果准确性的主要前提之一。相较于核基因,线粒体基因为母系遗传,在所有需氧生物中普遍存在,其基因组通常为环状的双链闭合环状DNA分子,结构紧凑,没有内含子,在进化过程中趋向于简化,有助于提高基因表达的效率和准确性,且线粒体通过二分裂而非有性重组的方式进行繁殖,不易受到插入、缺失、重排等基因变异事件的影响,常被用于系统发育和物种鉴定的相关研究。Arida等[16]基于线粒体COX1基因建立DNA条形码方法在印度尼西亚西巴布亚省进行脊椎动物种群调查,最终获得264种分子分类单元,但其中75种无法直接辨识,不同类群中能够区分到种的比例各不相同,鱼类为71%、鸟类为100%、哺乳类为77%、两栖类为65%、爬行类为48%,无法排除由于条形码间隙不显著以及最大种内遗传距离和最小种间遗传距离差异不显著导致难以明确区分物种的可能,提示通用性较高的方法可能存在特异性较差的缺点,对于某些科、属内的分化时间较短、基因序列差异较小的动物往往难以明确区分到种。
长期以来,COX1基因也被广泛用作昆虫、鱼类、鸟类、蛇类、猫科动物等纲、目、科级别种群的条形码标记[17-21],许亚春等[22]同样通过DNA条形码技术,基于COX1基因鉴定熊胆粉及其混伪品,对收集的熊胆粉样品进行 DNA 提取、PCR 扩增、测序和遗传距离分析,结果显示黑熊与棕熊在NJ进化树聚为一支,均可明显区分于伪品及混伪品,但该研究的熊科动物目标样本只涉及黑熊胆粉,也没有进一步验证该方法对其他6种熊科动物的适用性。此外,Meyer等[23]、Huang等[24]指出基于COX1基因的鉴定方法对于识别某些近缘物种可能存在挑战。反之,相较于核基因或线粒体基因的其他基因段,COX2基因进化迅速、饱和度高,更适合用于区分近缘物种[25]。Ling等[26]在一项针对大熊猫的研究中比较了COX2基因序列在熊科物种间的差异,指出在进化过程中大熊猫的COX2基因具有高度保守性,同源序列进化分析结果也表明,COX2基因在该群体中具有不同的进化模式。Talbot等[27]对熊科动物线粒体细胞色素c氧化酶1(cytochrome c oxidase subunit 1,COX1)基因序列进行比对分析,发现大熊猫和眼镜熊在进化树上的位置较为原始,是最早分化的两个分支,其余熊科物种则在上新世中期至更新世早期经历快速进化,在相对较短的地质时间内显著多样化。Yu等[28]针对熊科线粒体基因组的研究也印证了熊科物种分化历程短而快的特点。上述研究均提示熊科物种间线粒体基因序列的一致性可能较高,不易于从基因层面区分种间差异。
3 结语
本研究结合特异性PCR与DNA条形码技术,在熊科线粒体的高度保守区域——COX2基因中有望区分8个物种的序列片段设计上下游引物,建立了高特异性、高灵敏度的鉴定方法,能够满足微量、痕量分子生物学检测鉴定的要求,对打击偷猎、走私、掺假等违法犯罪行为具有重要意义。研究过程中测定的亚洲黑熊(NCBI登录号PQ182686.1)和大熊猫(NCBI登录号PP871704.1)线粒体全基因序列进一步丰富了公共基因数据库,为后续相关研究增添基础材料。根据鉴定结果构建的NJ进化树亦呈现出与既往同类研究相符的进化分支关系[27],形成相互印证。然而,通过DNA条形码进行物种鉴定的可行性对基因数据库,尤其是NCBI和生命条形码数据系统(Barcode of Life Data Systems,BOLD)中高质量参考序列的可及性和适用性依赖程度高[29],后续工作中有必要积极完善数据库资料,提供更多高质量的物种DNA序列,与全球相关领域工作者共享基础数据,共同促进行业发展。此外,由于来源稀缺,本实验室暂未获得马来熊、懒熊、眼镜熊和美洲黑熊的组织样本,虽已通过上述4种熊科动物的目标序列合成质粒完成验证流程,后续工作中仍需积极完善相关组织样本的鉴定结果,以进一步确立所建立方法的适用性。
参考文献
[1]扈觐玺, 李亚金, 王淳, 等. 熊胆的药用历史及现代研究进展的知识图谱分析[J]. 中药材, 2024(7): 1862-1870.
[2] Hebert P D N, Ratnasingham S, De Waard J R. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proceedings of the Royal Society of London. Series B: Biological Sciences, 2003, 270(suppl_1): S96-S99.
[3] Trail P W. Morphological species identification of wildlife forensic evidence based on digital images[J]. Forensic Science International: Animals and Environments, 2021, 1: 100021.
[4]张丽华, 苑广信. 熊胆的显微指纹特征研究[J]. 时珍国医国药, 2008(7): 1679-1680.
[5]杨令家, 邓勇, 谷宏伟, 等. 熊胆, 狗胆的薄层色谱鉴别[J]. 时珍国药研究, 1997, 8(3): 239-239.
[6]谭文敏. 兔胆粉和熊胆粉成分分析及药理作用比较研究[D]. 长春: 吉林农业大学, 2015.
[7]李炎桂, 臧埔, 曲正义, 等. 貉胆生药学鉴定研究[J]. 特产研究, 2020, 42(1): 25-28.
[8]何柯, 张荣, 晁若冰. 熊胆川贝枇杷膏中药材的薄层色谱鉴别和牛磺熊去氧胆酸及岩白菜素的测定[J]. 华西药学杂志, 2016, 31(6): 650-653.
[9]张怡欣, 孙帅, 程绒绒, 等. 基于超高效液相色谱串联电喷雾式检测器新技术的熊胆粉特征指纹图谱研究[J]. 上海中医药大学学报, 2020, 34(3): 83-90.
[10]简龙海, 闻宏亮, 毛秀红, 等. 痰热清注射液中熊胆粉部分指纹图谱和成分测定[J]. 中成药, 2013, 35(1): 109-113.
[11]王杰, 熊爱珍, 程绒绒, 等. 不同来源引流熊胆粉的化学成分系统分析[J]. 中国中药杂志, 2018(11): 2326-2332.
[12]李菲. 基于物质成分相似性的体外培育拟天然熊胆粉的基质选择研究[D]. 重庆: 重庆大学, 2015.
[13]田娜, 袁媛, 金艳, 等. 胆粉(汁)类药材的DNA指纹图谱鉴别方法研究[J]. 中国中药杂志, 2020, 45(5): 1064-1069.
[14]何永盛, 连晓聪, 高利海, 等. 基于荧光PCR技术的熊胆粉真伪鉴别研究[J]. 中药材, 2018, 41(11): 2534-2536.
[15]赵远, 贾慧建, 宋顺佳, 等. 真伪熊胆粉物种基源分子生物学鉴定方法的建立及评价[J]. 吉林大学学报 (医学版), 2022, 48(3): 801-808.
[16] Arida, Evy, Ashari, et al. Exploring the vertebrate fauna of the Bird’s Head Peninsula (Indonesia, West Papua) through DNA barcodes[J]. Molecular Ecology Resources, 2021, 21(7): 2369-2387.
[17]蔡延森, 李佳凌, 赵矫, 等. 猫科动物DNA条形码及线粒体假基因对物种鉴定的影响[J]. 四川动物, 2017, 36(4): 425-430.
[18]宋南, 刘杰, 彩万志, 等. DNA条形码在昆虫分类中的应用[J]. 四川动物, 2013, 32(3): 470-474.
[19]苏海英, 王湘君, 陈文, 等. 蛇类DNA条形码研究进展[J]. 内江科技, 2018, 39(11): 81-83.
[20]郑晓聪, 朱崧琪, 徐浩, 等. 应用DNA条形码技术对深圳市售花胶鱼种鉴定与分析[J]. 食品安全质量检测学报, 2022, 13(3): 963-971.
[21]马明义, 闫颖, 王译伟, 等. 我国32种鸟类DNA条形码分析[J]. 四川动物, 2012, 31(5): 729-733.
[22]许亚春, 熊超, 姜春丽, 等. DNA 条形码技术在动物类药材熊胆粉及其混伪品鉴定中的应用[J]. 中国中药杂志, 2018, 43(4): 645-650.
[23] Meyer, Christopher P, Paulay, et al. DNA barcoding: error rates based on comprehensive sampling[J]. Plos Biology, 2005, 3(12): e422.
[24] Huang, Danwei, Meier, et al. Slow mitochondrial COⅠsequence evolution at the base of the metazoan tree and its implications for DNA barcoding[J]. Journal of Molecular Evolution, 2008, 66: 167-174.
[25] Mueller, Rachel Lockridge. Evolutionary rates, divergence dates, and the performance of mitochondrial genes in Bayesian phylogenetic analysis[J]. Systematic Biology, 2006, 55(2): 289-300.
[26] Ling, S S, Zhu, et al. Analysis of the cytochrome c oxidase subunit II (COX2) gene in giant panda, Ailuropoda melanoleuca[J]. Genetics and molecular research, 2017, 16(1): 1-9.
[27] Talbot, Sandra L, Shields, et al. A phylogeny of the bears (Ursidae) inferred from complete sequences of three mitochondrial genes[J]. Molecular Phylogenetics and Evolution, 1996, 5(3): 567-575.
[28] Yu, Li, Li, et al. Analysis of complete mitochondrial genome sequences increases phylogenetic resolution of bears (Ursidae), a mammalian family that experienced rapid speciation[J]. BMC Evolutionary Biology, 2007, 7: 1-11.
[29] Antil, Sandeep, Abraham, et al. DNA barcoding, an effective tool for species identification: a review[J]. Molecular Biology Reports, 2023, 50(1): 761-775.
表1 熊科动物组织样本9份
Table 1 Nine tissue samples of Ursidae
样本编号 | 动物组织 |
1 | 胆 |
2 | 胆 |
3 | 爪 |
4 | 胆 |
5 | 肉 |
6 | 爪 |
7 | 毛 |
8 | 毛 |
9 | 肝 |
表2 陆生和水生动物组织样本16份
Table 2 Sixteen tissue samples of terrestrial and aquatic animals
序号 | 动物组织 | 物种 | 学名 |
1 | 皮 | 网纹蟒 | Malayopython reticulatus |
2 | 皮 | 赤狐 | Vulpes vulpes |
3 | 皮 | 欧洲水貂 | Mustela lutreola |
4 | 毛 | 亚洲狮 | Panthera leo persica |
5 | 毛 | 东北虎 | Panthera tigris altaica |
6 | 角 | 高鼻羚羊 | Saiga tatarica |
7 | 肉 | 家猫 | Felis catus Linnaeus |
8 | 肉 | 家鼠 | Mus musculus |
9 | 肉 | 家猪 | Sus scrofa domestica |
10 | 肉 | 牦牛 | Bos mutus |
11 | 肉 | 水牛 | Bubalus bubalis |
12 | 肉 | 山羊 | Capra hircus |
13 | 肉 | 绵羊 | Ovis aries |
14 | 肉 | 原鸡 | Gallus gallus domesticus |
15 | 肉 | 番鸭 | Cairina moschata |
16 | 肉 | 鲩鱼 | Ctenopharyngodon idella |
表3 熊科动物合成质粒4份
Table 3 Four synthetic plasmids of Ursidae
合成质粒序号 | 物种 | 学名 | NCBI登录号 |
1 | 马来熊 | Helarctos malayanus | MN807949.1 |
2 | 懒熊 | Melursus ursinus | NC_009970.1 |
3 | 眼镜熊 | Tremarctos ornatus | NC_009969.1 |
4 | 美洲黑熊 | Ursus americanus | NC_003426.1 |
表7 基于Kimura两参数模型的基因序列遗传距离
Table 7 Genetic distances of gene sequences based on the Kimura two-parameter model
序号 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | 16 | 17 | 18 | 19 | 20 | 21 | 22 | 23 | 24 | 25 |
1 | |||||||||||||||||||||||||
2 | 0.012 | ||||||||||||||||||||||||
3 | 0.014 | 0.012 | |||||||||||||||||||||||
4 | 0.003 | 0.009 | 0.010 | ||||||||||||||||||||||
5 | 0.009 | 0.003 | 0.009 | 0.005 | |||||||||||||||||||||
6 | 0.123 | 0.109 | 0.121 | 0.120 | 0.114 | ||||||||||||||||||||
7 | 0.123 | 0.109 | 0.121 | 0.120 | 0.114 | 0.000 | |||||||||||||||||||
8 | 0.132 | 0.118 | 0.130 | 0.129 | 0.122 | 0.024 | 0.024 | ||||||||||||||||||
9 | 0.182 | 0.174 | 0.178 | 0.177 | 0.174 | 0.208 | 0.208 | 0.210 | |||||||||||||||||
10 | 0.073 | 0.069 | 0.071 | 0.071 | 0.069 | 0.101 | 0.101 | 0.106 | 0.182 | ||||||||||||||||
11 | 0.075 | 0.071 | 0.073 | 0.073 | 0.071 | 0.099 | 0.099 | 0.103 | 0.185 | 0.002 | |||||||||||||||
12 | 0.102 | 0.097 | 0.100 | 0.099 | 0.097 | 0.114 | 0.114 | 0.112 | 0.177 | 0.081 | 0.079 | ||||||||||||||
13 | 0.104 | 0.099 | 0.102 | 0.101 | 0.099 | 0.116 | 0.116 | 0.114 | 0.180 | 0.075 | 0.073 | 0.005 | |||||||||||||
14 | 0.014 | 0.002 | 0.014 | 0.010 | 0.005 | 0.107 | 0.107 | 0.116 | 0.174 | 0.071 | 0.073 | 0.095 | 0.097 | ||||||||||||
15 | 0.009 | 0.003 | 0.009 | 0.005 | 0.000 | 0.114 | 0.114 | 0.122 | 0.174 | 0.069 | 0.071 | 0.097 | 0.099 | 0.005 | |||||||||||
16 | 0.123 | 0.109 | 0.121 | 0.120 | 0.114 | 0.000 | 0.000 | 0.024 | 0.208 | 0.101 | 0.099 | 0.114 | 0.116 | 0.107 | 0.114 | ||||||||||
17 | 0.123 | 0.109 | 0.121 | 0.120 | 0.114 | 0.000 | 0.000 | 0.024 | 0.208 | 0.101 | 0.099 | 0.114 | 0.116 | 0.107 | 0.114 | 0 | |||||||||
18 | 0.129 | 0.116 | 0.127 | 0.127 | 0.120 | 0.022 | 0.022 | 0.005 | 0.207 | 0.103 | 0.101 | 0.114 | 0.116 | 0.113 | 0.120 | 0.022 | 0.022 | ||||||||
19 | 0.129 | 0.116 | 0.127 | 0.127 | 0.120 | 0.023 | 0.023 | 0.005 | 0.207 | 0.103 | 0.101 | 0.114 | 0.116 | 0.113 | 0.120 | 0.023 | 0.023 | 0.003 | |||||||
20 | 0.182 | 0.174 | 0.178 | 0.177 | 0.174 | 0.208 | 0.208 | 0.210 | 0.000 | 0.182 | 0.185 | 0.177 | 0.180 | 0.174 | 0.174 | 0.208 | 0.208 | 0.207 | 0.207 | ||||||
21 | 0.182 | 0.174 | 0.178 | 0.177 | 0.174 | 0.208 | 0.208 | 0.210 | 0.000 | 0.182 | 0.185 | 0.177 | 0.180 | 0.174 | 0.174 | 0.208 | 0.208 | 0.207 | 0.207 | 0 | |||||
22 | 0.112 | 0.103 | 0.110 | 0.110 | 0.107 | 0.103 | 0.103 | 0.111 | 0.182 | 0.095 | 0.093 | 0.091 | 0.093 | 0.101 | 0.107 | 0.103 | 0.103 | 0.109 | 0.109 | 0.182 | 0.182 | ||||
23 | 0.112 | 0.103 | 0.110 | 0.110 | 0.107 | 0.103 | 0.103 | 0.111 | 0.182 | 0.095 | 0.093 | 0.091 | 0.093 | 0.101 | 0.107 | 0.103 | 0.103 | 0.109 | 0.109 | 0.182 | 0.182 | 0 | |||
24 | 0.176 | 0.170 | 0.174 | 0.173 | 0.170 | 0.170 | 0.170 | 0.180 | 0.200 | 0.147 | 0.149 | 0.178 | 0.175 | 0.172 | 0.170 | 0.170 | 0.170 | 0.177 | 0.177 | 0.200 | 0.200 | 0.192 | 0.192 | ||
25 | 0.176 | 0.170 | 0.174 | 0.173 | 0.170 | 0.170 | 0.170 | 0.180 | 0.200 | 0.147 | 0.149 | 0.178 | 0.175 | 0.172 | 0.170 | 0.170 | 0.170 | 0.177 | 0.177 | 0.200 | 0.200 | 0.192 | 0.192 | 0 |
注: 1.亚洲黑熊(熊胆); 2.亚洲黑熊(熊胆); 3.亚洲黑熊(熊胆); 4.亚洲黑熊(熊爪); 5.亚洲黑熊(熊肉); 6.棕熊(熊爪); 7.棕熊(熊毛); 8.北极熊(熊毛); 9.大熊猫(熊肝); 10.美洲黑熊(OQ318937); 11.美洲黑熊(OQ318942); 12.马来熊(OQ564480); 13.马来熊(FM177765); 14.亚洲黑熊(NC_011118); 15.亚洲黑熊(PQ182686); 16.棕熊(OK512947); 17.棕熊(OK512925); 18.北极熊(JX196383); 19.北极熊(OK001275); 20.大熊猫(PP871704); 21.大熊猫(EF196663); 22.懒熊(MH931229); 23.懒熊(MH910095); 24.眼镜熊(OR999077); 25.眼镜熊(NC_009969)
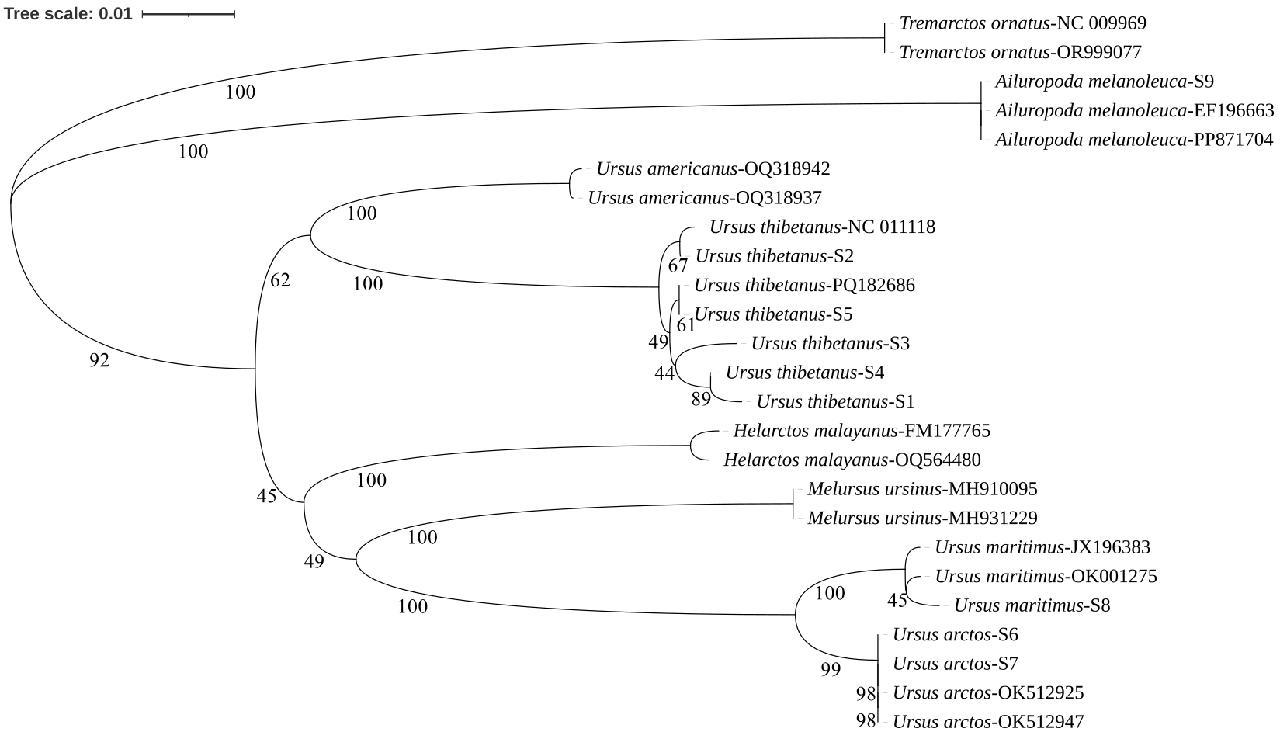
图3 基于COX2基因序列构建的熊科系统发育树
Fig.3 Phylogenetic tree of Ursidae constructed based on the COX2 gene sequence
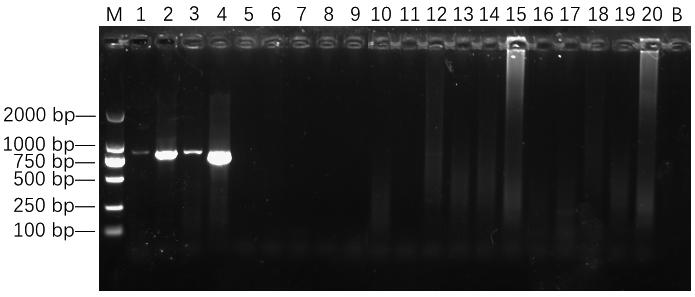
样本编号: 1—4; 序号: 5—20; B: 空白对照
图4 特异性试验电泳结果
Fig.4 Electrophoresis results of specificity test
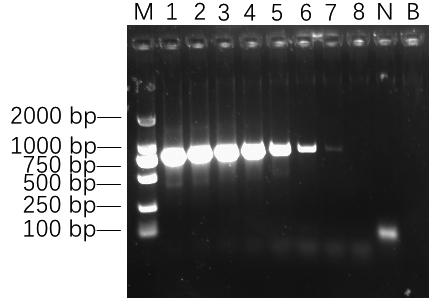
浓度为4.30×108~4.30×101拷贝/μL的质粒DNA: 1—8; N; 阴性对照; B: 空白对照
图5 灵敏度试验电泳结果
Fig.5 Electrophoresis results of sensitivity test







