CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
跨境传染病测序平台建设及示范应用
作者:周健 何凤侠 林志伟 张璐 何丽润 刘静宇 戴俊 师永霞
周健 何凤侠 林志伟 张璐 何丽润 刘静宇 戴俊 师永霞
周 健 1 何凤侠 1 林志伟 1 张 璐 1 何丽润 1 刘静宇 1 戴 俊 1 师永霞 1 *
摘 要 为提升海关对新发突发传染病病原体的筛查和及早发现能力,本研究从全球公开基因数据库检索并下载传染病病原体基因组序列及相关信息,形成传染病病原微生物数据库,并在此基础上,结合NGS数据质控分析模块、未知宿主快速鉴定及去除、基于Kmer迭代的短序列拼接、短序列重比对等技术,构建涵盖从数据拼接到报告生成的宏基因组/宏转录组测序分析平台。使用测序分析平台对阳性临床样本进行测序分析,并与第三方分析平台进行相互验证,以评价建立的跨境传染病测序平台分析检测能力。本研究构建的数据库共涵盖8509种病原体,包括细菌4863种、病毒2501种、原虫100种、真菌1045种,涉及呼吸道传染病、消化道传染病、虫媒传染病等不同症候群的病原体。病原体鉴定和丰度统计结果显示,检测准确性和丰度分布符合病原体筛查与诊断需求。与第三方数据分析平台比较结果显示,在属水平和种水平上,两种平台的病原体数量差异均处于可接受范围内,且在部分病原体检测方面,灵敏度更高。本研究开发的跨境传染病测序平台数据库基本涵盖目前常见传染病病原体,覆盖范围广,检测灵敏度和准确率符合临床要求,能够应用于口岸入境人群样本检测,提升口岸新发突发传染病病原体筛查能力。
关键词 传染病;测序平台;数据库
Construction and Demonstration Application of Cross- border Infectious Disease Sequencing Platform
ZHOU Jian1 HE Feng-Xia1 LIN Zhi-Wei1 ZHANG Lu1
HE Li-Run1 LIU Jing-Yu1 DAI Jun1 SHI Yong-Xia1*
Abstract To enhance the screening and early detection capabilities of customs authorities for emerging infectious disease pathogens, this study established a pathogen database and genomic sequencing platform for port-focused pathogens through integrating gene sequencing and bioinformatics technologies. The pathogen database was constructed by retrieving and downloading genomic sequences and related information of infectious pathogens from global public genetic databases. This platform was further developed by incorporating an analytical pipeline that included next-generation sequencing (NGS) data quality control modules, rapid identification/removal of unknown host sequences, K-mer iteration-based short-read assembly, and short-sequence realignment technologies, thereby building a comprehensive metagenomic and metatranscriptomic sequencing analysis platform spanning from data assembly to automated report generation. The platform’s analytical performance was evaluated by sequencing positive clinical samples and conducting comparative validation with third-party analysis platforms. The final database encompassed 8,509 pathogens, including 4,863 bacterial species, 2,501 viral strains, 100 protozoan species, and 1,045 fungal species, covering pathogens associated with respiratory, gastrointestinal, vector-borne, and other syndromic diseases. Pathogen identification and abundance quantification demonstrated that the detection accuracy and taxonomic distribution met the requirements for clinical screening and diagnostic applications. Comparative analysis revealed that the differences in pathogen detection counts at both genus and species levels between the two platforms were within acceptable thresholds, while our platform exhibited higher sensitivity for specific pathogens. The developed cross-border pathogen surveillance database basically covers prevalent infectious pathogens with broad taxonomic coverage. The detection sensitivity and accuracy meet clinical standards, enabling its application in port entry population screening to strengthen the capacity for detecting emerging infectious disease pathogens.
Keywords infectious diseases; sequencing platform; database
基金项目:海关总署科研项目(2023HK010);广州市科技局项目(2023A04J1279)
第一作者:周健(1995—),男,汉族,湖南衡阳县人,主管技师,硕士,主要从事卫生检疫工作,E-mail: zhouj@iqtcnet.cn
通信作者:师永霞(1976—),女,汉族,河南安阳县人,主任技师,博士,主要从事卫生检疫工作,E-mail: shiyx@iqtcnet.cn
1. 广州海关技术中心 广州 510700
2. Guangzhou Customs Technology Center, Guangzhou 510700
下一代测序技术(Next-generation sequencing technology,NGS)又叫高通量测序技术,或者大规模平行测序技术,是伴随着人类基因组计划发展而来的新兴测序技术,这项技术使得同时独立对数百万DNA片段进行测序成为可能[1]。与一代测序技术相比,该技术在降低测序成本的同时提升了测序基因组数量和多样性[2]。因此,基于宏基因组学的NGS技术可以通过一次测序获得样本中物种基因信息,从而能够识别样本中存在的病原微生物,极大地减少逐个排除可疑病原所耗费的时间及人力物力。同时,与传统病原体检测方式相比,NGS技术不需要对病原体进行分离培养,可以直接对样本核酸进行检测且具有高灵敏性,极大地提升难以培养甚至无法培养的病原体以及稀有或低丰度病原体的检测能力,同时降低检测时间及操作的复杂性,提高检测效率[3]。目前,NGS技术在公共卫生监测、病原体检测以及传染病溯源等方面得到了广泛应用。
在全球化、城镇化、工业化快速发展的形势下,传染病跨境、跨区传播流行的风险持续加大,对我国公共卫生体系带来挑战。海关作为外防输入的第一道关口,如何在第一时间快速鉴定和发现新发未知病原体是防范传染病跨境传播和蔓延的关键。NGS技术的出现,使得快速、准确地发现未知病原成为可能。现阶段的NGS技术应用平台主要为临床病原体检测及科研应用设计,此类平台数据库多为开源数据库,难以满足海关的安全性要求。同时,该类测序平台主要关注临床常见病原体,无法覆盖口岸关注的重要新发突发及罕见输入性传染病,而且也无法满足口岸检测样本的多样性及复杂性需求。
本研究立足于海关对病原体检测的实际需求,对口岸重点关注的传染病病原体列表进行序列的收录,形成海关自有的NGS数据库,并且部署在“海关总署病原体基因测序平台”测试环境上,避免测序数据泄露风险。同时,开发出针对NGS数据的未知宿主快速鉴定与去除技术,降低后续的短序列组装和微生物鉴定分析的难度,并构建涵盖从数据拼接到报告生成的宏基因组/宏转录组测序分析平台。
1 材料
1.1 数据来源
从世界卫生组织(WHO)、全球共享流感数据倡议组织(GISAID)、NextStrain、美国国家生物技术信息中心(NCBI)和国家微生物科学数据中心等权威机构的公开数据库中收集病原体基因序列。
1.2 测试样本来源
本实验室使用广州口岸入境人员的临床样本进行测序,样本采用荧光PCR检测方法进行核酸检测确认。第三方应用验证样本分别由广州海关、上海海关、青岛海关和南宁海关所属国际旅行卫生保健中心采集并测序,测试样本使用符合相关规定。
2 方法
2.1 构建传染病数据库
根据口岸重点关注传染病和国家微生物科学数据中心开发的全球病原微生物名录,获取病原体的列表(https://nmdc.cn/gcpathogen/)。根据该名录中物种名(拉丁文)、Taxonomy ID等表征该病原体的信息,运用Taxonkit工具在NCBI的Taxonomy库里面搜索并下载其对应的基因组序列,收录各病原体的谱系分类信息等。若库中没有收录,则从GISAID、WHO、NextStrain等其他权威数据库中搜索。若遇到有多个基因组的情况,则选RefSeq数据库下载最新且序列长度最长的基因组,再根据特定物种的生物学特征,结合该物种的注释基因集信息,确定序列的最佳分割窗口大小。然后,对各个窗口序列创建散列表,根据散列表建立快速索引。由此,构成一个传染病数据库。
2.2 分析平台界面设计
分析平台搭载在“海关总署病原体基因测序平台”上,采用“一键分析式平台”形式。平台界面主要分为用户界面和管理员界面两部分。用户输入网址,通过海关统一身份认证授权系统授权后,访问Web界面,通过专门的测序数据上传工具进行数据上传,通过点击“项目设计”进行任务命名、分析流程选择,然后一键启动选择的分析流程任务,同时也具有对分析流程的终止、删除权限。通过“流程配置”工具可对分析流程进行自定义,在“项目设计”工具实时查询任务进度以及查看或从“检测报告”一栏中下载测序分析报告。管理员则具有维护管理测序平台以及用户授权的权限,同时还可以对用户上传至数据库的测序数据进行审核。
2.3 分析平台流程设计
通过上传测序数据到平台,利用平台集成的FastQC软件对测序数据进行质控,基于Kmer迭代构建的德布鲁因图将质控后的数据组装成contig序列,利用本实验室对于宿主核心基因,例如COⅠ、COⅡ、Cytb等关键线粒体基因的研究构建的宿主物种标签序列库对contig序列进行宿主鉴定及去宿主操作。最后,将去除宿主后的contig序列与数据库进行比对进行病原体物种鉴定,同时依靠数据库中的参考基因对contig序列进行组装,再将组装好的contig和构建的传染病病原体数据库进行比对。通过自动化分析流程可以实现一键式从数据拼接到报告生成的全流程(图1)。
2.4 技术架构设计
分析平台部署在CentOS服务器上,平台安装了Docker引擎、MySQL数据库软件,集成病原体基因数据库以及RINS(Rapid Identification of Non-Human Sequences)病原微生物检测算法,然后通过对分析流程进行自动化设计来完成平台的开发[4]。基于Java进行Web后台系统开发,最后利用 Docker容器技术对分析平台进行封装。同时,依托Spring-cloud微服务架构应用支撑平台。
2.5 测试样本测序
本实验室测序阳性样本共47份,已通过qPCR验证为阳性,采用北京全式金生物公司的核酸提取试剂盒提取样本核酸。使用微远基因公司的宏转录组/宏基因组建库试剂盒进行建库。测序平台为Illumina NextSeq™550,测序方式为单端测序,每份样本分配数据量为20 M reads,每条reads读长为75 bp。测序下机数据经bcl2fastq软件进行转换及拆分。同时,将测序数据上传到本实验室本地化部署的IDseq AS分析平台进行分析,与本研究开发的“跨境传染病测序平台”的分析结果对比。
2.6 第三方示范应用
第三方示范应用样本共132份,分别由广州海关、上海海关、青岛海关和南宁海关所属国际旅行卫生保健中心实验室自行开展测序并将测序数据上传至“跨境传染病测序平台”进行生物信息学分析,并与各实验室现有测序分析平台比较结果一致性。因为第三方示范应用由多个实验室开展,各实验室现有分析平台来源名称不一致,故下文以第三方测序平台统称,具体信息见表1。
3 研究结果
3.1 分析平台的建立
调研全球权威的公共数据库,获得目标物种相关的核酸数据库的出处、数据库结构及相关表型信息,基于Python开发爬虫程序对数据库进行爬取并进行数据清洗。数据库涵盖病原体数目共计8509种,包括细菌4863种、病毒2501种、原虫100种、真菌1045种,部分病原体清单见表2—表5。基于Java分布式计算框架MapReduce对拆分后的测序数据进行预处理,通过分布式计算缩短包括序列分型、序列信息格式化、序列位置确认等预处理时间,然后通过MapReduce对预处理后的数据进行序列特征信息提取,并将提取后的特征信息转化为可比对格式,再通过差异增量的方式将可比对格式文件合并成一个字典文件,用于快速检索及比对。通过对测序样本进行分析,得到结果文件,涵盖了各项主要信息,满足生物信息学分析所必要的数据信息,具体检测指标信息见表6。与本实验室现有的IDseq AS分析平台相比,“跨境传染病测序平台”的分析时间少于30 min,分析速度相对更快、效率更高。
3.2 软件部署及安全测试
数据库与分析流程在“海关总署病原体基因测序平台”部署,并已通过漏洞扫描测试,以保障数据信息安全。
3.3 病原体鉴定与丰度统计
将本实验室测序数据与2.6所述第三方示范应用产生的测序数据以fastq格式上传至“跨境传染病测序平台”进行病原体鉴定和丰度统计,结果显示丰度均高于10%,表明检测准确性和丰度分布满足病原体筛查与诊断需求(表7)。
3.4 平台检测数量对比结果
各实验室将测序数据同步上传至各自现有生物信息学分析平台上进行独立分析,具体分析平台信息见表1。统计各实验室现有第三方测序平台与“跨境传染病测序平台”分析结果,从属水平和种水平上比较两个平台分析检出病原体数量的差异,结果发现两者属水平和种水平上的病原体数量差异在可接受范围内。表8为“跨境传染病测序平台”与各实验室现有第三方测序平台比较,部分测序数据的比对结果。
3.5 关键病原体的检测结果
以相对丰度(Relativate Abundance)等于1为标准,相对丰度大于1则表示能测出对应病原体,关键病原体检测比对结果见表9。本研究发现,与各实验室现有第三方测序平台相比,检测结果大体一致,并且在疟疾和腹泻部分阳性病原体检测上,开发的“跨境传染病测序平台”灵敏度更高。
4 讨论
传统病原微生物检测方法有涂片镜检法、血清免疫法、PCR扩增法以及基因芯片法等,这些方法主要基于“假设-试验-验证”的技术路线,对于重组(人工或自然)、变异的病原检测效果差,无法及时发现新的病原体,可以鉴定的微生物种类有限[5-7]。相比传统病原微生物检测方法,基于NGS的病原微生物检测具有无需预先对标本进行培养、检测速度快、灵敏度高等特点,为输入性传染病的防控和监测增添了新的解决方案[8]。目前,市面上虽然有众多生物信息学分析平台,但大多数平台主要是针对临床诊断或科学研究设计的,无法满足海关测序过程中存在的宿主多样性和样本来源复杂以及数据安全性的需求。
本研究自主研发的平台立足于海关对于口岸测序的实际需求,数据库中收录了口岸重点关注传染病病原体数据,形成海关自有的宏基因组/宏转录组数据库;通过构建宿主物种标签序列库快速地鉴定并最终去除样品中潜在的一个或多个宿主;通过对Docker容器进行合理地分配CPU、内存等计算资源,做到分析全面性与分析效率的平衡,缩短了分析时间;该平台集中部署到“海关总署测序平台”上内网运行,避免了海关测序数据泄露的风险,同时平台源代码通过了海关总署漏洞扫描测试,从源码层面保证了本平台的安全性。通过将“跨境传染病测序平台”在广州海关、南宁海关、青岛海关和上海海关所属国际旅行卫生保健中心进行了示范应用,表明该测序平台支持不同海关之间的信息共享和技术协作,实现了跨区域、跨部门的联防联控,这将大大提升我国整体的传染病病原体监测、预警和快速响应能力。同时,不同关区的示范应用结果表明,本研究开发的“跨境传染病测序平台”与其他第三方测序平台相比,测试结果大体一致,而且在部分病原体检测上准确率更高,能有效检测出样本携带病原体。这得益于本实验室多年来口岸病原体检测领域的经验积累,在平台开发早期针对长期口岸样本检测过程中某些样本类型中病原体载量普遍偏低的情况,对特定病原体检测阈值参数进行了优化调整。该技术的改进有效提升了低丰度病原体的检出灵敏度,使检测性能更加契合口岸卫生检疫场景的实际需求。目前,该测序平台可以覆盖呼吸道传染病、消化道传染病、虫媒传染病等多种病原体的检测,操作简单易上手,实现了从创建项目、选择流程到分析的一键化操作;报告内容较为丰富,包括了种名、属名、相对丰度、特异比对序列数等,有效提升了关口检测能力和检测覆盖面。
综上所述,本研究自主研发平台整体分析速度快,结果准确率高,数据库内容丰富,满足口岸传染病宏基因组测序分析需求,使口岸传染病检测工作更加智能化、精准化、协同化,是推进智慧海关建设的重要技术支撑,具有广泛的推广应用前景。
参考文献
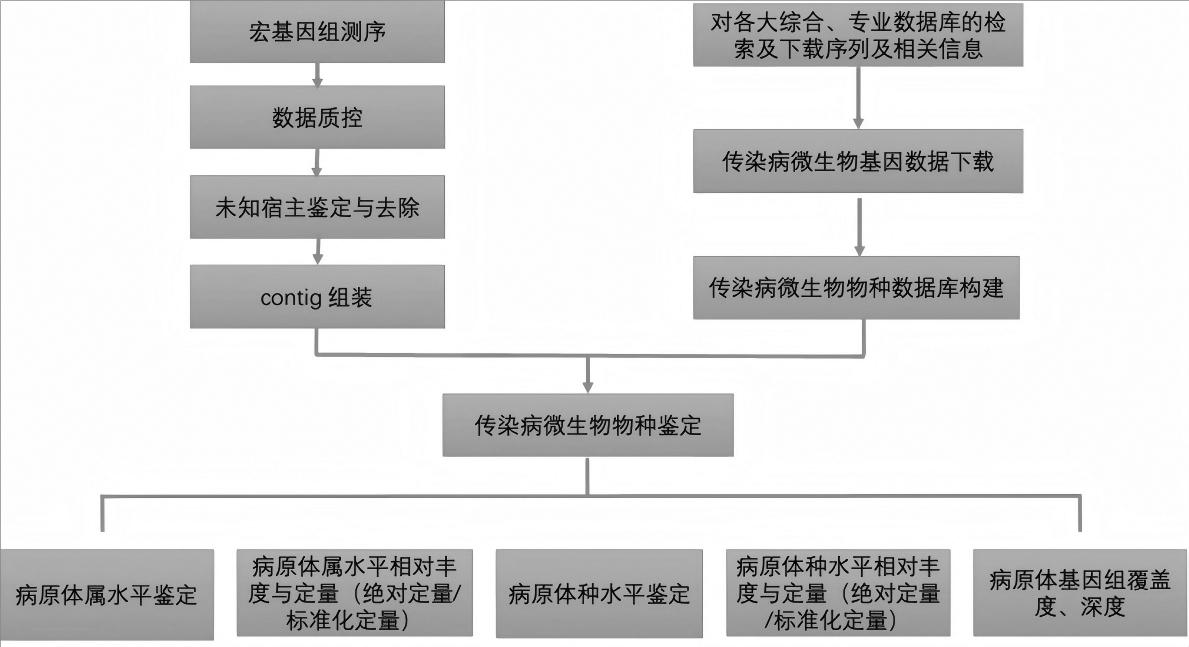
图1 跨境传染病测序平台技术流程图
Fig.1 Technical flowchart of cross-border infectious disease sequencing platform
表1 测序平台信息表
Table 1 Information sheet of sequencing platform
应用实验室 | 测序分析平台 | 测序平台开发公司 |
广州国际旅行卫生保健中心 (广州海关口岸门诊部) | IDseq AS | 微远基因 |
上海国际旅行卫生保健中心 (上海海关口岸门诊部) | EPI2ME Labs | Nanopore |
青岛国际旅行卫生保健中心 (青岛海关口岸门诊部) | 微生物快速识别软件 (PFI), 微生物组装溯源软件 (MGAP) | 华大智造 |
广西国际旅行卫生保健中心 (南宁海关口岸门诊部) | 根据现有国内外文献自行开发的分析流程及数据库 | / |
表2 部分病毒清单
Table 2 Partial list of viruses
病原体名称 | 分类号 | 病原体名称 | 分类号 | 病原体名称 | 分类号 |
人类免疫缺陷病毒 1 型 | 11676 | 口蹄疫病毒 | 12110 | 基孔肯雅病毒 | 37124 |
乙型肝炎病毒 | 10407 | 丁型肝炎病毒 | 12475 | 戊肝病毒 | 1678143 |
诺如病毒 | 11983 | 人肠道病毒 | 1193974 | 猴免疫缺陷病毒 | 11723 |
登革病毒 | 12637 | 拉沙病毒 | 3052310 | 人类疱疹病毒5型 | 10359 |
猴痘病毒 | 10244 | 乙型人腺病毒 | 108098 | 正内罗病毒 | 3052518 |
人类嗜T细胞病毒1型 | 11908 | 东部马脑炎病毒 | 11021 | 日本脑炎病毒 | 11072 |
丙型肝炎病毒 | 3052230 | 人类单纯疱疹病毒1型 | 10298 | 蜱传脑炎病毒 | 11084 |
狂犬病病毒 | 11292 | 乙型鼻病毒 | 147712 | 人类单纯疱疹病毒3型 | 10335 |
西尼罗病毒 | 11082 | 人类单纯疱疹病毒2型 | 10310 | 腮腺炎病毒 | 2560602 |
人类疱疹病毒 4 型 | 10376 | 黄热病毒 | 11089 | 札幌病毒 | 95342 |
禽腮腺炎病毒1型 | 2560319 | 人鼻病毒属 | 169066 |
表3 部分细菌清单
Table 3 Partial list of bacteria
病原体名称 | 分类号 | 病原体名称 | 分类号 | 病原体名称 | 分类号 |
沙门氏菌 | 28901 | 绿脓杆菌 | 287 | 肺炎克雷伯菌 | 573 |
大肠杆菌 | 562 | 屎肠球菌 | 1352 | 大肠弯曲杆菌 | 195 |
金黄色葡萄球菌 | 1280 | 鲍曼不动杆菌 | 470 | 肺炎链球菌 | 1313 |
空肠弯曲菌 | 197 | 艰难梭状芽孢杆菌 | 1496 | 福氏志贺氏菌 | 623 |
李斯特菌 | 1639 | 宋内氏志贺氏菌 | 624 | 副溶血弧菌 | 670 |
表4 部分真菌清单
Table 4 Partial list of fungi
病原体名称 | 分类号 | 病原体名称 | 分类号 | 病原体名称 | 分类号 |
酿酒酵母 | 4932 | 耳念珠菌 | 498019 | 白色念珠菌 | 5476 |
尖孢镰刀菌 | 5507 | 腐皮镰孢 | 169388 | 欧洲光滑念珠菌 | 5478 |
烟曲霉 | 746128 | 禾谷镰孢菌 | 5518 | 疣状瓶霉 | 39412 |
黄曲霉菌 | 5059 | 胶红酵母 | 5537 | 链格孢菌 | 5599 |
新生隐球菌 | 5207 | 木霉属 | 5543 | 黑曲霉菌 | 5061 |
表5 部分原虫清单
Table 5 Partial list of protozoa
病原体名称 | 分类号 | 病原体名称 | 分类号 | 病原体名称 | 分类号 |
恶性疟原虫 | 5833 | 变形藻丝囊霉 | 112090 | 小隐孢子虫 | 5807 |
卡耶塔环孢子虫 | 88456 | 人隐孢子虫 | 237895 | 间日疟原虫 | 5855 |
克氏锥虫 | 5693 | 约氏疟原虫 | 5861 | 隐袭腐霉菌 | 114742 |
弓形虫 | 5811 | 溶组织内阿米巴虫 | 5759 | 威基疟原虫 | 5860 |
肠贾第虫 | 5741 | 杜氏利什曼原虫 | 5661 | 诺氏疟原虫 | 5850 |
表6 检测指标信息表
Table 6 Information table of detection indicators
序号 | 指标名称 | 说明 |
1 | Sample | 样本编号 |
2 | TaxName | 病原体名字 (拉丁文) |
3 | TaxID | 病原体物种ID |
4 | Genome Length (bp) | 病原体基因组大小 (单位bp) |
5 | Count | 检测到的病原体序列数 |
6 | Ave Depth | 对检测到的病原体的平均测序深度 |
7 | Coverage | 对检测到的病原体基因组的覆盖度百分比 |
8 | Relativate Abundance (CPM) | 测序结果中, 每100万条序列含检测到的病原体的序列数是病原体在样品中的标准化后的丰度指标 |
9 | Genus Name | 属名 (英文) |
10 | Genus Relativate Abundance (%) | 属相对丰度 (%) |
11 | Genus specific Count | 属特异性比对序列数 |
12 | Genus specific Abundance | 属特异性比对序列数 (标准化后) |
13 | Species Name | 种名 (英文) |
14 | Species Relativate Abundance (%) | 种相对丰度 (%) |
15 | Species Specific Count | 种特异性比对序列数 |
16 | Species Specific Abundance | 种特异性比对序列数 (标准化后) |
表7 病原体鉴定和丰度统计结果
Table 7 Identification and abundance statistics of pathogens
样品名称 | 病原体数量 (属水平) | 病原体数量 (种水平) | 所有病原体占所测数据含量 (%) |
BS1IQTC741-1-N847S22 | 253 | 772 | 23.80 |
BS2IQTC742-1-N848S23 | 257 | 783 | 19.50 |
BS3IQTC743-1-N849S24 | 243 | 744 | 29.17 |
BS4IQTC744-1-N850S25 | 268 | 831 | 38.66 |
Cao-BS-IQTC0055-1-T030R | 197 | 613 | 93.80 |
Cao-TS-IQTC0056-1-T032R | 223 | 629 | 47.91 |
Cui-BS-IQTC0054-1-T029R | 238 | 714 | 40.79 |
IQTC0061-1-T038D | 208 | 537 | 35.13 |
IQTC0062-1-T039D | 230 | 600 | 31.24 |
IQTC0063-1-T041D | 218 | 582 | 18.97 |
IQTC0064-1-T042D | 267 | 764 | 31.63 |
IQTC0065-1-T043D | 228 | 588 | 33.03 |
IQTC0096-1-T010R | 223 | 678 | 77.04 |
IQTC0097-1-T011R | 250 | 751 | 56.91 |
IQTC0098-1-T012R | 221 | 685 | 66.18 |
IQTC0099-1-T013R | 226 | 695 | 58.77 |
IQTC0100-1-T014R | 228 | 712 | 69.45 |
IQTC0101-1-T015R | 212 | 656 | 71.84 |
IQTC0102-1-T016R | 225 | 690 | 84.48 |
IQTC0103-1-T017R | 229 | 682 | 55.43 |
IQTC0104-1-T018R | 228 | 716 | 78.88 |
IQTC0105-1-T019R | 226 | 697 | 64.30 |
IQTC0106-1-T020R | 176 | 409 | 55.14 |
IQTC0108-1-T022R | 209 | 572 | 48.49 |
IQTC0109-1-T023R | 229 | 635 | 12.66 |
IQTC0110-1-T024R | 231 | 697 | 26.06 |
IQTC0111-1-T025R | 228 | 632 | 19.81 |
IQTC0112-1-T026R | 173 | 473 | 56.56 |
IQTC0113-1-T027R | 197 | 495 | 12.16 |
IQTC0114-1-T028R | 189 | 505 | 54.57 |
IQTC0115-1-T029R | 202 | 535 | 14.17 |
IQTC0270-1-T033R | 221 | 684 | 67.53 |
IQTC0271-1-T034R | 229 | 707 | 59.37 |
IQTC0282-1-T048D | 223 | 661 | 78.55 |
IQTC0283-1-T092D | 188 | 505 | 16.50 |
IQTC151 | 162 | 495 | 21.35 |
IQTC152 | 180 | 543 | 52.39 |
IQTC153 | 182 | 564 | 16.17 |
IQTC154 | 193 | 562 | 13.39 |
IQTC155 | 183 | 560 | 16.10 |
IQTC156 | 176 | 524 | 22.16 |
JieBangGuaShuai-TGS-3 | 126 | 274 | 34.54 |
SRR10971381 | 251 | 753 | 75.23 |
IQTC0243-1-T079D | 151 | 363 | 15.89 |
IQTC0243-1-T038D | 119 | 236 | 45.39 |
IQTC0283-1-T092D | 188 | 505 | 16.50 |
表7(续)
表8 平台检测数量对比结果表
Table 8 Comparative analysis of platform detection numbers
样品名称 | 跨境传染病测序平台 | 第三方测序平台 | |||
病原体数量 (属水平) | 病原体数量 (种水平) | 病原体数量 (属水平) | 病原体数量 (种水平) | ||
IQTC0061-1-T038D | 208 | 537 | 188 | 472 | |
IQTC0062-1-T039D | 230 | 600 | 188 | 553 | |
IQTC0063-1-T041D | 218 | 582 | 224 | 541 | |
IQTC0064-1-T042D | 267 | 764 | 241 | 630 | |
IQTC0065-1-T043D | 228 | 588 | 169 | 426 | |
IQTC0096-1-T010R | 223 | 678 | 531 | 1407 | |
IQTC0097-1-T011R | 250 | 751 | 647 | 1546 | |
IQTC0098-1-T012R | 221 | 685 | 564 | 1512 | |
IQTC0099-1-T013R | 226 | 695 | 546 | 1422 | |
IQTC0100-1-T014R | 228 | 712 | 596 | 1438 | |
IQTC0101-1-T015R | 212 | 656 | 657 | 1619 | |
IQTC0102-1-T016R | 225 | 690 | 624 | 1459 | |
IQTC0103-1-T017R | 229 | 682 | 530 | 1383 | |
IQTC0104-1-T018R | 228 | 716 | 595 | 1582 | |
IQTC0105-1-T019R | 226 | 697 | 644 | 1531 | |
IQTC0106-1-T020R | 176 | 409 | 184 | 445 | |
IQTC0107-1-T021R | 242 | 700 | 358 | 901 | |
IQTC0108-1-T022R | 209 | 572 | 331 | 857 | |
IQTC0109-1-T023R | 229 | 635 | 264 | 738 | |
IQTC0110-1-T024R | 231 | 697 | 478 | 1253 | |
IQTC0111-1-T025R | 228 | 632 | 366 | 921 | |
IQTC0112-1-T026R | 173 | 473 | 253 | 645 | |
IQTC0113-1-T027R | 197 | 495 | 251 | 656 | |
IQTC0114-1-T028R | 189 | 505 | 232 | 619 | |
IQTC0115-1-T029R | 202 | 535 | 266 | 671 | |
IQTC0270-1-T033R | 221 | 684 | 585 | 1624 | |
IQTC0271-1-T034R | 229 | 707 | 591 | 1609 | |
IQTC0282-1-T048D | 223 | 661 | 551 | 1593 | |
IQTC0283-1-T092D | 188 | 505 | 438 | 1410 | |
表9 平台应用测试报告
Table 9 Test report of platform application
应用实验室 | 样本类型 | 样本量 | 检测结果覆盖率 (%) | |||
小计 | 总计 | 平台结果 | 第三方结果 | |||
广州海关技术中心 | 甲流阳性样本 | 6 | 47 | 100 | 100 | |
乙流阳性样本 | 6 | 83.3 | 83.3 | |||
登革热阳性样本 | 9 | 100 | 100 | |||
疟疾阳性样 | 2 | 100 | 90.9 | |||
猴痘阳性样本 | 1 | 100 | 100 | |||
冠状病毒阳性样本 | 3 | 100 | 100 | |||
水痘阳性样本 | 1 | 100 | 100 | |||
其他 | 19 | 100 | 100 | |||
(广州海关口岸门诊部) | 艾滋病毒阳性样本 | 10 | 29 | 100 | 10 | |
结核菌培养物 | 10 | 100 | 100 | |||
疟疾阳性样本 | 9 | 100 | 55.5 | |||
(上海海关口岸门诊部) | 腹泻样本 (病毒阳性) | 8 | 30 | 92.3 | 38.5 | |
腹泻样本 (细菌阳性) | 5 | 60 | 60 | |||
腹泻样本 (荧光PCR阴性) | 17 | 4份阴性, 其他检出 | 3份阴性, 其他检出 | |||
(青岛海关口岸门诊部) | 新型冠状病毒核酸阳性样本 | 8 | 22 | 100 | 100 | |
未知病原样本 | 5 | 100 | 100 | |||
SRA数据库下载数据 | 9 | 100 | 100 | |||
(南宁海关口岸门诊部) | 口岸入境有症状人员样本 | 51 | 51 | 100 | 100 | |
合计 | 179 | |||||







