CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
岩牡丹属植物DNA条形码研究
作者:水克娟 闫邦奇 李碧霞 曹野 李尚霖 黎财慧 张卫东
水克娟 闫邦奇 李碧霞 曹野 李尚霖 黎财慧 张卫东
水克娟 1 闫邦奇 1 李碧霞 1 曹 野 1 李尚霖 2 黎财慧 1 张卫东 1 *
摘 要 本研究利用DNA条形码技术对岩牡丹属(Ariocarpus)植物进行物种鉴定,共收集了42份岩牡丹属样品,涵盖6个物种(勃氏牡丹、黑牡丹、龙角牡丹、龟甲牡丹、龙舌兰牡丹和岩牡丹),并选取7个基因片段(ITS、PEPC、psbA-trnH、rbcL、matK、trnS-trnG和atpH-atpI)设计特异性引物进行PCR扩增和测序拼接。结果显示,matK、trnS-trnG和atpH-atpI 片段扩增效率及测序成功率均为100%,通用性良好。经过进一步单倍型分析显示,单一基因片段(如matK、trnS-trnG或atpH-atpI)仅能实现部分物种鉴别,而多片段组合策略(trnS-trnG+matK和atpH-atpI+matK)可准确区分全部6个物种。此外,利用MEGA 11软件分析种内/种间遗传距离并构建邻接(Neighbor-Joining,NJ)系统发育树,结果显示岩牡丹属种间存在显著遗传分化,但种内变异差异不显著;单个DNA片段对岩牡丹属物种的鉴别率较低,而2种片段组合条形码(trnS-trnG matK和atpH-atpI+matK)可将岩牡丹属6个物种完全区分开,分辨率达100%。研究结果表明,多片段组合的DNA条形码技术可为岩牡丹属物种鉴定、濒危保护及分类学研究提供分子依据,其中,trnS-trnG+matK和atpH-atpI+matK组合序列可作为岩牡丹属的DNA条形码。
关键词 DNA条形码;岩牡丹属;物种鉴定;遗传距离;系统发育分析
� Application of DNA Barcoding for Species Identification in Ariocarpus
SHUI Ke-Juan1 YAN Bang-Qi1 LI Bi-Xia1 CAO Ye1
LI Shang-Lin2 LI Cai-Hui1 ZHANG Wei-Dong1*
Abstract This study aimed to utilize DNA barcoding technology for species identification within the genus Ariocarpus, providing molecular evidence for endangered species conservation and taxonomic research. We collected 42 Ariocarpus samples from six species: A. bravoanus, A. kotschoubeyanus, A. scapharostrus, A. fissuratus, A. agavoides, and A. retusus. Seven gene regions (ITS, PEPC, psbA-trnH, rbcL, matK, trnS-trnG, and atpH-atpI) were selected for primer design, PCR amplification, and sequencing. Our results demonstrated that matK, trnS-trnG, and atpH-atpI fragments exhibited 100% amplification efficiency and sequencing success rate, demonstrating high universality. Haplotype analysis revealed that single-gene barcodes (e.g., matK, trnS-trnG, or atpH-atpI) could only partially distinguish species, whereas multi-locus combinations (trnS-trnG+matK and atpH-atpI+matK) achieved 100% species resolution. Furthermore, genetic distances (intraspecific vs. interspecific) were calculated using MEGA11, and neighbor-joining (NJ) phylogenetic trees were constructed. Significant interspecific divergence was observed, while intraspecific variation was minimal. Individual DNA fragments showed limited species discrimination. However, two-locus barcodes (trnS-trnG+matK and atpH-atpI+matK) achieved 100% accuracy in distinguishing all six Ariocarpus species. In summary, this study confirms that multi-fragment DNA barcoding effectively resolves species identification challenges in Ariocarpus, providing essential molecular data for species authentication, conservation prioritization, and taxonomic clarification. The combined sequences trnS-trnG+matK and atpH-atpI+matK can be recommended as DNA barcodes for Ariocarpus species identification.
Keywords DNA barcoding; Ariocarpus; species identification; genetic distance; phylogenetic analysis
基金项目:拱北海关科研项目(2023GK006);科技基础资源调查专项(2023FY100402)
第一作者:水克娟(1981—),女,汉族,河北保定人,硕士,农艺师,主要从事植物濒危物种鉴定及植物检疫工作,E-mail: 121050174@qq.com
通信作者:张卫东(1979—),男,汉族,浙江浦江人,硕士,研究员,主要从事濒危物种鉴定及植物检疫工作,E-mail: 68724000@ qq.com
1. 拱北海关技术中心 珠海 519000
2. 福建农林大学生物安全全国重点实验室 福州 350002
1. Technical Center of Gongbei Customs District, Zhuhai 519000
2. State Key Laboratory of Agricultural and Forestry Biosecurity, Fujian Agriculture and Forestry University, Fuzhou 350002
岩牡丹属(Ariocarpus)是仙人掌科中具有显著生态特化特征的肉质植物类群,该属物种主要分布于墨西哥北部的奇瓦瓦沙漠地区[1],包括以下6个物种:具三角状疣突的岩牡丹(A. retusus)、表皮具辐射状沟纹的龟甲牡丹(A. fissuratus)、扁平莲座状的黑牡丹(A. kotschoubeyanus)、刺座退化的龙舌兰牡丹(A. agavoides)、疣突螺旋排列的龙角牡丹(A. scaphirostris)和墨西哥产地特有种勃氏牡丹(A. bravoanus)[2-4]。据报道,岩牡丹属植物含有多种重要化合物,如萜类和皂苷,酚类衍生物(如苯酚、酚酸、黄酮类、花青素),以及第三类由含氮化合物组成的代谢物(包括生物碱),这些成分均具有显著的生物药理活性,使其成为天然药物筛选的重要研究对象,尤其是在抗菌制剂和抗氧化剂开发领域具有显著提取价值,在生物活性成分化学提取方面需求广泛[5]。在园艺栽培领域,该岩牡丹属植物因其缓慢的生长速率(年均增长0.5~1.0 cm)和独特的石质化外观,成为小型盆栽仙人掌类中的珍稀观赏品种,被多肉植物爱好者收集和珍藏。由于其独特的生长形态和观赏价值,岩牡丹属植物长期遭受盗采(尤其是园艺贸易),导致其野生种群数量下降[6]。另一方面,因农业扩张和城市化建设等原因,岩牡丹属等植物栖息地被占用或破坏,加剧了其濒危程度。因此,自1992年起,墨西哥立法将岩牡丹属植物列为特殊保护物种,限制其国际贸易。
为有效保护岩牡丹属植物,亟待整合分子生物学与野外生态学等研究领域,保障这一独特物种的生存。以往岩牡丹属植物相关研究主要集中在形态与显微结构观察[3]、植物化学组分分析[5]、离体培养体系构建(如愈伤组织诱导与细胞悬浮培养)[6]等方面。尽管近年来分子生物学技术逐步应用于该属研究[2],但基于DNA条形码技术对该物种进行准确鉴定的研究仍较少。DNA条形码技术是一种利用短DNA片段实现物种快速准确鉴定的技术,在动植物鉴定中是对传统鉴定方法的重要补充[7-9]。此外,该技术还在临床医学、农业生产、司法鉴定、非法贸易监管及生态保护等领域具有广泛应用价值[10-13]。植物DNA条形码不仅可用于鉴定未知样本的物种身份,尤其在形态鉴定困难或样本为幼体等情况下具有优势,而且对于受国际贸易公约保护的植物物种,DNA条形码可用于区分保护物种和非保护物种,从而为物种保护工作提供技术支持。DNA条形码可成为植物保护的有力工具,其核心价值在于能够鉴定未知植物材料,例如海关查获的非法采集标本[14]。同时,该技术不受形态特征或生理状态限制,无需专业分类学知识即可实现物种鉴定,通过通用引物扩增标准化DNA条形码片段,为岩牡丹属植物鉴定建立了普适性鉴定标准。大量研究已证实DNA条形码技术在物种真伪鉴定中具有可靠的应用价值[15]。
1 材料与方法
1.1 试验材料
1.1.1 样品来源
本研究收集了岩牡丹属不同来源个体,共6个物种42份样品(表1),全部由拱北海关在口岸查获。其中,勃氏牡丹(A. bravoanus)6个、黑牡丹(A. kotschoubeyanus)7个、龙角牡丹(A. scaphirostrus)4个、龟甲牡丹(A. fissuratus)6个、龙舌兰牡丹(A. agavoides)5个、岩牡丹(A. retusus)14个。样品均由相关专家复核鉴定,并采集新鲜植物组织或留存冷冻组织进行后续试验。试验材料具体信息见表1。
1.1.2 主要试剂
2×Taq Master Mix(艾克瑞生物);2000 bp Marker(艾克瑞生物);其他试剂均为分析纯。
1.1.3 主要仪器
全自动核酸提取仪(KingFisher mL,赛默飞世尔科技(中国)有限公司);NanoDrop超微量分光光度计(ND-1000,赛默飞世尔科技(中国)有限公司);PCR 扩增仪(TC-96/G/H(b)B,杭州博日科技股份有限公司);电泳仪(PowerPac Basic,伯乐生命医学产品(上海)有限公司);凝胶成像系统(Chemidoc Go,伯乐生命医学产品(上海)有限公司)。
1.2 试验方法
1.2.1 基因组DNA提取、基因扩增及测序
从6个物种42份样品中采集其组织,加入液氮充分研磨,采用本实验室改良的CTAB-磁珠法[16]提取基因组DNA,分别利用Nanodrop和1%琼脂糖凝胶电泳对DNA质量和浓度进行检测和定量,检测合格后稀释至30 ng/μL 用作PCR扩增的模板DNA,于-20℃冷冻保存。
参考美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)中GenBank收录的岩牡丹属叶绿体基因组序列matK(ON602050.1)、trnS-trnG(HQ324615.1)、atpH-atpI(HQ324551.1)设计了用于扩增岩牡丹属相应DNA片段的特异性引物(表2),并委托引物合成公司合成。各基因片段的PCR扩增反应体系为30 μL,其中ApexHF HSTaq DNA聚合酶0.6 μL,2×Taq Master Mix 15 μL[包含2X ApexHF CL缓冲液(Mg2+, dNTP plus)],正反向引物各1.2 μL,DNA模板2 μL,ddH2O 10 μL。PCR 反应条件及扩增程序见表2。PCR 扩增产物经琼脂糖凝胶电泳检测后,送交测序公司进行双向测序。
1.2.2 序列分析和系统进化树构建
使用DNASTAR Lasergene 7.1软件包中的Seq-Man软件[17]对测序活动的原始序列进行拼接和校对,手动去除低质量区和引物区,获得matK、trnS-trnG、atpH-atpI基因序列,并保存为FASTA格式。利用 DnaSP 6.12.03软件[18]统计每个基因片段的插入或缺失碱基(Indel)和单核苷酸多态性(Single Nucleotide Polymorphism,SNP),并进行单倍型分析。用MEGA 11软件[19]对测序得到的基因序列进行序列比对分析,统计变异位点和简约信息位点,并计算种间、种内Kimura双参数模型(K2P)遗传距离;采用邻接法(Neighbor-Joining,NJ)构建系统进化树,只有同一个物种的不同个体在构建的NJ系统树上形成单系分支才认为物种鉴定成功[20]。
2 结果与分析
2.1 样品的PCR检测结果与序列特征分析
由于前期预实验中,利用核基因ITS片段、PEPC以及2个叶绿体基因片段psbA-trnH、rbcL在岩牡丹属植物6个物种中的PCR扩增及测序结果均不理想,同时,鉴于matK、trnS-trnG和atpH-atpI作为DNA条形码在物种鉴定中已经广泛应用。因此,本研究参考岩牡丹属植物在NCBI上的叶绿体基因组序列,针对3个候选叶绿体DNA片段(matK、trnS-trnG和atpH-atpI)重新设计合成引物(表2),并对收集的岩牡丹属6个物种的42个个体(表1)进行了PCR扩增。所有样品的基因组 DNA 提取质量较高,并成功进行了 3 个叶绿体DNA片段的 PCR 扩增及 PCR 产物测序,扩增和测序成功率均为 100%。PCR产物经1.0%琼脂糖凝胶电泳后,于凝胶成像系统中呈现特征性单一条带(图1),且条带位置与预期片段长度基本一致。其中,matK DNA片段大小均为1150 bp(图1A),trnS-trnG DNA片段大小均为810 bp(图1B),atpH-atpI DNA片段长度分别为620 bp或770 bp(图1C),这是由于黑牡丹不同个体中存在Barcoding gap差异,导致atpH-atpI DNA片段扩增出现2种不同长度的PCR产物。
获得的PCR片段经测序分析结果见表3,候选的3条DNA条形码序列(trnS-trnG、matK和atpH-atpI)的GC含量按从高到低依次排列为:trnS-trnG>matK>atpH-atpI。序列的变异性通过变异位点与简约信息位点占总序列长度的百分比来表示。其中,atpH-atpI 展现出最高的变异性,变异位点占总序列的5.96%,表明该DNA条形码具有最高的变异程度;其次为trnS-trnG,其变异位点占总序列的2.59%,显示出中等程度的变异性;变异性最低的是matK,其变异位点仅占总序列的0.96%。
2.2 不同 DNA 片段序列单倍型分析
单倍型分析结果显示,在取样的全部 42 份岩牡丹属材料中,matK 序列共出现了5个单倍型,分别将其命名为M1—M5。具体而言,5个龙舌兰牡丹为M1单倍型,6个勃氏牡丹和4个龙角牡丹为M2单倍型,6个龟甲牡丹为M3单倍型,7个黑牡丹为M4单倍型,14个岩牡丹为M5单倍型。以上结果说明单个matK序列无法有效地对勃氏牡丹和龙角牡丹进行区分。对trnS-trnG序列分析发现,出现了共6种单倍型,分别将其命名为T1—T6。具体而言,4个龙角牡丹和6个龟甲牡丹为T1单倍型;5个龙舌兰牡丹为T2单倍型;6个勃氏牡丹为T3单倍型;3个黑牡丹(Ak001H、Ak002H、Ak006Z)为T4单倍型;4个黑牡丹(Ak007Z、Ak008Z、Ak014H、Ak027J)为T5单倍型;14个岩牡丹为T6单倍型。以上结果说明单个trnS-trnG序列无法有效地对龙角牡丹和龟甲牡丹进行区分。对atpH-atpI序列分析发现,出现了共7种单倍型,分别将其命名为A1—A6。具体而言,4个龙角牡丹和6个龟甲牡丹为A1单倍型;5个龙舌兰牡丹为A2单倍型;4个勃氏牡丹(Ab001Z、Ab002Z、Ab003H、Ab025J)为A3单倍型;2个勃氏牡丹(Ab011Z、Ab012Z)为A4单倍型;3个黑牡丹(Ak001H、Ak002H、Ak006Z)为A5单倍型;4个黑牡丹(Ak007Z、Ak008Z、Ak014H、Ak027J)为A6单倍型;14个岩牡丹为A7单倍型。以上结果说明单个atpH-atpI序列也无法对龙角牡丹和龟甲牡丹有效地进行区分。
结合上述分析结果,由于单个trnS-trnG和atpH-atpI序列均无法有效地对龙角牡丹和龟甲牡丹进行区分,因此本研究利用上述DNA片段组合(trnS-trnG+matK,atpH-atpI+matK)进行单倍型分析。结果表明,对trnS-trnG+matK组合序列分析发现,共出现了7种单倍型,分别将其命名为TM1—TM7(表4)。其中,4个龙角牡丹为TM1单倍型,5个龙舌兰牡丹为TM2单倍型,6个勃氏牡丹为TM3单倍型,6个龟甲牡丹为TM4单倍型,3个黑牡丹(Ak001H、Ak002H、Ak006Z)为TM5单倍型,4个黑牡丹(Ak007Z、Ak008Z 、Ak014H、Ak027J)为TM6单倍型,14个岩牡丹为TM7单倍型。因此,trnS-trnG+matK组合序列能够有效地对42个岩牡丹材料进行区分。
经对atpH-atpI+matK组合序列进一步分析发现,共出现了8种单倍型,分别将其命名为AM1—AM8(表5)。其中,4个龙角牡丹为AM1单倍型,5个龙舌兰牡丹为AM2单倍型,4个勃氏牡丹(Ab001Z、Ab002Z、Ab003H 、Ab025J)为AM3单倍型,2个勃氏牡丹(Ab011Z、Ab012Z])为AM4单倍型, 6个龟甲牡丹为AM5单倍型,3个黑牡丹(Ak001H、Ak002H、Ak006Z)为AM6单倍型,4个黑牡丹(Ak007Z、Ak008Z 、Ak014H、Ak027J)为AM7单倍型,14个岩牡丹为AM8单倍型。因此,atpH-atpI+matK组合序列可以有效地对42个岩牡丹材料进行区分。
2.3 遗传距离和 Barcoding gap分析
为进一步评估3个DNA条形码及其组合的Barcoding gap,本研究分别对它们的种间和种内遗传距离进行了分布统计,并对结果进行可视化展示(图2)。分析发现,所有DNA条形码序列的种间与种内遗传距离均存在一定程度的重叠情况。种内遗传距离主要集中在0~0.06这一区间,而种间遗传距离分布相对更为离散。其中,trnS-trnG+matK与 atpH-atpI+matK的重叠度最小且分布有偏向两端的趋势。一般而言,种内样品的分布相对集中,变异程度较小;种间样品的分布则较为分散,变异程度较大[21-22],这种特性有利于将其应用于岩牡丹属植物种质资源的鉴定。相比之下,其余条形码均存在明显的种间与种内遗传距离重叠现象,且分布趋势不够清晰明确。因此,trnS-trnG+matK与 atpH-atpI+matK片段组合具备作为鉴别岩牡丹属植物DNA条形码的应用潜力。
2.4 分子系统发育树的聚类分析
由于对matK、trnS-trnG与atpH-atpI 3个DNA片段序列单独建树无法完全将6种岩牡丹属植物进行区分(进化树结果未展示),这与前面单倍型以及Barcoding gap分析结果一致。因此,本研究利用叶绿体基因组trnS-trnG+matK和atpH-atpI+matK片段组合分别构建 NJ 进化树,靴带值(Bootstrap)1000次检验各分支的置信度,得到的系统发育树均能很好地聚成6个进化支系(图3),可以准确鉴别6种岩牡丹属植物。其中,龙舌兰牡丹(A. agavoides)聚在一起组成Clade l,黑牡丹(A.kotschoubeyanus)聚在一起组成Clade 2,岩牡丹(A. retusus)聚在一起组成Clade 3,龙角牡丹(A. scaphirostrus)聚在一起组成Clade 4,勃氏牡丹(A. bravoanus)聚在一起组成Clade 5,龟甲牡丹(A. fissuratus)聚在一起组成Clade 6。上述结果表明,trnS-trnG+matK和atpH-atpI+matK两种片段组合对物种分辨率高达100%,可将岩牡丹属6个物种完全区分开。
3 讨论
理想的DNA条形码需兼具通用引物扩增效率与测序成功率,同时具备显著的物种鉴别能力[23-24]。本研究所选的7个DNA片段中,3个候选叶绿体DNA片段(matK、trnS-trnG和atpH-atpI)呈现100%的PCR扩增与测序成功率(表2),另外1个核基因ITS片段以及3个叶绿体基因片段(PEPC、psbA-trnH、rbcL)均存在非特异扩增或测序结果不理想等情况,该结果与早期研究结论一致[14]。Yessson等[14]通过实验发现,使用通用引物进行ITS扩增时仅成功扩增了3个物种,经特异性引物优化后所有物种的PCR均产生多条带,导致无法直接测序,推测这与仙人掌核DNA基因家族的进化特征(如假基因或未完全协同进化)有关。相比之下,matK片段序列成功区分了71%的岩牡丹属物种,但其种内遗传变异范围限制了鉴定分辨率。因此,ITS因个体内多态性不适用于岩牡丹属植物的分子鉴定,而仅依赖matK的变异度不足以实现岩牡丹属植物的物种水平鉴定[14]。此外,李井干等[2]利用PEPC序列可以对岩牡丹属6种物种21份材料较好地进行区分。然而,本文作者在利用其研究中所提供的引物对本研究42个岩牡丹属植物样品进行扩增时发现,部分样品DNA测序结果不理想,无法得到其DNA序列,初步推测可能是由于本研究样本量较大(扩大了一倍),部分材料的来源与遗传背景与李井干等[2]使用的试验材料存在较大的差异,导致无法获得完整的PEPC序列。
鉴于单一位点序列在低阶分类鉴定中往往无法提供充分信息,研究者常采用DNA多片段组合从而显著提升物种鉴别能力[15,25]。例如,在蔷薇科李属(Prunus L.)的物种鉴定中,研究人员采用atpB-rbcL与trnL-F或atpF-H等组合标记,成功实现了该属5个物种的完全鉴别[26]。Parveen等[27]通过系统比较兰科植物中rbcL、rpoB、rpoC1、matK及ITS等标记组合的鉴别效能,发现ITS与matK的协同使用可达到最大物种分辨率。Liu等[28]通过筛选评估,发现由3个DNA片段(ITS+trnP-psaJ-rpI33+psbB-psbH)组成的一组条形码,能够快速且可靠地区分6个类群45种野生百合(Lilium)物种,为野生百合的分类、濒危物种保护和选择性百合育种提供了有效方法。以上研究证实,基于功能互补原则的标记组合策略能有效突破单一位点标记的鉴别局限。
在本研究中,单个叶绿体DNA片段(matK、trnS-trnG和atpH-atpI)均无法对6种42个岩牡丹属植物进行有效区分,最多只能区分其中4种,如matK序列无法有效地对勃氏牡丹和龙角牡丹进行区分,而单个trnS-trnG或atpH-atpI序列无法有效地对龙角牡丹和龟甲牡丹进行区分。为解决上述问题,本文作者组合使用trnS-trnG+matK与atpH-atpI+matK片段,从而很好地实现了6种岩牡丹属植物物种间的有效鉴别。因此,建议将trnS-trnG+matK与atpH-atpI+matK片段组合作为岩牡丹属物种鉴定的DNA条形码。但本研究在野生种群代表性方面还需进一步完善,未来仍需纳入更多野生种群样本,以便进一步验证DNA条形码的通用性。
参考文献
[1] Aguilar-Morales G, Martínez-Peralta C, Feria Arroyo T, et al. Distribución geográfica del género Ariocarpus Scheidweiler (Cactaceae)[J]. Cact Succulentas Mex, 2011, 56: 49-63.
[2] 李井干, 刘晓宇, 伏建国, 等. PEPC序列在濒危岩牡丹属植物分子鉴定中的应用[J]. 植物检疫, 2023, 37(3): 15-21.
[3] Morales L J. Distribution, ecology and conservation of the genus Ariocarpus (Cactaceae) in Tamaulipas State, México[J]. Kaktusy I Inne, 2007, 2: 3-30.
[4] 兑宝峰. 仙人掌科岩牡丹属植物[J]. 中国花卉园艺, 2020(10): 42-44.
[5] Rodríguez Garza R, Morales M, Verde-Star M, et al. Antibacterial and antifungal activity of species Ariocarpus kotsch- oubeyanus (Lemaire) and Ariocarpus retusus (Scheidweiler) (Cactaceae)[J]. Revista Mexicana de Ciencias Farmaceuticas, 2010, 41: 55-59.
[6] Fiedler I, Santacruz-Ruvalcaba F, Portillo L. Establishment of callus and cell suspension cultures of Ariocarpus retusus (Scheid- weiler)[J]. Journal of the Professional Association for Cactus Develo- pment, 2022, 24: 1-17.
[7] 张伟, 范晓虹, 邵秀玲, 等. DNA条形码在检疫性杂草银毛龙葵鉴定中的应用研究[J]. 植物检疫, 2013, 27(3): 60-65.
[8] 程芳婷, 李忠虎, 刘春艳, 等. 地黄属植物的DNA条形码研究[J]. 植物科学学报, 2015, 33(1): 25-32.
[9] 高连明. DNA条形码在生物多样性编目与评价中的应用[J]. 生物多样性, 2015, 23(3): 286-287.
[10] 施佳辉, 田飞, 唐蜀昆, 等. DNA条形码技术及其在微生物资源鉴别保护中的应用研究[J]. 微生物学杂志, 2018, 38(4): 77-83.
[11] 田旭飞, 曲波. DNA条形码在入侵植物鉴定中的应用进展[J]. 生物安全学报, 2016, 25(2): 99-105.
[12] 蔡金龙, 谢世清, 张广辉, 等. 药用植物DNA条形码鉴定研究进展[J]. 植物科学学报, 2017, 35(3): 452-464.
[13] 张柠, 赵斐, 杨朔, 等. DNA条形码技术在法医学中的研究进展[J]. 昆明医科大学学报, 2018, 39(10): 130-133.
[14] Yesson C, Bárcenas R T, Hernández H M, et al. DNA barcodes for Mexican Cactaceae, plants under pressure from wild collecting[J]. Molecular Ecology Resources, 2011, 11(5): 775-783.
[15] Li D Z, Liu J Q, Chen Z D, et al. Plant DNA barcoding in China[J]. Journal of Systematics and Evolution, 2011, 49(3): 165-168.
[16] 权永兵, 黄永辉, 徐淼锋, 等. 改良十六烷基三甲基溴化铵—磁珠核酸提取方法及在植物转基因检测中的应用[J]. 食品安全质量检测学报, 2019, 10(23): 8042-8047.
[17] Burland T G. DNASTAR’s Lasergene sequence analysis software[J]. Methods in Molecular Biology (Clifton, N.J.), 2000, 132: 71-91.
[18] Rozas J, Ferrer-Mata A, Sánchez-DelBarrio J C, et al. DnaSP 6: DNA sequence polymorphism analysis of large data sets[J]. Molecular Biology and Evolution, 2017, 34(12): 3299-3302.
[19] Tamura K, Stecher G, Kumar S. MEGA11: Molecular evolutionary genetics analysis version 11[J]. Molecular Biology and Evolution, 2021, 38(7): 3022-3027.
[20] Li D Z, Gao L M, Li H T, et al. Comparative analysis of a large dataset indicates that internal transcribed spacer (ITS) should be incorporated into the core barcode for seed plants[J]. Proceedings of the National Academy of Sciences, 2011, 108(49): 19641-19646.
[21] 卢志忠, 刘巧珍, 潘蔚霖, 等. 基于DNA条形码的南蛇藤属植物分子鉴定[J/OL]. 分子植物育种, (2025-01-12)[2025-09-12]. https://link.cnki.net/urlid/46.1068.S.20250325.2124.002.
[22] 丁泓帆, 刘向东, 蒋文秀, 等. 兰属植物核心DNA条形码的筛选与评价[J/OL]. 分子植物育种, (2025-01-14)[2025-09-12]. https://link.cnki.net/urlid/46.1068.S.20230529.1533.032.
[23] Kress W J, Erickson D L. A Two-locus global DNA barcode for land plants: The Coding rbcL gene complements the non-coding trnH-psbA spacer region[J]. Plos One, 2007, 2(6): e508. DOI: 10.1371/journal.pone.0000508
[24] Kress W J, Wurdack K J, Zimmer E A, et al. Use of DNA barcodes to identify flowering plants[J]. Proceedings of the National Academy of Sciences, 2005, 102(23): 8369-8374.
[25] Jiang K W, Zhang R, Zhang Z F, et al. DNA barcoding and molecular phylogeny of Dumasia (Fabaceae: Phaseoleae) reveals a cryptic lineage[J]. Plant Diversity, 2020, 42(5): 376-385.
[26] Quan X, Zhou S L. Molecular identification of species in Prunus sect. Persica (Rosaceae), with emphasis on evaluation of candidate barcodes for plants[J]. Journal of Systematics and Evolution, 2011, 49(2): 138-145.
[27] Parveen I, Singh H K, Malik S, et al. Evaluating five different loci (rbcL, rpoB, rpoC1, matK, and ITS) for DNA barcoding of Indian orchids[J]. Genome, 2017, 60(8): 665-671.
[28] Liu Y, Zhang M, Chen X, et al. Developing an efficient DNA barcoding system to differentiate between Lilium species[J]. BMC Plant Biology, 2021, 21(1): 465.
表1 试验材料相关信息
Table 1 Sample numbers and information of experimental materials
编号 | 试样编号 | 种类 | 拉丁文 | 样品类型 |
1 | Ab001Z | 勃氏牡丹 | Ariocarpus bravoanus | 新鲜植物组织 |
2 | Ab002Z | 勃氏牡丹 | Ariocarpus bravoanus | 新鲜植物组织 |
3 | Ab003H | 勃氏牡丹 | Ariocarpus bravoanus | 新鲜植物组织 |
4 | Ab025J | 勃氏牡丹 | Ariocarpus bravoanus | 冷冻组织 |
5 | Ab011Z | 勃氏牡丹 | Ariocarpus bravoanus | 新鲜植物组织 |
6 | Ab012Z | 勃氏牡丹 | Ariocarpus bravoanus | 新鲜植物组织 |
7 | Ak006Z | 黑牡丹 | Ariocarpus kotschoubeyanus | 新鲜植物组织 |
8 | Ak001H | 黑牡丹 | Ariocarpus kotschoubeyanus | 新鲜植物组织 |
9 | Ak002H | 黑牡丹 | Ariocarpus kotschoubeyanus | 新鲜植物组织 |
10 | Ak007Z | 黑牡丹 | Ariocarpus kotschoubeyanus | 新鲜植物组织 |
11 | Ak008Z | 黑牡丹 | Ariocarpus kotschoubeyanus | 新鲜植物组织 |
12 | Ak014H | 黑牡丹 | Ariocarpus kotschoubeyanus | 新鲜植物组织 |
13 | Ak027J | 黑牡丹 | Ariocarpus kotschoubeyanus | 冷冻组织 |
14 | As009Z | 龙角牡丹 | Ariocarpus scaphirostrus | 新鲜植物组织 |
15 | As010Z | 龙角牡丹 | Ariocarpus scaphirostrus | 新鲜植物组织 |
16 | As005H | 龙角牡丹 | Ariocarpus scaphirostrus | 新鲜植物组织 |
17 | As015H | 龙角牡丹 | Ariocarpus scaphirostrus | 新鲜植物组织 |
18 | Af023Z | 龟甲牡丹 | Ariocarpus fissuratus | 新鲜植物组织 |
19 | Af024Z | 龟甲牡丹 | Ariocarpus fissuratus | 新鲜植物组织 |
20 | Af007H | 龟甲牡丹 | Ariocarpus fissuratus | 新鲜植物组织 |
21 | Af011H | 龟甲牡丹 | Ariocarpus fissuratus | 新鲜植物组织 |
22 | Af094J | 龟甲牡丹 | Ariocarpus fissuratus | 冷冻组织 |
23 | Af194J | 龟甲牡丹 | Ariocarpus fissuratus | 冷冻组织 |
24 | Aa015Z | 龙舌兰牡丹 | Ariocarpus agavoides | 新鲜植物组织 |
25 | Aa016Z | 龙舌兰牡丹 | Ariocarpus agavoides | 新鲜植物组织 |
26 | Aa004H | 龙舌兰牡丹 | Ariocarpus agavoides | 新鲜植物组织 |
27 | Aa009H | 龙舌兰牡丹 | Ariocarpus agavoides | 新鲜植物组织 |
28 | Aa080J | 龙舌兰牡丹 | Ariocarpus agavoides | 冷冻组织 |
29 | Ar013Z | 岩牡丹 | Ariocarpus retusus | 新鲜植物组织 |
30 | Ar014Z | 岩牡丹 | Ariocarpus retusus | 新鲜植物组织 |
31 | Ar017Z | 岩牡丹 | Ariocarpus retusus | 新鲜植物组织 |
32 | Ar018Z | 岩牡丹 | Ariocarpus retusus | 新鲜植物组织 |
33 | Ar006H | 岩牡丹 | Ariocarpus retusus | 新鲜植物组织 |
34 | Ar008H | 岩牡丹 | Ariocarpus retusus | 新鲜植物组织 |
35 | Ar012H | 岩牡丹 | Ariocarpus retusus | 新鲜植物组织 |
36 | Ar026J | 岩牡丹 | Ariocarpus retusus | 冷冻组织 |
37 | Ar078J | 岩牡丹 | Ariocarpus retusus | 冷冻组织 |
38 | Ar085J | 岩牡丹 | Ariocarpus retusus | 冷冻组织 |
39 | Ar114J | 岩牡丹 | Ariocarpus retusus | 冷冻组织 |
40 | Ar115J | 岩牡丹 | Ariocarpus retusus | 冷冻组织 |
41 | Ar139J | 岩牡丹 | Ariocarpus retusus | 冷冻组织 |
42 | Ar140J | 岩牡丹 | Ariocarpus retusus | 冷冻组织 |
表1(续)
表2 DNA条形码片段扩增引物及反应条件
Table 2 DNA barcode primers and amplification program
条形码 | 引物名称 | 引物序列 | PCR反应条件 | 长度 (bp) | 来源 |
matK | matK-Ar-F | CAAACTCTTCGTTACTGGGTG | 94°C 1 min; 98°C 10 s, 56°C 30 s, 68°C 45 s, 35 cycles; 68°C 5 min | 1150 | 自行设计 |
matK-Ar-R | ACTTACTTATTCCGTCATTCATTTAGG | ||||
atpH-atpI | atpH-atpI-Ar-F | TTCCAGGCCCAATAGAAGCAAG | 94°C 3 min; 98°C 10 s, 60°C 30 s, 68°C 45 s, 35 cycles; 68°C 5 min | 620 ~770 | 自行设计 |
atpH-atpI-Ar-R | AGCCGCAGCTTATATAGGCG | ||||
trnS-trnG | trnS-trnG-Ar-F | GTTAGACTTCCAGGAGAGGAAG | 94°C 3 min; 98°C 10 s, 60°C 30 s, 68°C 45 s, 35 cycles; 68°C 5 min | 810 | 自行设计 |
trnS-trnG-Ar-R | CTAAACTATACCCGCTACAGTTC |
表3 岩牡丹属植物3个DNA条形码序列特征
Table 3 Characteristics of three DNA barcodes in Ariocarpus
序列 | (扩增成功率%) | (测序成功率%) | 序列长度 (bp) | 变异位点 (占比%) | 简约信息位点 (占比%) | GC含量 (%) |
matK | 42 (100) | 42 (100) | 826 | 8 (0.96) | 8 (0.96) | 33.66 ~34.02 |
trnS-trnG | 42 (100) | 42 (100) | 773 | 20 (2.59) | 20 (2.59) | 40.49 ~41.27 |
atpH-atpI | 42 (100) | 42 (100) | 581~755 | 45 (5.96) | 45 (5.96) | 29.95 ~31.60 |
A: matK DNA扩增片段; B: trnS-trnG DNA扩增片段; C: atpH-atpI DNA扩增片段
M: DNA Marker DL2000; 1: 龙舌兰牡丹; 2: 岩牡丹; 3: 龟甲牡丹; 4: 勃氏牡丹; 5: 龙角牡丹; 6~7: 黑牡丹
图1 matK、trnS-trnG、atpH-atpI DNA片段凝胶电泳图
Fig.1 Electrophoretogram of matK/trnS-trnG/atpH-atpI gene sequence amplification products
表4 岩牡丹属植物trnS-trnG+matK片段组合基因单倍型数量及变异位点
Table 4 Haplotype distribution and polymorphic sites of the combined trnS-trnG+matK regions in Ariocarpus
单倍型 | trnS-trnG+matK序列变异位点 | |||||||||||||||||||||||||||
9 | 29 | 163 | 262 | 309 | 321 | 324 | 338 | 350 | 354 | 479 | 540 | 556 | 582 | 684 | 697 | 735 | 742 | 749 | 758 | 800 | 921 | 1033 | 1158 | 1391 | 1447 | 1533 | 1564 | |
TM 1 | A | A | G | T | A | T | C | G | C | G | T | T | A | G | A | G | G | G | T | G | G | A | C | A | G | G | T | A |
TM 2 | G | A | A | C | G | C | T | G | C | G | G | A | T | T | A | G | G | G | C | A | A | T | C | T | G | G | T | T |
TM 3 | A | A | G | T | G | T | C | T | C | G | T | T | A | G | A | G | G | G | T | G | G | A | C | A | G | G | T | A |
TM 4 | A | A | G | T | A | T | C | G | C | G | T | T | A | G | A | G | G | G | T | G | G | A | C | A | G | G | C | A |
TM 5 | A | G | A | T | G | C | T | G | T | T | G | A | A | T | C | G | T | G | T | G | G | T | C | T | T | A | T | T |
TM 6 | A | G | A | T | G | C | T | G | T | T | G | A | A | T | A | T | T | T | T | G | G | T | C | T | T | A | T | T |
TM 7 | A | A | G | T | G | C | C | G | C | G | G | T | A | T | A | G | G | G | T | A | A | T | T | T | G | G | T | A |
表5 岩牡丹属植物atpH-atpL+matK片段组合基因单倍型数量及变异位点
Table 5 Haplotype distribution and polymorphic sites of the combined atpH-atpL+matK regions in Ariocarpus
单倍型 | atpH-atpL+matK序列变异位点 | |||||||||||||||||||
48 | 193-197 | 68-240 | 327 | 378 | 394 | 395 | 648 | 716 | 723 | 729-737 | 743 | 782 | 903 | 1015 | 1140 | 1373 | 1429 | 1515 | 1546 | |
AM 1 | T | -5 bp | +172 bp | T | G | T | A | T | G | T | +9 bp | G | G | A | C | A | G | G | T | A |
AM 2 | T | +5 bp | +172 bp | T | G | A | A | G | T | T | +9 bp | G | A | T | C | T | G | G | T | T |
AM 3 | T | -5 bp | +172 bp | T | G | T | T | T | G | A | +9 bp | G | G | A | C | A | G | G | T | A |
AM 4 | T | -5 bp | +172 bp | T | G | T | T | T | G | T | +9 bp | G | G | A | C | A | G | G | T | A |
AM 5 | T | -5 bp | +172 bp | T | G | T | A | T | G | T | +9 bp | G | G | A | C | A | G | G | C | A |
AM 6 | -1 bp | -5 bp | -172 bp | T | T | T | A | G | G | T | +9 bp | G | G | T | C | T | T | A | T | T |
AM 7 | -1 bp | -5 bp | -172 bp | T | T | T | A | G | G | T | -9 bp | A | G | T | C | T | T | A | T | T |
AM 8 | T | -5 bp | +172 bp | C | G | T | A | G | G | T | +9 bp | G | A | T | T | T | G | G | T | A |
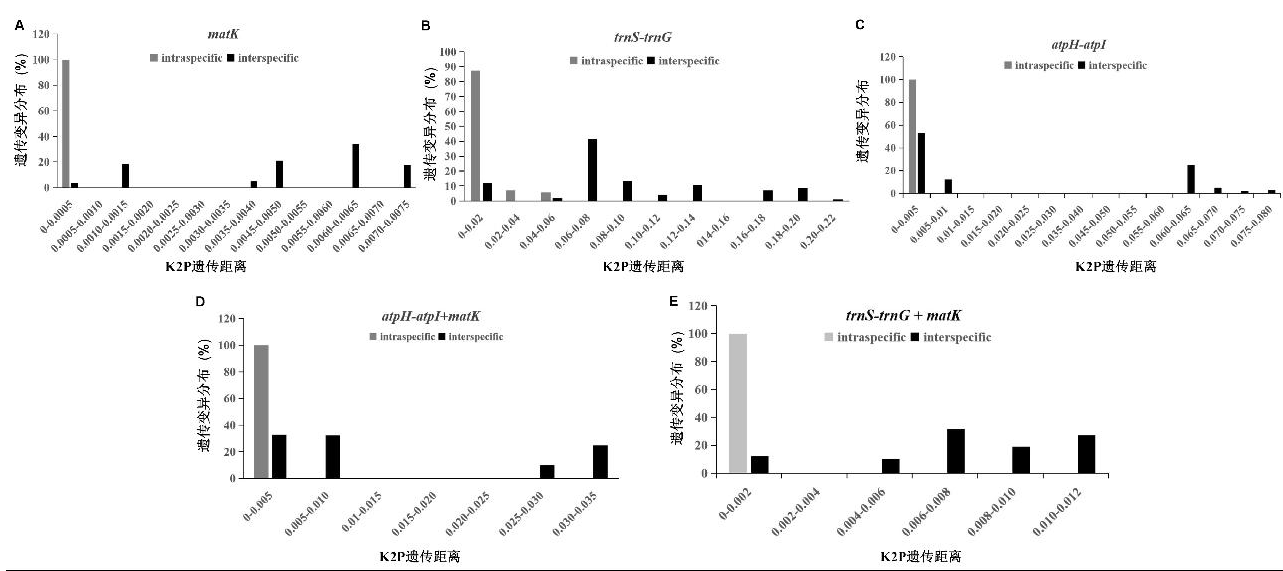
A: matK序列; B: trnS-trnG序列; C: atpH-atpI序列; D: atpH-atpI+matK序列; E: trnS-trnG+matK序列
图2 岩牡丹属植物种内和种间变异的Barcoding gap分布
Fig.2 Barcoding gap analysis of interspecific and intraspecific variation in Ariocarpus

A: 基于trnS-trnG+matK片段组合建树; B: 基于atpH-atpI+matK片段组合建树
图3 基于3个叶绿体DNA片段组合构建的岩牡丹属植物NJ系统发育树
Fig.3 Neighbor-joining (NJ) phylogenetic tree of Ariocarpus species based on combined chloroplast DNA sequences







