CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
重组酶聚合酶扩增技术在粮谷相关病原检测中的应用
作者:刘培海 姜涛 陈颖 李林杰 王凯 相湛昌 吴兴海 雷质文
刘培海 姜涛 陈颖 李林杰 王凯 相湛昌 吴兴海 雷质文
刘培海 1 姜 涛 1 陈 颖 2 李林杰 1 王 凯 1 相湛昌 1 吴兴海 3 雷质文 3 *
摘 要 重组酶聚合酶扩增技术(Recombinase Polymerase Amplification,RPA)是一种新型核酸等温扩增技术,具有灵敏度高、特异性强等特点,在食品安全、物种鉴定、病原检测等方面得到了广泛应用。本文概述了RPA方法的技术原理、RPA产物的分析方法和RPA技术在粮谷相关病原检测中的研究进展情况,为RPA技术在粮谷相关病原检测的研究和应用提供参考。
关键词 粮谷;重组酶聚合酶扩增技术;病原检测
Application of Recombinase Polymerase Amplification in the Detection of Grain and Cereal-Related Pathogen
LIU Pei-Hai1 JIANG Tao1 CHEN Ying2 LI Lin-Jie1 WANG Kai1
XIANG Zhan-Chang1 WU Xing-Hai3 LEI Zhi-Wen3*
Abstract As a novel isothermal nucleic acid amplification technology with high sensitivity and specificity, recombinase polymerase amplification (RPA) has been widely applied in many fields, such as food safety, pathogen detection and identification of species.This paper outlines the technical principles of RPA, analytical methods for RPA products, and research progress in RPA-based detection of grain and cereal-related pathogens, providing valuable insights for future research and application of RPA technology in this field.
Keywords grains and cereals; recombinase polymerase amplification (RPA); pathogen detection
植物病害是制约粮食作物产量、影响粮食安全的重要因素,因此粮谷相关病原的快速检测对保证粮食安全、植物病害防控具有重要意义[1-2]。目前,粮谷相关病原的检测方法主要有酶联免疫吸附法(Enzyme-Linked Immunosorbent Assay,ELISA)、聚合酶链式反应(Polymerase Chain Reaction,PCR)法和实时定量PCR法,这些方法仪器设备昂贵,检测时间较长,而且需要严格规范的实验设施和经验丰富的专业技术人员[3]。近年来,随着分子生物学技术的发展,研制了不依赖于温度循环扩增仪器的等温扩增技术,可在恒温下实现核酸扩增,甚至无需加热,简化核酸提取过程,使得现场检测和快速检测成为可能,可满足粮谷相关病原快速检测的要求。其中,重组酶聚合酶扩增技术(Recombinase Polymerase Amplification,RPA)对环境条件要求低、特异性强且不依赖于昂贵的仪器设备,能与其他技术结合实现可视化检测,适用于粮谷病原的快速检测和现场检测。本文概述了RPA方法的技术原理、RPA产物的分析方法和RPA技术在粮谷相关病原中的研究情况,旨在为后续研究和应用提供参考。
1 重组酶聚合酶扩增技术的原理
RPA技术是一种新型核酸等温扩增技术,是仿照T4噬菌体DNA的复制机制进行扩增。主要依赖于3种蛋白:来自T4噬菌体的重组酶uvsX及其装载辅助因子uvsY、单链结合蛋白(Single-Stranded DNA-Binding Protein,SSB)和链置换DNA聚合酶。在ATP的作用下,重组酶可与引物结合形成重组酶—引物复合体,该复合体可识别并定位引物在DNA双链上的同源序列,形成双链DNA上的重组环,从而使得DNA双链变性解链,RPA反应的解链过程无需加热。DNA双链解链后,被引物置换的母链与单链DNA结合蛋白结合,稳定其结构。重组酶—引物复合体主动水解反应体系中的ATP,重组酶和引物解离,引物3'端暴露出来,链置换DNA聚合酶结合延长引物,启动DNA双链扩增,进而完成ATP指数扩增反应[4-5]。RPA扩增原理及流程如图1所示。
2 RPA的技术特点
相比其他核酸扩增技术,RPA技术有其自身优势。RPA技术是恒温扩增技术,在温度为20~42℃时进行,无需昂贵的PCR扩增仪器设备。RPA技术对核酸纯度要求不高,能耐受核酸提取过程中的抑制剂,核酸提取过程可采用组织或细胞粗提取,大大提高工作效率[6-8]。RPA扩增仅需2条引物即可,如进行荧光检测,需设计一条探针,相比环介导等温核酸扩增(Loop-Mediated Isothermal Amplification,LAMP)方法需要设计4~6条引物,RPA方法特异性强,灵敏度一般比PCR方法或逆转录酶聚合酶链反应(Reverse Transcription-Polymerase Chain Reaction,RT—PCR)方法高10倍。
但RPA技术也有其自身的不足,没有专门的引物设计软件,需要自行设计引物;RPA扩增片段一般不超过500 bp,重组酶聚合酶扩增技术偶联侧向层析法(Recombinase Polymerase Amplification-Lateral Flow Dipstick,RPA-LFD)扩增片段一般不超过150 bp。RPA扩增产物因含有大量的蛋白,需纯化后才能进行下游工作,如琼脂糖凝胶电泳或基因克隆;RPA技术对引物造成的错配较为敏感,引物错配会导致产生假阳性,引物的一个错配即可降低反应的特异性[9-12]。
RPA技术与其他核酸扩增技术方法性能的比较见表1[13-14]。
3 RPA扩增产物的分析方法
3.1 琼脂糖凝胶电泳法
RPA产物主要采用琼脂糖凝胶电泳法对RPA基础反应体系中的扩增产物进行检测,由于反应体系中添加了多种酶和蛋白质,需对反应产物使用酚—氯仿法纯化后再进行琼脂糖凝胶电泳,防止其中的引物、酶、蛋白成分对凝胶电泳的干扰。经EB染色后,在紫外光的照射下生成可以检测的目的条带[15]。
3.2 实时荧光法
实时荧光方法是在反应体系中加入核酸外切酶(exonuclease Ⅲ,exo)和特异性荧光标记的exo探针。exo探针包括一个荧光报告基团和一个荧光淬灭基团,分别结合在极为接近的T碱基上(中间相隔1~5个碱基),两荧光基团中间位置设计有一个四氢呋喃(Tetrohydrofuran,THF)位点(脱甲基位点类似物)。在探针3'末端需要添加合适的阻隔基团,以阻断链的延伸。当结构保持完整时,体系内荧光强度较低。探针与目的序列结合时,THF位点被核酸外切酶Ⅲ识别并剪切,荧光报告基团与淬灭基团分离,扩增产物与荧光信号同步积累,可通过检测荧光信号强度实时监测目的基因片段的扩增情况。除exo探针外,fpg探针也可用于实时荧光定量的检测[16]。两种方法的原理图如图2所示[3]。
3.3 侧向层析法
使用RPA-LFD检测时,反应体系中需加入核酸内切酶Ⅳ(Endonuclease Ⅳ,nfo)、nfo探针和3'带有生物素标记的反向引物。nfo探针5'端带有FAM荧光报告基团标记,中间有THF碱基间隔,3'末端有C3阻隔基团。探针结合时,THF位点被核酸外切酶Ⅳ识别并剪切,产生3'端聚合酶结合位点,启动链的延伸,形成的扩增产物包含FAM标记和生物素标记,可使用包被有抗生物素抗体和抗FAM抗体的硝酸纤维素膜观察检测。方法的原理如图3所示[17]。
3.4 CRISPR/Cas系统法
RPA技术可与CRISPR/Cas技术相结合,用于快速检测病毒核酸。该技术首先通过RT-RPA等温条件下扩增目标序列,然后将扩增产物转入含有Cas12a、CrRNA等检测试剂的新反应管中,CRISPR/Cas12a可激活核酸酶活性进行检测。Gong等[18]建立了人类呼吸道合胞病毒A或B的RPA-CRISPR/Cas12荧光检测方法,可检测浓度为13.8 copies/μL的目的序列,能在37 min区分人类呼吸道合胞病毒A或B的感染。Qian等[19]建立了人类诺如病毒的RPA-CRISPR/Cas12a快速检测方法,最低检出限为9.65×103 copies/μL。CRISPR/Cas13a蛋白也可用于核酸检测,与RPA结合用于检测非洲猪瘟病毒[20]。
4 RPA技术在粮谷相关病原检测中的应用
RPA技术以其反应快速、反应条件要求低、灵敏度高、特异性强等优势在食品安全、物种鉴定、病原检测中得到广泛应用,在粮谷相关的真菌、细菌和病毒检测中的研究也取得系列进展,表2总结了国内外粮谷相关病原RPA检测方法的报道。
RPA技术反应快速、不依赖于昂贵的设备,而且可耐受核酸提取过程中的抑制剂,适合于粮谷相关病原的现场检测。目前,RPA技术在粮谷相关病原中的应用还处于初期研究阶段,虽然真菌、细菌和病毒相关方法均有涉及,但是覆盖病原种类特别是检疫性病原还较少。
4.1 真菌
4.1.1 大豆疫霉病菌
大豆疫霉病菌(phytophthora sojae)是大豆作物最具毁灭性的致病菌之一,可导致幼苗倒伏,成年植物根茎腐烂,遭受暴雨的地区植物会死亡,产量损失可达到100%。Dai等[22]以Ypt1基因为靶基因,建立了大豆疫霉病菌的RPA检测方法,反应时间仅25 min,与其他疫霉病菌、腐霉病菌无交叉反应,方法灵敏度高,50 μL反应体系可检测到10 pg/μL的DNA。将该方法用于130个大豆根际样品进行检测,结果与LAMP、PCR、叶片感染方法相比,检测阳性率分别为55.4%、54.6%、46.9%、38.5%~40.0%,RPA方法检测阳性率高于其他3种方法。
4.1.2 白术疫霉病菌
除大豆疫霉病菌外,20世纪90年代还发现了另一种大豆根部的致病菌,2009年被命名为白术疫霉病菌(phytophthora sansomeana)。J. Alejandro Rojas等[23] 以atp9-nad9保守区域为目的片段,建立了同时检测大豆疫霉病菌和白术疫霉病菌的实时荧光RPA方法,检测灵敏度对2种病菌均达到1 pg DNA,使用2013年和2014年收集的115份植物和土壤样品对方法进行了验证,结果表明方法正确性与实时定量PCR方法相当。
4.1.3 禾顶囊壳小麦变种
由禾顶囊壳小麦变种(Gaeumannomyces graminis var. tritici)引起的小麦全蚀病是危害极大的小麦土传真菌病害。鞠玉亮等[34]以β-tubulin为靶基因建立了小麦全蚀病菌的RPA-LFD快速检测技术,可在30 min内完成反应,实现小麦全蚀病菌的可视化检测,检测效率明显优于PCR方法和LAMP方法,检测限可达到10 pg/μL,与普通PCR方法相当,将该方法用于安徽地区9份疑似小麦全蚀病菌土块样品进行检测,结果与PCR方法一致。
4.2 细菌
4.2.1 玉米细菌性枯萎病菌
玉米细菌性枯萎病菌(Pantoeastewartii subsp. Stewarii,PSS)是维管束型病害,受害后植株矮缩或枯萎,对玉米造成极大伤害,是我国进境植物检疫性病原之一。目前的主要检测方法为分离培养、血清学检测、分子生物性检测,但是这些方法操作复杂、耗时较长。单长林等[32]以参与磷酸代谢的pst操纵子和相邻的glmS基因区间序列(pstS-glmS)为目的片段,建立了玉米细菌性枯萎病的RPA—实时荧光检测方法,灵敏度可达到280 fg/μL,与荧光定量PCR方法相当,应用该方法对30份样品进行检测,结果与SN/T 1375—2004《玉米细菌性枯萎病检疫鉴定方法》检测结果一致。
4.2.2 玉米内州萎蔫病菌
玉米内州萎蔫病菌(Clavibacter michiganensis subsp. nebraskensis)是玉米病害中又一种重要的细菌性病害,也是我国重点关注的检疫性病害之一。该病首先在美国内布拉斯加州中部发现,后逐步扩展至美国多个州和加拿大部分地区。单长林等[31]以celB基因为目的基因,建立了RPA—荧光检测方法,灵敏度可达130 fg/μL,利用该方法对4份玉米模拟阳性样品、8份真实样品进行检测,检测结果与GB/T 36840—2018《玉米内州萎蔫病菌检疫鉴定方法》检测结果一致。
4.3 病毒
4.3.1 番茄斑萎病毒
番茄斑萎病毒(Tomato Spotted Wilt Virus,TSWV)属于布尼亚病毒目、番茄斑萎病毒科、正番茄斑萎病毒属,基因组由3条单链RNA组成,分别为L(large)-RNA、M(medium)-RNA和S(small)-RNA,其中L-RNA为负义链RNA,其互补链编码RNA依赖性RNA聚合酶。M-RNA和S-RNA为双义链RNA,M-RNA的互补链编码2个糖蛋白G1和G2,病毒正义链编码非结构蛋白NSm,为病毒的运动蛋白(Movement Protein,MP);S-RNA的互补链编码外壳蛋白N(Nucleocapsid Protein),病毒链编码非结构蛋白NSs[35]。番茄斑萎病毒可随进境粮食传播,也是进境粮食重点关注的病毒项目。Zhang W [25]等以N蛋白编码基因为靶基因,建立了番茄斑萎病毒的RPA检测方法,使用CRISPR/Cas13a检测系统对扩增产物进行检测,20 min即可完成反应,检测限可达2.26×102 copies/μL,灵敏度是RT-PCR方法的10倍,并将该方法用于番茄叶子和西花蓟马传播载体上的病毒,结果与RT-PCR方法结果一致。
4.3.2 大麦黄矮病毒
大麦黄矮病毒(Barley Yellow Dwarf Virus,BYDV)是黄体病毒属的成员之一,是世界上谷物作物分布最广、破坏力最严重的病毒之一,可影响到大部分禾本科作物,包括大麦、小麦、玉米、燕麦和黑麦草等。Na-Kyeong Kim等[17]以大麦黄矮病毒包被蛋白基因保守序列为目标序列,建立了RPA-LFD快速检测方法,方法仅需20 min即可完成,与粮谷中其他病毒无交叉反应,灵敏度与RT-PCR方法相当,并将该方法用于现场收集的燕麦样品和带有病毒的蚜虫传播载体进行了验证。
4.3.3 玉米褪绿斑驳病毒
玉米褪绿斑驳病毒(Maize Chlorotic Mottle Virus,MCMV)是番茄从矮病毒科、玉米褪绿斑驳病毒属的正链RNA病毒[36],1974年首次在秘鲁发现。与其他感染玉米的马铃薯Y科病毒如甘蔗花叶病毒、玉米矮花叶病毒或小麦条纹花叶病毒共同感染时,会造成严重的损失。玉米致死性坏死病经常由玉米褪绿斑驳病毒和甘蔗花叶病毒共同感染造成。Gao等[26]建立了玉米中玉米褪绿斑驳病毒和甘蔗花叶病毒同时检测的RPA方法,反应温度为38℃,反应时间为30 min,检测限为102 copies/μL,灵敏度比RT-PCR方法高10倍。
邝瑞瑞等[28]也以MCMV外壳蛋白(Coat Protein,CP)基因作为靶基因,建立了RPA-LFD快速检测方法,方法特异性好,可与其他病毒很好地区分。最佳反应温度37℃,整个反应时间仅需13 min,对MCMV RNA的检测灵敏度为8.6×10-6 ng/μL,比常规RT-PCR方法灵敏度高10倍,对MCMV CP质粒DNA拷贝数的灵敏度为92.5 copies/μL,比RT-PCR方法高10倍。冯黎霞等[33]以MCMV CP基因作为目的基因,建立了玉米褪绿斑驳病毒的RPA—琼脂糖凝胶电泳快速检测方法,与传统PCR方法相比,反应时间更短。
4.3.4 玉米矮花叶病毒
玉米矮花叶病毒(Maize Dwarf Mosaic Virus,MDMV)属于马铃薯Y病毒科、马铃薯Y病毒属,病毒粒体成弯曲杆状。玉米矮花叶病毒病是一种世界性病害,一直威胁着玉米的生产。该病毒于2021年被新增至我国检疫性有害生物名录中,是我国重点关注的入侵物种。赵振兴等[29]以玉米矮花叶病毒CP基因为目的基因,建立了MDMV的RPA快速检测方法,方法反应时间为20 min,对MDMV CP阳性质粒的灵敏度可达到105 copies/μL,高于RT-PCR方法的灵敏度。
5 结语与展望
RPA技术具有反应快速、等温扩增、不依赖于昂贵的设备以及对核酸的广泛适应性等特点[37],在粮谷相关原检测中陆续得到研究和应用,特别是在现场检测方面具有广阔的发展潜力。但是,RPA方法也有不足之处,一是商用的RPA试剂盒价格较高,且试剂盒生产厂家较为单一[38];二是RPA方法一方面存在引物形成二聚体及引物错配问题,另一方面使用琼脂糖凝胶电泳观察结果存在气溶胶污染风险等;三是RPA技术在粮谷等作物相关病原的检测方法还未转化为标准,推广应用还需加强。因此,在后续的RPA技术研究中,还应继续研究开发相关试剂,进一步降低试剂生产成本,以便于RPA技术的推广应用。另外,RPA技术还应与LFD技术、CRISPR技术偶联,提高检测灵敏度,并与便携式设备、生物传感器、微流体装置等结合,开发用于现场便携、易操作的检测方法[39],为粮谷相关病原的现场检测提供技术支撑。
参考文献
[1] 罗雪琮, 安梦楠, 吴元华, 等. 重组酶聚合酶扩增技术在植物病毒检测中的应用[J]. 生物技术通报, 2022, 38(2): 269-280.
[2] Yiping ZouID, Michael Glenn Mason, Jose Ramon Botella. Evaluation and improvement of isothermal amplification methods for point-of-need plant disease diagnostics[J]. PLoS ONE, 2020, 15(6): e0235216. https://doi.org/10.1371/journal.pone.0235216.
[3] Binoy Babu, Francisco M. Ochoa-Corona, Mathews L. Paret. Recombinase polymerase amplification applied to plant virus detection and potential implications[J]. Analytical Biochemistry, 2018, 546: 72-77.
[4]施奕, 徐昌平, 余蓓蓓, 等. 重组酶聚合酶扩增技术研究进展[J]. 病毒学报, 2020, 36(3): 522-532.
[5] PiPEPENBURGO O, Williams C H, Stemple D L, et al. DNA detection using recombinase proteins[J]. Plos Biology, 2006, 4(7): e204. DOI: 10.1371/journal.pbio.0040204.
[6] Timothy D. Miles, Frank N. Martin, Michael D. Coffey. Development of Rapid Isothermal Amplification Assays for Detection of Phytophthora spp. in Plant Tissue[J]. Phytopathology, 2015, 105(2): 265-278.
[7] Craw P, Balachandran W. Isothermal nucleic acid amplification technologies for point of care diagnostics: a critical review[J]. Lab on a Chip, 2012, 12: 2469-2486.
[8] Rosser A, Rollinson D, Forrest M, et al. Isothermal recombinase polymerase amplification (RPA) of Schistosoma haematobium DNA and oligochromatographic lateral flow detection[J]. Parasites Vectors, 2015, 8: 446. DOI: 10.1186/s13071-015-1055-3.
[9] Liu L, Wan J, Geng Y, et al. Equipment-free recombinase polymerase amplification assay using body heat for visual and rapid point-of-need detection of canine parvovirus 2[J]. Molecular and Cellular Probes, 2018, 39: 41-46.
[10] Safenkova I V, Ivanov A V, Slutskaya E S, et al. Key significance of DNA-target size in lateral flow assay coupled with recombinase polymerase amplification[J]. Analytica Chimica Acta, 2020, 1102: 109-118.
[11] Boyle D S, Lehman D A, Lillis L, et al. Rapid detection of HIV-1 proviral DNA for early infant diagnosis using recombinase polymerase amplification[J]. mBio, 2013, 4(2): e00135. DOI: 10.1128/mBio.00135-13.
[12] Daher R K, Stewart G, Boissinot M, et al. Influence of sequence mismatches on the specificity of recombinase polymerase amplification technology[J]. Molecular and Cellular Probes, 2015, 29: 116-121.
[13]秦雪, 付世骞, 杨鑫焱, 等. 重组酶聚合酶等温扩增技术在食源性致病菌检测中的应用 [J]. 食品工业科技, 2021, 42(20): 449-455.
[14] Tan M, Liao C, Liang L, et al. Recent advances in recombinase polymerase amplification: Principle, advantages, disadvantages and applications[J]. Frontiers in Cellular and Infection Microbiology, 2022, 12: 1019071. DOI: 10.3389/fcimb.2022.1019071.
[15] 马文娣, 田逸英, 焦志远, 等. 重组酶聚合酶扩增技术在植物病原快速检测中的应用[J]. 植物保护, 2021, 47(3): 1-5.
[16] Euler M, Wang Y J, Heidenreich D, et al. Development of a panel of recombinase polymerase amplification assays for detection of biothreat agents[J]. Journal of Clinical Microbiology, 2013, 51(4): 1110-1117.
[17] Na-Kyeong Kim, Hyo-Jeong Lee, Sang-Min Kim, et al. Rapid and Visual Detection of Barley Yellow Dwarf Virus by Reverse Transcription Recombinase Polymerase Amplification with Lateral Flow Strips[J]. The Plant Pathology Journal, 2022, 38(2): 159-166.
[18] Gong L, Wang X, Li Z, et al. Integrated trinity test with RPA-CRISPR/Cas12a-fluorescence for real-time detection of respiratory syncytial virus a or b[J]. Front. Microbiol. 2022,13: 819931. DOI: 10.3389/fmicb.2022.819931.
[19] Qian W, Huang J, Wang X, et al. CRISPR-Cas12a combined with reverse transcription recombinase polymerase amplification for sensitive and specific detection of human norovirus genotype GII.4[J]. Virology, 2021, 564: 26-32.
[20] Ren M, Mei H, Zhou J, et al. Early diagnosis of rabies virus infection by RPA-CRISPR techniques in a rat model[J]. Archives of Virology, 2021, 166(4): 1083-1092.
[21] Xinyu Lu, Ying Zheng, Fan Zhang, et al. A Rapid, Equipment-Free Method for Detecting Phytophthora infestans in the Field Using a Lateral Flow Strip-Based Recombinase Polymerase Amplification Assay[J]. Plant Disease, 2020, 104(11): 2774-2778.
[22] Tingting Dai, Xiao Yang, Tao Hu, et al. Comparative Evaluation of a Novel Recombinase Polymerase Amplification-Lateral Flow Dipstick (RPA-LFD) Assay, LAMP, Conventional PCR, and Leaf-Disc Baiting Methods for Detection of Phytophthora sojae[J]. Frontiers in Microbiology, 2019, 10: 1884. DOI: 10.3389/fmicb.2019.01884.
[23] J Alejandro Rojas, Timothy D Miles, Michael D Coffey, et al. Development and application of qPCR and RPA genus- and species- specific detection of phytophthora sojae and P. sansomeana root pathogens of soybean[J]. Plant Disease, 2017, 101: 1171-1181.
[24] Salih Yilmaz, Ozgur Batuman. Development of a reverse transcription recombinase polymerase amplification combined with lateral flow assay for equipment-free on-site field detection of tomato chlorotic spot virus[J]. Virology Journal, 2023, 20: 136. DOI: 10.1186/s12985-023-02097-w.
[25] Wanhong Zhang, Yubing Jiao, Chengying Ding, et al. Rapid Detection of Tomato Spotted Wilt Virus With Cas13a in Tomato and Frankliniella occidentalis[J]. Frontiers in Microbiology, 2021,12: 745173. DOI: 10.3389/fmicb.2021.745173.
[26] Xinran Gao, Yuan Chen, Xuecong Luo, et al. Recombinase Polymerase Amplification Assay for Simultaneous Detection of Maize Chlorotic Mottle Virus and Sugarcane Mosaic Virus in Maize[J]. American Chemical Society omega, 2021, 6(28): 18008-18013.
[27] 陈吴健, 范宇博, 严颖鹏, 等. 大豆北方茎溃疡病菌荧光RAA检测方法的建立[J]. 浙江农业科学, 2021, 62(7): 1382- 1384+1446.
[28] 邝瑞瑞, 雷荣, 孙夕雯, 等. 基于重组酶介导等温扩增—侧流层析试纸条的玉米褪绿斑驳病毒快速检测方法[J]. 植物保护学报, 2022, 49(5): 1457-1463.
[29] 赵振兴, 范奇璇, 冯黎霞, 等. 基于重组酶聚合酶扩增技术的玉米矮花叶病毒快速检测方法的建立[J]. 植物保护, 2022, 48(6): 83-89.
[30] 袁俊杰, 龙阳, 渭婷玉, 等. 添加扩增内标的逆转录重组酶聚合酶扩增技术检测三种大豆病毒[J]. 农业生物技术学报, 2020, 28(12): 2261-2269.
[31] 单长林, 周圆, 任琰, 等. 玉米内州萎蔫病菌荧光重组酶介导等温扩增检测方法的建立[J]. 浙江农业学报, 2021, 33(9) :1676-1685.
[32]单长林, 周圆, 李孝军. 玉米细菌性枯萎病菌荧光重组酶介导等温扩增检测方法的建立与应用[J]. 广东农业科学, 2021, 48(1): 111-118.
[33] 冯黎霞, 魏霜, 余辛, 等. 重组酶聚合酶扩增技术(RPA)快速检测玉米褪绿斑驳病毒[J]. 植物保护学报, 2020, 47(1): 217-218.
[34] 鞠玉亮, 沈鹏飞, 范志朋, 等. 小麦全蚀病菌重组酶聚合酶检测方法的建立及应用[J]. 植物保护, 2020, 46(5): 150-155.
[35] 冯云丽, 储天然, 王昊, 等. 番茄斑萎病毒的发生与研究进展[J]. 中国蔬菜, 2025(6): 47-55.
[36] Redinbaugh M G, Stewart L R. Maize lethal necrosis: an emerging, synergistic viral disease[J]. Annual Review of Virology, 2018, 5: 301-322.
[37] 张娟, 徐颖, 张涛, 等. 重组酶聚合酶扩增技术在病原检测中的研究进展[J]. 中国口岸科学技术, 2025, 7(6): 20-24.
[38] 胡清霞, 李颖, 张琰杰, 等. 重组酶聚合酶扩增技术在动物疫病诊断中的应用研究进展[J]. 动物医学进展, 2023, 44(9): 82-86.
[39] 张蕾, 陈亮, 江波涛, 等. 重组酶聚合酶扩增技术在动物病原菌检测中的应用进展[J]. 动物医学进展, 2024, 45(1): 89-94.
基金项目:海关总署科研项目(2019HK028);青岛市科技惠民示范专项(25-1-5-xdny-4-nsh)
第一作者:刘培海(1980—),男,汉族,山东聊城人,硕士,高级工程师,主要从事病毒及病原菌检测工作,E-mail: liuph_513@sina.com
通信作者:雷质文(1969—),男,汉族,湖北罗田人,硕士,研究员,主要从事病原菌检测工作,E-mail: leizw@sohu.com
1. 日照海关综合技术服务中心 日照 276826
2. 青岛国际旅行卫生保健中心(青岛海关口岸门诊部) 青岛 266071
3. 青岛海关技术中心 青岛 266000
1. Comprehensive Technology Service Center of Rizhao Customs, Rizhao 276826
2. Qingdao International Travel Health Care Center (Outpatient Department of Qingdao Customs), Qingdao 266071
3. Qingdao Customs Technology Center, Qingdao 266000
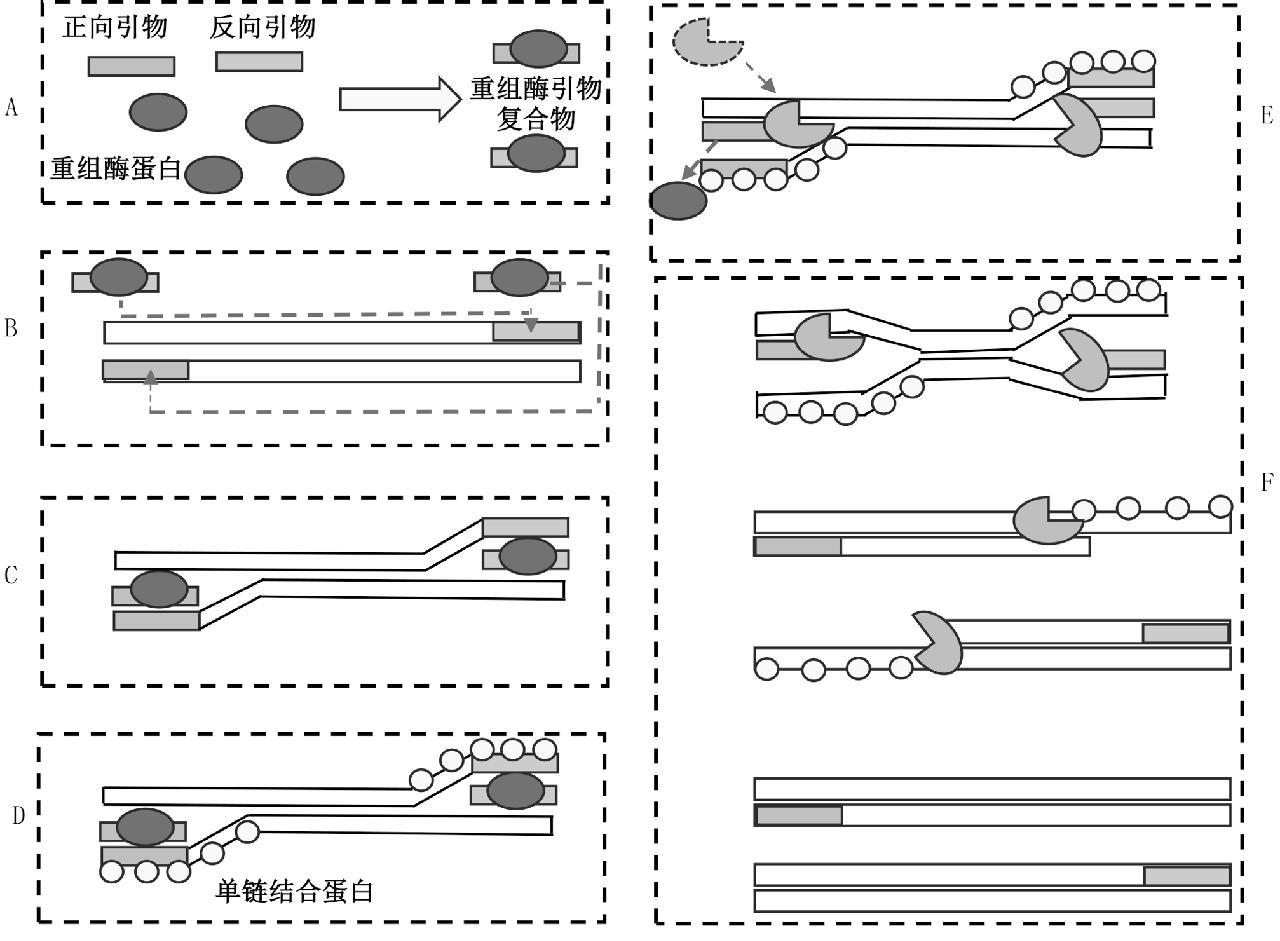
A: 重组酶—引物复合体的形成; B: 定位模板上的同源序列; C: 引发链置换反应形成D环结构; D: 单链结合蛋白稳定被置换的DNA链; E: DNA聚合酶识别重组酶解离后暴露出的引物3 '端; F: DNA扩增
图1 RPA反应原理及步骤
Fig.1 Principle and steps of recombinase polymerase amplification
表1 RPA技术与其他核酸扩增技术方法性能的比较
Table 1 Comparison of RPA and several nucleic acid amplification methods
方法 | 模板 | (条) | (种) | (min) | (℃) | 优点 | 缺点 |
PCR | DNA/RNA | 2 | 1 | 90 | 热循环 | 引物设计简单, 技术成熟, 有稳定的商业化产品 | 需要热循环扩增; 反应时间较长, 依赖于仪器设备, 容易造成交叉污染 |
LAMP | DNA/RNA | 4-6 | 1 | 60 | 65 | 特异性高, 检测方法简单, 耐受抑制剂 | 引物设计复杂; 容易出现非特异性扩增 |
(Nucleic Acid Sequence-Based Mplification, NASBA) | RNA | 2-3 | 2-3 | 180 | 37 | 对RNA分子特异性高, 不受DNA分子的干扰, 不需要cDNA处理过程 | 反应成分复杂, 需要多种酶; 不适合于DNA病毒的检测 |
链替代扩增 (Strand Displacement Amplification, SDA) | DNA | 2 | 2 | 30 | 37 | 反应条件温和, 反应快速 | 对于长片段的扩增效率较低; 非特异性反应较强, 开始阶段需要热变性 |
等温扩增 (Rolling Circle Amplification, RCA) | DNA/RNA | 3 | 3 | 60 | 37 | 指数扩增容易, 锁式探针使得反应特异性很高 | 低纯度的环状模板难以控制连接效率; 模板需要单链环状结构 |
依赖解旋酶的扩增 (Helicase-Dependent Amplification, HDA) | DNA/RNA | 2 | 2 | 90 | 65 | 反应温度恒定, 反应结构简单 | 缓冲体系优化复杂 |
RPA | DNA/RNA | 3 | 3 | 5~-20 | 37~42 | 反应时间短; 可耐受抑制剂; 引物设计简单, 可支持多重扩增反应 | 没有专门的引物设计软件; 试剂成本高, 商业化试剂盒少 |
图2 exo和fpg探针用于重组酶聚合酶扩增(RPA)产物的检测
Fig.2 Detection of RPA products with exo and fpg probe
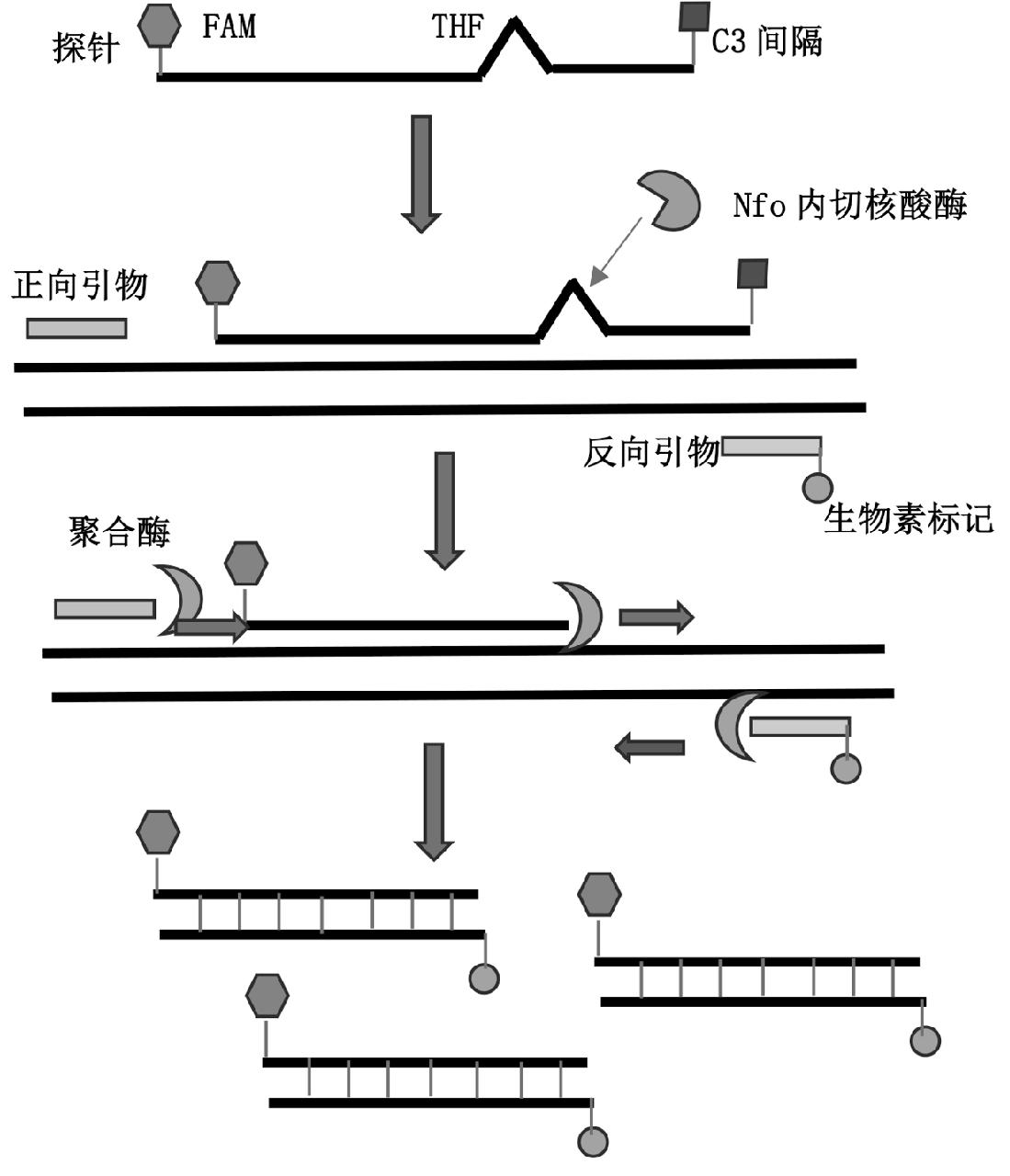
图3 LFD法检测RPA产物的原理
Fig.3 Principle of detecting RPA products using the Lateral Flow Dipstick (LFD) method
表2 国内外关于粮谷相关病原的RPA检测
Table 2 RPA detection of grain and cereal-related pathogens from domestic and international studies
病原 | 基因 | 方法 | (min) | 特异性 | 灵敏度 | 比较 | 提取方法 | 基质/宿主 | 参考文献 |
病菌 | Ypt1 | 侧向层析法 | 30 | 无交叉反应, 特异性高 | 500 fg DNA | 灵敏度比PCR方法高10倍 | PEG-NaOH法 | 马铃薯叶片 | [21] |
大豆疫霉病菌 | Ypt1 | 侧向层析法 | 25 | 与24株疫霉病菌、14株真菌无交叉反应, 特异性高 | 10 pg DNA | 灵敏度比PCR方法高100倍, 比LAMP方法高10倍 | 植物DNA提取试剂盒 | 大豆根际样品 | [22] |
白术疫霉病菌、大豆疫霉病菌 | 线粒体基因组Atp9-nad9区域 | 荧光检测 | 20 | 与100株疫霉病菌、腐霉病菌无交叉反应, 特异性高 | 1 pg DNA | 灵敏度比实时定量PCR方法低 | 植物组织粗提取方法 | 大豆根部样品 | [23] |
番茄褪绿斑驳病毒 | Nsm | 侧向层析法 | 15 | 与其他感染病毒无交叉反应 | 6 pg RNA | 比RT-PCR方法高100倍, 比RPA-琼脂糖凝胶电泳方法高10倍 | 植物组织粗提取物 | 番茄叶子 | [24] |
大麦黄矮病毒 | 包被蛋白基因 | 侧向层析法 | 20 | 与其他感染病毒无交叉反应, 特异性高 | 1 pg DNA | 灵敏度与PCR方法相当 | 总RNA提取试剂盒 | 燕麦叶子样品 | [17] |
番茄斑萎病毒 | N基因 | Cas13a系统 | 20 | 与其他感染病毒无交叉反应, 特异性高 | 226 copies | 灵敏度比RT-PCR方法高10倍 | RNA纯化试剂盒 | 西花蓟马昆虫 | [25] |
玉米褪绿斑驳病毒、甘蔗花叶病毒 | 基因组保守序列 | 琼脂糖凝胶电泳 | 30 | 与其他感染病毒无交叉反应, 特异性高 | 100 copies | 灵敏度比RT-PCR方法高10倍 | TRNzol Universal总RNA提取方法 | 玉米叶子样品 | [26] |
大豆北方茎溃疡病菌 | 基因组保守序列 | 实时荧光法 | 20 | 与其他菌株无交叉反应 | 103/mL | / | QIAGEN DNeasy Plant Mini kit | 大豆种子样品 | [27] |
玉米褪绿斑驳病毒 | 外壳蛋白基因 | 侧流层析试法 | 13 | 特异性强 | 8.6×10-6ng/μL | 灵敏度比PCR方法高10倍 | EasyPure® Plant RNA Kit RNA 提取试剂盒 | / | [28] |
玉米矮花叶病毒 | 外壳蛋白基因 | 琼脂糖凝胶电泳 | 20 | 与其他感染病毒无交叉反应, 特异性高 | 369 fg/μL | 灵敏度比PCR方法高, 是PCR方法的10倍 | 天根植物总RNA提取试剂盒 | 玉米叶片 | [29] |
大豆花叶病毒、菜豆荚斑驳病毒、南方菜豆花叶病毒 | 特异性保守序列 | 琼脂糖凝胶电泳 | 40 | 无交叉反应 | 0.05 ng | / | Tiangen (北京) 植物总 RNA 提取试剂盒 | 大豆种子 | [30] |
玉米内州萎蔫病菌 | celB | 实时荧光 | 20 | 与2株近缘亚种菌株和9株其他菌株无交叉反应, 特异性高 | 130 fg/μL | 灵敏度比LAMP方法稍低 | 细菌基因组 DNA 提取试剂盒 | 玉米种子 | [31] |
玉米细菌性枯萎病菌 | PstS-glmS | 实时荧光法 | 20 | 与3株属内其他菌株及其他8株病菌无交叉反应 | 280 fg/μL | / | 植物基因组 DNA 提取试剂盒 | 玉米种子 | [32] |
玉米褪绿斑驳病毒 | 外壳蛋白基因 | 琼脂糖凝胶电压 | 40 | 与其他感染病毒无交叉反应 | 可检测到稀释10-4倍的样品 | 灵敏度与PCR方法相当 | 植物总RNA提取试剂盒 | / | [33] |
注: “/”表示文献中未提及.







