CopyRight 2009-2020 © All Rights Reserved.版权所有: 中国海关未经授权禁止复制或建立镜像
H5N1亚型D1.1基因型禽流感病毒NS1生物信息学分析
作者:谷慧敏 李琰 谭士贵 江亚娟 窦慧馨 贾勇健 王小羽 焦伯延
谷慧敏 李琰 谭士贵 江亚娟 窦慧馨 贾勇健 王小羽 焦伯延
啜微微 1 白子龙 2 耿庆华 1 * 贾 赟 3 史喜菊 2 *
摘 要 本研究经过序列比对,选择羊口疮病毒的囊膜蛋白F1L基因和羊痘病毒P32基因作为靶基因设计引物,建立了可以同时检测羊口疮病毒和羊痘病毒的双重PCR检测方法。当样品中含有羊口疮病毒时,可以扩增出916 bp目的片段,当样品中含有羊痘病毒时,可以扩增出523 bp目的片段,如果样品中含有羊口疮病毒和羊痘病毒两种病毒时,则可以同时扩增出2条片段。研究结果表明,该方法特异性高,对于羊的常见传染病均不能扩增出目的条带。方法敏感性试验结果表明,该方法可以检测到最低浓度为4.05×10-6 ng/μL病毒核酸。对46份来自养殖场的灭活病料,利用已有标准方法和该研究方法检测,结果符合率为100%,表明该方法可以用于羊口疮病毒和羊痘病毒的检测和鉴别诊断。
关键词 羊口疮病毒;羊痘病毒;双重PCR;鉴别诊断
Establishment and Application of Dual PCR Detection Method for Distinguishing Orf Virus and Capripox Virus
CHUO Wei -W ei 1 B AI Zi -L ong 2 GEN G Qing -H ua 1* J IA Yun 3 SHI Xi -J u 2*
Abstract Following sequence alignment, the envelope protein F1L gene of Orf virus and the P32 gene of capripox virus were selected as targets for primer design, and a duplex PCR assay that simultaneously detects Orf virus and capripox virus was developed. Amplification of a 916 bp fragment indicates the presence of Orf virus, whereas a 523 bp product is indicative of capripox virus; co-infection yields both amplicons. The assay showed high specificity: no target amplicons were obtained from any of the common ovine pathogens tested. Sensitivity evaluation revealed a detection limit of 4.05×10-6 ng/μL of viral nucleic acid. Forty-six heat-inactivated field samples were examined in parallel with the duplex PCR and a validated reference method, giving 100% concordance. The established protocol is therefore suitable for detection and differential diagnosis of Orf virus and capripox virus.
Keywords orfr virus; capripox virus; duplex PCR; differential diagnosis
基金项目:“十四五”国家重点研发计划(2022YFD1800705)
第一作者:啜微微(1986—),汉族,辽宁沈阳人,本科,兽医师,主要从事动物检疫及外来物种鉴定研究工作,E-mail: 495636337@qq.com
通信作者:耿庆华(1977—),汉族,辽宁本溪人,博士,研究员,主要从事动物检疫及外来物种鉴定研究工作 ,E-mail: genghua77@163.com
共同通信作者:史喜菊(1975—),汉族,山西乡宁人,博士,研究员,主要从事动物检疫及境外动物卫生风险评估工作,E-mail: shixijubjciq@163.com
1. 沈阳海关技术中心 沈阳 110016
2. 中国海关科学技术研究中心 北京 100026
3. 大连海关技术中心 大连 116001
1. Shenyang Customs Technology Center, Shenyang 110016
2. China Customs Science and Technology Research Center, Beijing 100026
3. Dalian Customs Technology Center, Dalian 116001
羊痘又称羊天花,是由痘病毒科羊痘病毒属的羊痘病毒(Capripox Virus,CaPV)引起的一种急性、 热性、高度接触性人兽共患病。羊痘病毒包括山羊痘病毒(Goat Pox Virus,GTPV)和绵羊痘病毒(Sheep Pox Virus,SPPV)[1]。羊口疮又称羊传染性脓疱,是由痘病毒科副痘病毒属的羊口疮病毒(Orf Virus,ORFV)引起的一种嗜上皮性人兽共患病[2]。当绵羊和山羊共同饲养时,一个群体中可以同时感染2种以上的病毒。世界上许多地方的小反刍动物群体中均发生过绵羊痘、山羊痘和羊口疮的混合感染[3-5]。由于羊痘具有高死亡率和高发病率,羊口疮不仅在成年人中发病率很高,而且在羔羊中死亡率甚至高达90%[6]。由于山羊痘病毒表现出宿主特异性和发病机制的变化,可能对山羊和绵羊造成疾病威胁[7]。同样,由于ORFV的持续性和宿主免疫逃避策略,越来越多的羊口疮暴发可能对小反刍动物构成严重威胁[8]。CaPV可以混合方式同时发生在受ORFV感染的绵羊和山羊身上。CaPV和ORFV的流行情况、传播方式及临床症状极为相似,很难以临床症状表现判定感染何种病毒。因此,有必要建立一种可以同时鉴别诊断CaPV和ORFV的检测方法。
羊口疮病毒主要关键基因包括B2L基因、F1L基因、VEGF基因和VIR等基因家族,其中F1L基因为高度保守基因,在病毒流行过程中不易发生变异,适合用于建立诊断方法[9]。羊痘病毒的基因组主要包含P32基因、RING finger基因和G 蛋白偶联趋化因子受体基因等。其中,P32基因是羊痘病毒中相对保守的基因,常被用于病毒检测和分类鉴定[10]。本研究针对羊口疮病毒的囊膜蛋白F1L基因和羊痘病毒P32基因设计了一套引物组合,建立了可以同时检测CaPV和ORFV的双重PCR方法,该方法可以检测CaPV和ORFV单独感染或混合感染,可为临床工作提供技术支撑。
1 材料与方法
1.1 材料
1.1.1 主要试剂
PCR Mix(2×Taq,大连宝生物公司);DNA Marker(100 bp Lader,大连宝生物公司);DNA Marker(DL2000,大连宝生物公司);核酸提取试剂(MVN02,上海之江生物科技股份有限公司)。
1.1.2 毒株、病料及核酸
羊口疮细胞灭活毒、山羊痘细胞灭活毒、绵羊痘细胞灭活毒、接种羊口疮病毒和山羊痘病毒的细胞混合液(经灭活)、小反刍兽疫病毒核酸、蓝舌病毒核酸由中国动物卫生与流行病学中心国家外来动物疫病监测与研究中心馈赠。羊败血性链球菌病灭活疫苗和山羊传染性胸膜肺炎灭活疫苗购自哈药集团生物疫苗有限公司;口蹄疫O型/A型二价疫苗购自天康生物股份有限公司。布氏杆菌病活疫苗购自重庆澳龙生物制品有限公司。经鉴定含有羊口疮病毒、山羊痘病毒和绵羊痘病毒的绵羊和山羊的鼻拭子、羊毛和羊皮等经灭活的病料,共46份,分别来源于5家养殖场。
1.2 方法
1.2.1 引物合成
从GenBank中分别获得ORFV和CaPV的全基因序列,经过比较分析,用Oligo 6设计ORFV和CaPV特异性引物,扩增ORFV引物O1和O2,可扩增片段916 bp,扩增CaPV引物C1和C2,可扩增523 bp。具体序列见表1。
1.2.2 病毒核酸的制备
本实验中所使用的核酸样本由利用全自动核酸提取仪(EX3600,上海之江生物科技股份有限公司),按照核酸提取试剂说明书提取。
1.2.3 ORFV 和CaPV的普通 PCR方法建立
(1)ORFV的普通PCR方法建立。采用25 μL的PCR反应体系,2×Taq PCR Mix 12.5 μL、引物O1、O2浓度分别选取5 μM/L、10 μM/L和20 μM/L的各1 μL进行条件筛选、去离子水补足到20 μL,混匀后瞬时离心,分装到各反应孔中;每个反应孔加入对应的核酸模板5 μL;PCR循环参数为:94℃ 预变性2 min, 95℃ 30 s,退火温度分别选取56℃、58℃和60℃,退火时间选取30 s、45s和60 s进行条件筛选,72℃ 20 s,40个循环后,72℃延伸5 min。PCR反应后,取5 μL PCR产物进行20 g/L琼脂糖凝胶电泳。
表1 用于多重PCR的引物信息
Table 1 Primer information for multiplex
PCR引物 | 序列 (5 ' ~3 ' ) | 片段长度 (bp) |
O1 | CAGGTATGCCAGGATGAAGATGT | 916 |
O2 | CGAAATCACGGCCTACATAATCG | |
C1 | AAATCATATCCCCCTGTGTACGA | 523 |
C2 | ATGTTATACCAATCGTTGGTCGC |
(2)CaPV的普通 PCR方法建立。采用25 μL的PCR反应体系,2×Taq PCR Mix 12.5 μL,引物C1、C2浓度分别选取5 μM/L、10 μM/L和20 μM/L的各1 μL进行条件筛选、去离子水补足到20 μL,混匀后瞬时离心,分装到各反应孔中;每个反应孔加入对应的核酸模板5 μL;PCR循环参数为:94℃ 预变性2 min,95℃ 30 s,退火温度分别选取56℃、 58℃和60℃,退火时间选取30 s、45 s和60 s进行条件筛选,72℃ 20 s,40个循环后,72℃延伸5 min。PCR反应后,取5 μL PCR产物进行20 g/L琼脂糖凝胶电泳。
(3)PCR扩增产物的鉴定。ORFV 和CaPV PCR扩增产物送生工生物工程(上海)股份有限公司进行序列测定。
1.2.4 ORFV和CaPV双重PCR方法的建立及产物鉴定
取羊口疮细胞毒、山羊痘细胞毒、接种羊口疮病毒和山羊痘病毒的细胞混合液和接种羊口疮病毒和绵羊痘病毒的细胞混合液,按照1.2.2方法提取核酸后进行PCR反应,2×Taq PCR Mix 12.5 μL、5 μM/L的引物01、02、C1和C2各1 μL,去离子水补足到20 μL,混匀后瞬时离心,分装到各反应孔中;以提取的上述核酸为模板,每个反应孔加入对应的核酸模板5 μL进行双重PCR反应,并对反应条件进行摸索;反应后,取5 μL PCR产物进行20 g/L琼脂糖凝胶电泳。将扩增产物送生工生物工程(上海)股份有限公司进行序列测定。
1.2.5 特异性检测
提取羊口疮细胞毒、山羊痘细胞毒、绵羊痘细胞毒、羊败血性链球菌、山羊传染性胸膜肺炎灭活疫苗、羊败血性链球菌病灭活疫苗、接种口蹄疫O型/A型二价疫苗、布氏杆菌病活疫苗、接种羊口疮病毒和山羊痘病毒的细胞混合液以及小反刍兽疫核酸、蓝舌病病毒核酸,用建立的双重PCR方法进行扩增。
1.2.6 敏感性试验
提取ORFV病毒和CaPV细胞毒混合物的核酸和正常MDCK细胞的核酸,提取后用分光光度计测定核酸浓度,将ORFV病毒取5 μL核酸加入到45 μL蒸馏水中,再从中取出5 μL进行倍比稀释,依次为10-1 ng/μL、10-2 ng/μL、10-3 ng/μL、10-4 ng/μL、10-5 ng/μL、10-6 ng/μL、10-7 ng/μL、10-8 ng/μL。分别用建立的双重PCR方法进行扩增。
1.2.7 双重PCR的初步应用及比对试验
对46份临床疑似病料样品用NY/T 3235—2018《羊传染性脓疱诊断技术》和NY/T 576—2015《绵羊痘和山羊痘诊断技术》的标准方法以及本研究建立的ORFV和CaPV双重PCR扩增方法同时进行检测并进行比对。
2 结果
2.1 ORFV的普通PCR方法扩增结果
经对PCR反应过程中的引物浓度、退火温度和时间等条件的优化,从扩增的特异性和扩增效率方面考虑选择最佳反应条件,确定最终反应条件:2×Taq PCR Mix 12.5 μL,上、下引物O1和O2浓度为5 μM/L 各1 μL,核酸模板5 μL,去离子水补足到25 μL; PCR循环参数为:94℃ 预变性2 min, 95℃ 30 s,56℃ 30 s,72℃ 20 s,40个循环后,72℃延伸5 min。反应产物扩增出特异性条带916 bp,与预期结果相符(图1)。
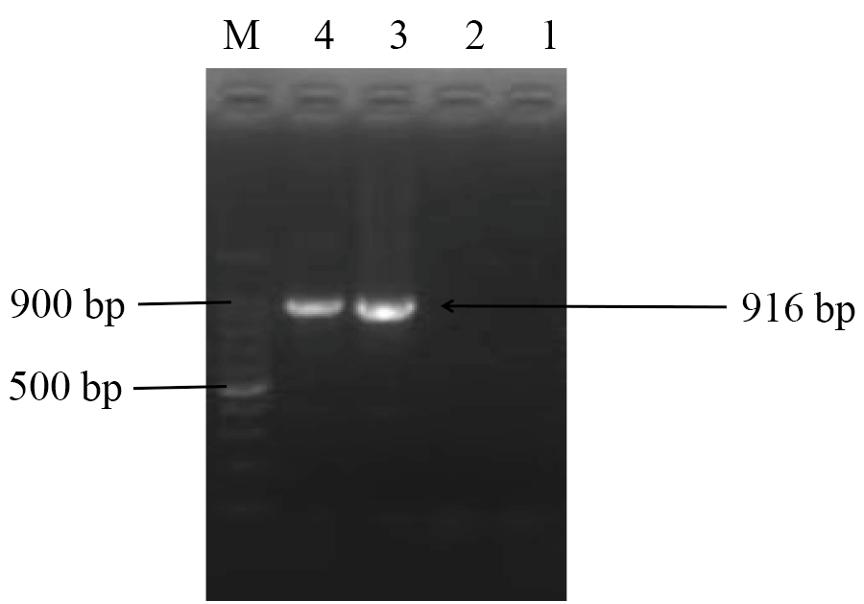
1: 水对照; 2: 健康羊毛对照; 3: 羊口疮病毒; 4: 感染羊口疮病毒的病料; M: DNA 标准 100 bp Lader
图1 ORFV的PCR扩增结果
Fig.1 ORFV PCR amplification results
2.2 CaPV的普通PCR方法扩增结果
经对PCR反应过程中的引物浓度、退火温度和时间等条件的优化,从扩增的特异性和扩增效率方面考虑选择最佳反应条件,确定最终反应条件:2×Taq PCR Mix 12.5 μL,上、下引物C1和C2浓度为5 μM/L 各1 μL,核酸模板5 μL,去离子水补足至25 μL;PCR循环参数为:94℃ 预变性2 min,95℃ 30 s,56℃ 或58℃ 30s,72℃ 20 s,40个循环后,72℃延伸5 min。反应产物扩增出特异性条带523 bp,与预期结果相符(图2)。
2.3 PCR产物的鉴定
ORFV和CaPV的 PCR扩增产物送生工生物工程(上海)股份有限公司进行序列测定。结果与GenBank发表的基因序列同源性均在99%以上。
2.4 ORFV和CaPV双重PCR扩增结果及产物鉴定
经对PCR反应过程中的退火温度、延伸时间、循环次数等条件的优化,确定最佳反应条件。反应体系为:2×Taq PCR Mix 12.5 μL,5 μM/L ORFV 和CaPV 的上、下引物O1、O2、C1和C2各1 μL,核酸模板5 μL,去离子水补足到25 μL;PCR循环参数为:94℃ 预变性2 min,95℃ 30 s,56℃ 30 s,72℃ 20 s,40个循环后,72℃延伸5 min。反应产物电泳结果如图3所示。双重PCR产物电泳后,扩增产物送生工生物工程(上海)股份有限公司进行序列测定,扩增的2个条带测序结果与GenBank上发表的 ORFV和CaPV的基因序列同源性均在99%以上。
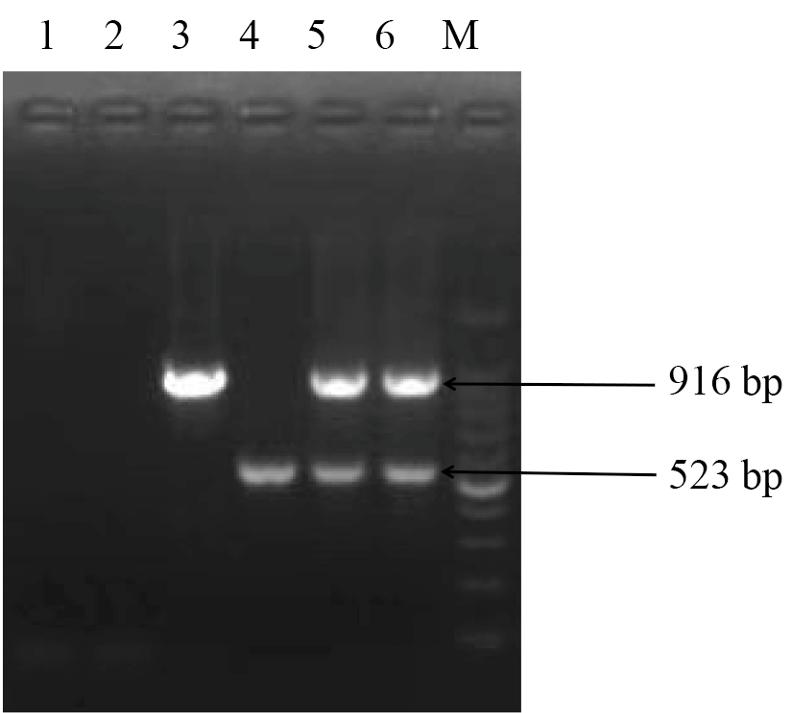
1: 水对照; 2: 健康羊毛对照; 3: 羊口疮病毒; 4: 山羊痘病毒; 5: 羊口疮病毒和山羊痘病毒; 6: 羊口疮病毒和绵羊痘病毒; M: DNA 标准 100 bp Lader
图3 ORFV和CaPV双重PCR扩增结果
Fig.3 Duplex PCR amplification results of ORFV and CaPV
2.5 特异性试验结果
特异性试验结果如图4所示,接种羊口疮病毒和山羊痘病毒或绵羊痘病毒的细胞混合液可以扩增出916 bp和523 bp的两条目的条带,接种ORFV的MDCK细胞只能扩增916 bp的条带,接种GTPV的MDCK细胞和接种SPPV的MDCK细胞只能扩增出523 bp的CaPV条带,其余接种的MDCK细胞和正常MDCK细胞中未出现任何条带。
2.6 敏感性试验结果
提取ORFV病毒和CaPV细胞毒混合物的核酸和正常MDCK细胞的核酸,提取后用分光光度计测定核酸浓度(4.05 ng/μL),将ORFV病毒取5 μL核酸加入到45 μL蒸馏水中,再从中取出5 μL进行倍比稀释,依次为10-1 ng/μL、10-2 ng/μL、10-3 ng/μL、10-4 ng/μL、10-5 ng/μL、10-6 ng/μL、10-7 ng/μL、10-8 ng/μL。分别用建立的双重PCR方法进行扩增,结果4.05×10-1~10-6 ng/μL的ORFV病毒和CaPV细胞毒混合物能扩增916 bp的ORFV条带和523 bp的CaPV条带、4.05×10-7~10-8 ng/μL的ORFV病毒和CaPV细胞毒混合物未能扩增可见条带,正常MDCK细胞中未出现任何条带,如图5所示。
2.7 双重PCR的初步应用及比对试验
46份临床疑似病料样品已用NY/T 3235—2018和NY/T 576—2015方法以及本专利研发的PCR扩增方法同时进行检测,结果见表2。试验结果表明:本研究建立的ORFV和CaPV的PCR检测方法检测结果与已有标准方法检测ORFV和CaPV结果完全符合。且其中8份ORFV阳性样品中同时扩增出了ORFV和CaPV,经测序与发表的ORFV和CaPV基因序列同源性为99.2%,表明该样品为两种病毒混合感染。与已有的病毒分离鉴定方法(“金标准”方法)检测结果一致。然而,传统的病毒分离鉴定方法检测耗时较长,而且对设施环境以及检测人员的能力要求很高,而本研究中的PCR方法则适合基层实验室的检测工作,检测人员经过简单培训即可操作。
3 讨论
据研究,在不同地区山羊养殖场中,ORFV感染羊群的现象普遍存在,羔羊发病率高达80%[11]。虽然羊群感染ORFV后死亡率较低,但感染该病的羊群食欲严重下降,病程的后期食欲废绝,进而影响其生长发育,免疫力降低。羊口疮和羊痘是目前国内外养殖中危害性较为严重的2种羊的传染病。由于这些疫病的发病表现近似,均以皮肤、黏膜产生疱疹、溃疡为相似临床症状,且常发生混合感染现象,临床上常难以进行快速、较准确而有效的鉴别诊断。两者虽然发病率都很高,但是羊痘的死亡率更高。因此,建立一种既快速,相对更准确、灵敏度高的病原联合共检方法,又可以同时鉴别这两种病的方法,对于疫病的快速诊检和采取有针对性的防控措施具有重要意义。
本研究选择羊口疮病毒的囊膜蛋白F1L基因是羊口疮病毒比较保守的基因,P32蛋白是羊痘病毒膜上的一个蛋白,由于该基因的高度保守性,因而常作为GTPV检测的理想基因[12]。经对双重PCR反应过程中的引物浓度、退火温度和退火时间等条件进行筛选。结果显示引物浓度在5 μM/L、10 μM/L和20 μM/L时均有特异性产物扩增,而在10 μM/L和20 μM/L时会偶有非特异性条带扩增,故选择引物浓度5 μM/L为最佳反应条件。ORFV在退火温度为56℃时,扩增的特异性和扩增效率最佳;而GTPV在退火温度为56℃或58℃时,扩增的特异性和扩增效率最佳;因而双重PCR最佳反应条件选择退火温度为56℃;退火时间选取30 s、45 s和60 s时均有很好的扩增效果,从减少检测时间、缩短检测时限方面考虑,选择了30 s作为最佳退火时间。
对于羊常见的传染病病原核酸进行特异性研究,结果表明含有小反刍兽疫病毒核酸、蓝舌病毒核酸、O型/A型口蹄疫病毒核酸、山羊传染性胸膜肺炎病原核酸、羊败血性链球菌病原核酸、布氏杆菌核酸均不能扩增出目的条带。利用含有ORFV和GTPV的细胞病毒进行敏感性研究,结果本研究建立的方法对4.05×10-6 ng/μL的核酸均有扩增结果,高于王玲玲等[13]建立的双重RPA-LFD检测方法1.63×10-5 ng/μL核酸的最低检出限。同时,在对养殖场实际采集的样品进行检测发现,与已有的标准方法相比较,检测结果符合率为100%。综上可以看出本研究建立的检测方法不仅操作简单,而且特异性强、敏感性较高,使用的检测设备廉价,适用于基层实验室以及大型养殖场对于羊口疮病毒和羊痘病毒的检测和鉴别诊断,为养殖场疫情的早期诊断和鉴别以及制定有针对性的防控措施提供技术保障。
参考文献
[1]黄学圣, 陈轶霞, 赵诗佳, 等. 羊痘病毒研究综述[J]. 甘肃畜牧兽医, 2020, 50(5): 11-14.
[2] Khalafalla A I, El-Sabagh I M, Al-Busada K A, et al. Phylogenetic analysis of eight Sudanese camel contagious ecthyma viruses based on B2L gene sequence[J]. Virology Journal. 2015, 12: 124.
[3] V. Bhanuprakash, M. Hosamani, R.K. Singh. Prospects of control and eradication of capripox from the Indian subcontinent: a perspective[J]. Antiviral Research 2011, 91(3): 225-232.
[4] Abdelfattah Selim, Mahmoud Elhaig, Jennifer Höche, et al. Molecular detection and analysis of Sheeppox and Orf viruses isolated from sheep from Qalubia, Egypt[J]. Berliner und Munchener Tierarztliche Wochenschrift, 2016, 129(7-8): 310-317.
[5] P. M. Beard, S. Sugar, E. Bazarragcha, et al. A description of two outbreaks of capripoxvirus disease in Mongolia[J]. Veterinary Microbiology, 2010, 142(3): 427-431.
[6] Ramakrishnan M A, Santhamani R, Pandey AB. Capripox outbreak in a mixed flock of sheep and goats in India[J]. Transboundary and Emerging Diseases. 2017, 64(1): 27-30.
[7] S. Babiuk, T R Bowden, D B Boyle, et al. Capripox viruses: an emerging worldwide threat to sheep, goats and cattle[J]. Transboundary and Emerging Diseases, 2008, 55(7): 263-272.
[8] Demiraslan H, Dinc G, Doganay M. An Overwiev of ORF Virus Infection in Humans and Animals[J]. Insulin resistance and Diabetes in HIV infection, 2017: 12(1): 21-30.
[9]张孟然, 丛潇, 顾月月, 等. 羊口疮病毒主要基因组编码蛋白及其功能[J]. 动物医学进展, 2022, 43(4): 96-10.
[10]陈轶霞.羊痘病毒基因组及其功能研究概况[J].中国兽医学报, 2015, 2(35): 340-344.
[11]毛从剑, 张兴民, 黄忍, 等. 口疮病毒四川流行株的分离鉴定及其遗传进化分析[J]. 中国兽医科学, 2019(7): 871-878.
[12]田启会, 祁光宇, 杨织瑞, 等. 山羊痘病毒P32基因的克隆及毕赤酵母中的表达[J]. 中国动物传染病学报, 2015, 23(6): 31-36.
[13]王玲玲, 马爱红, 宋前进, 等. 羊痘病毒与羊口疮病毒双重RPA-LFD检测方法的建立与应用[J]. 生物技术通报, 2024, 40(5): 103-111.
1: 水对照; 2: 健康羊毛对照; 3: 山羊痘细胞毒; 4: 绵羊痘细胞毒; 5: 接种绵羊痘病毒和山羊痘病毒的细胞混合液; m: DL2000
图2 CaPV的 PCR扩增结果
Fig.2 CaPV PCR amplification results
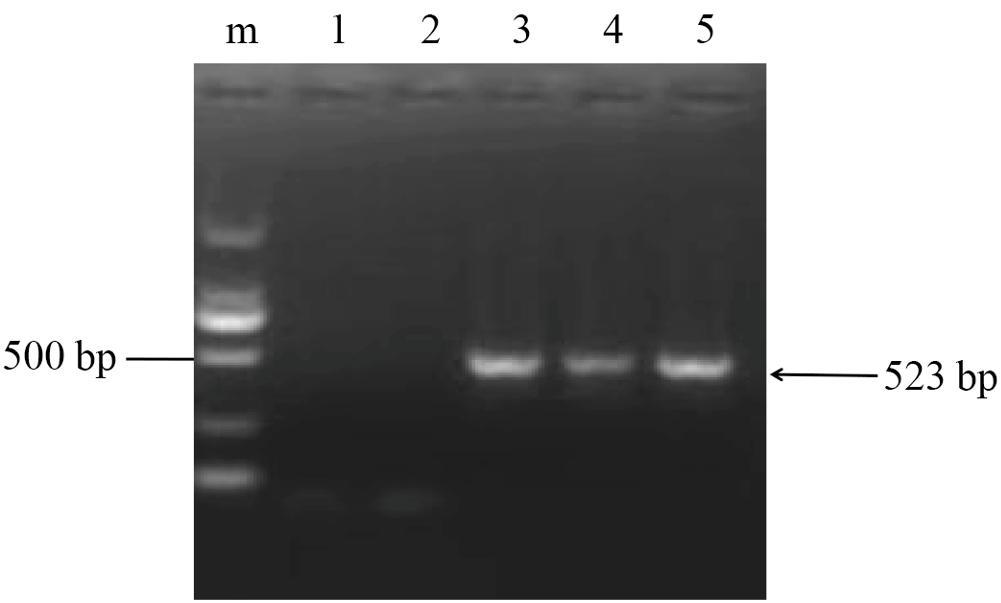
表2 双重PCR方法与已有标准方法检测结果比对
Table 2 Comparison of detection results between duplex PCR and validated reference method
病毒 | 已有标准方法 | 双重PCR方法 | |||
病料数量 | 阳性数 | 病料数量 | 阳性数 | ||
ORFV | 46 | 33 | 46 | 33 | |
CaPV | 46 | 21 | 46 | 21 | |
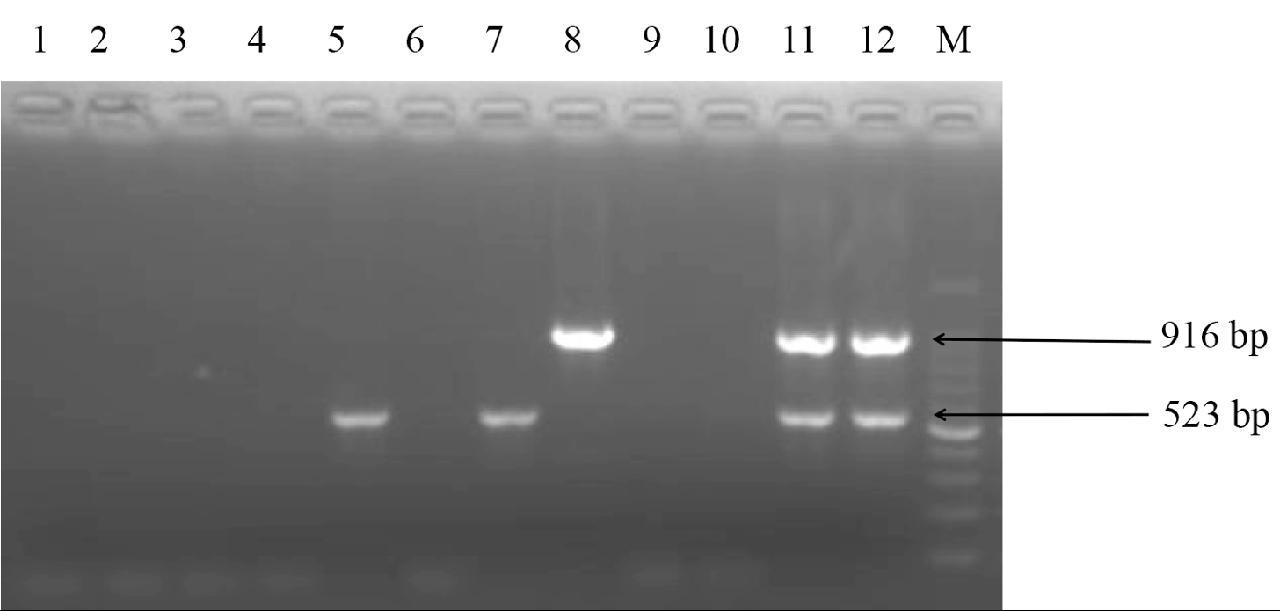
1: 水对照; 2: 羊败血性链球菌病灭活疫苗; 3: 山羊传染性胸膜肺炎灭活疫苗; 4: 羊败血性链球菌病灭活疫苗; 5: 山羊痘病毒; 6: 口蹄疫O型/A型二价疫苗; 7: 绵羊痘病毒; 8: 羊口疮病毒; 9: 小反刍兽疫病毒核酸; 10:蓝舌病毒核酸; 11: 羊口疮病毒和山羊痘病毒细胞混合液; 12: 羊口疮病毒和绵羊痘病毒细胞混合液; M: DNA 标准 100 bp Lader
图4 特异性试验结果
Fig.4 Specific test results
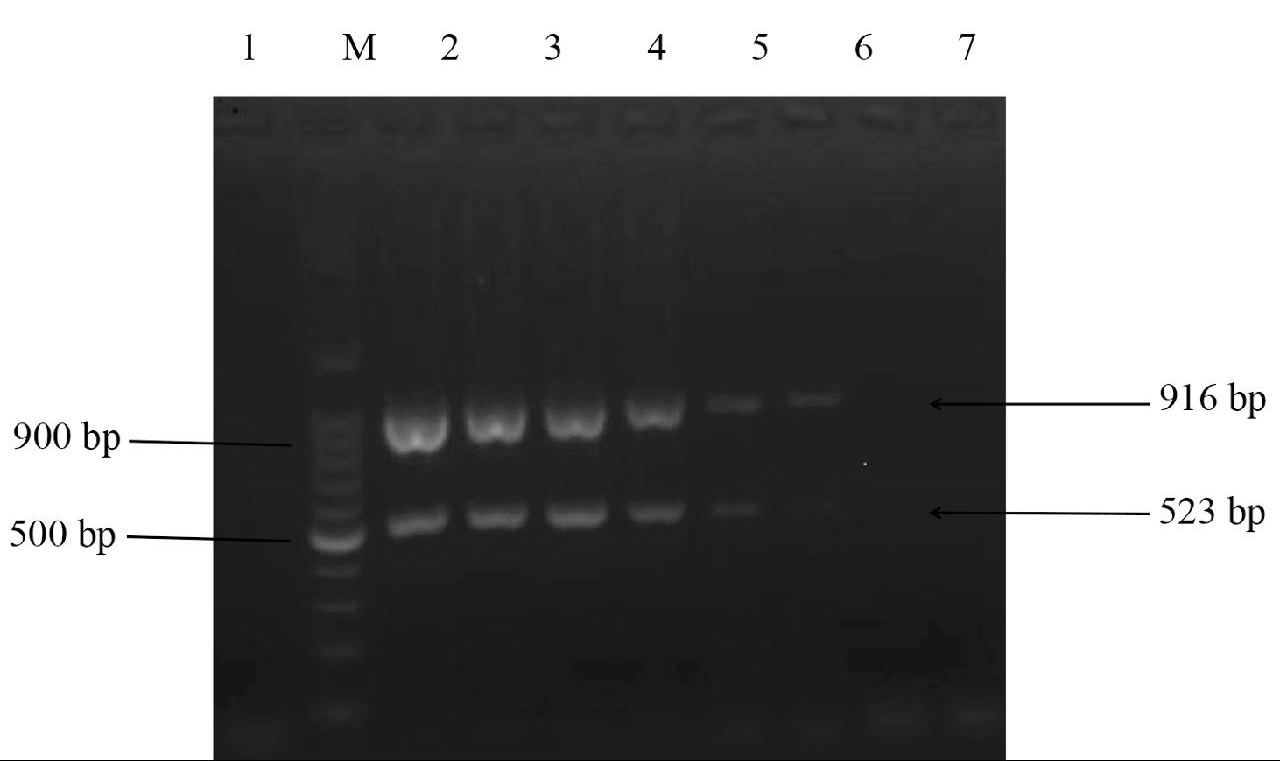
1: 水对照; 2 ~9: 提取羊口疮病毒和山羊痘病毒核酸10-1 ~10-8倍稀释; M: DNA 标准 100 bp Lader
图5 敏感性试验结果
Fig.5 Sensitivity test results
第7卷 第11期
2025年11月
生物技术 / Biotechnology
H5N1亚型D1.1基因型禽流感病毒NS1
生物信息学分析
谷慧敏 1 李 琰 2 谭士贵 2 江亚娟 2 窦慧馨 2 贾勇健 2 王小羽 2 * 焦伯延 2 *
摘 要 本研究利用生物信息学方法分析H5N1亚型D1.1基因型禽流感病毒非结构蛋白1(Non-Structural Protein 1,NS1)的生物学特征。在GISAID数据库获取D1.1基因型禽流感病毒NS1序列,利用生物信息学软件对NS1进化特点、理化性质、亲/疏水性、亚细胞定位、翻译后修饰位点、三级结构、配体结合位点、与基因组RNA结合、抗原表位等进行分析。结果显示,来源于人和奶牛的D1.1基因型禽流感病毒NS基因核苷酸相似性为99.16%~100%,NS1氨基酸相似性为98.68%~100%;其中,来源于人和奶牛的NS1高度同源。进一步分析显示,NS1含230个氨基酸,分子量25,991.83,属于亲水性蛋白,主要定位于细胞质、细胞核和线粒体;NS1含有15个磷酸化位点、1个N糖基化位点和1个O糖基化位点;NS1的配体结合位点含有9个氨基酸;N端的1—73位氨基酸能够结合病毒基因组RNA。另外,NS1含有6个辅助性T细胞抗原表位、3个杀伤性T细胞抗原表位和8个B细胞抗原表位。结果表明,D1.1基因型禽流感病毒NS1含有丰富的蛋白翻译后修饰位点、抗原表位、配体结合位点,本研究结果为D1.1基因型禽流感的防控提供了数据参考。
关键词 生物信息学;D1.1基因型;禽流感病毒;非结构蛋白1
Bioinformatics Analysis of Non-Structural Protein 1 of D1.1 Genotype of H5N1 Avian Influenza Virus
GU Hui-Min1 LI Yan2 TAN Shi-Gui2 JIANG Ya-Juan2
DOU Hui-Xin2 JIA Yong-Jian2 WANG Xiao-Yu2* JIAO Bo-Yan2*
Abstract This study employed bioinformatics approaches to analyze the biological characteristics of the Non-Structural Protein 1 (NS1) of genotype D1.1of the H5N1 subtype avian influenza virus. NS1 sequences of D1.1 genotype avian influenza viruses were obtained from the GISAID database. Bioinformatics software was used to analyze the evolutionary traits, physicochemical properties, hydrophilicity/hydrophobicity, subcellular localization, post-translational modification sites, tertiary structure, ligand-binding sites, genomic RNA-binding ability, and antigenic epitopes of NS1. Results showed that the nucleotide similarity of the NS gene in D1.1 genotype avian influenza viruses derived from humans and dairy cows ranged from 99.16% to 100%, while the amino acid similarity of NS1 ranged from 98.68% to 100%. NS1 proteins from human and bovine isolates exhibited high homology. NS1 consists of 230 amino acids with a molecular weight of 25,991.83 Da and is a hydrophilic protein primarily localized in the cytoplasm, nucleus, and mitochondria. It contains 15 phosphorylation sites, 1 N-glycosylation site, and 1 O-glycosylation site. The ligand-binding site comprises 9 amino acids, while the N-terminal region of amino acids 1–73 can bind viral genomic RNA. Additionally, NS1 harbors 6 helper T-cell epitopes, 3 cytotoxic T-cell epitopes, and 8 B-cell epitopes. The NS1 protein of D1.1 genotype avian influenza virus contains abundant post-translational modification sites, antigenic epitopes, and ligand-binding sites, providing valuable data for the prevention and control of D1.1 genotype avian influenza.
Keywords bioinformatics; D1.1 genotype; avian influenza virus; non-structural protein 1
基金项目:山东省医药卫生科技发展计划项目(202112060725,202412060875);济宁市重点研发计划(2023YXNS026)
第一作者:谷慧敏(1982—),女,汉族,山东济宁人,研究生,副主任技师,主要从事微生物检验工作,E-mail: 350972139@qq.com
通信作者:焦伯延(1983—),男,汉族,山东济宁人,博士,副主任技师,主要从事微生物检验工作,E-mail: j198319831983@126.com
共同通信作者:王小羽(1995—),女,汉族,山东济宁人,研究生,主管技师,主要从事微生物检验工作,E-mail: wangxiaomao 5236@163.com
1. 济宁市爱国卫生和健康促进中心 济宁 272000
2. 济宁市疾病预防控制中心 济宁 272000
1. Jining Patriotic Health Campaign and Health Promotion Center, Jining 272000
2. Jining Center for Disease Control and Prevention, Jining 272000
禽流感是甲型流感病毒引起的一类禽类传染病,部分亚型的禽流感病毒如H5N1亚型、H7N9亚型、H9N2亚型等可感染人类,引起人感染禽流感,是一类严重威胁人类生命健康的传染病[1-3]。H5N1亚型禽流感病毒具有极强的致病性,能够引起患者重症和死亡。H5N1亚型禽流感病毒2.3.4.4进化分支最早出现在2008年,2.3.4.4进化分支演化出的2.3.4.4b进化分支2016年通过候鸟在全球扩散[4],2021年底传入北美,随后感染了奶牛、狐狸、熊、猫等多种哺乳动物,并发生了多起人感染H5N1亚型禽流感病毒事件[5-6]。
D1.1基因型是H5N1亚型禽流感病毒2.3.4.4b进化分支的一种新的基因型,全球流感数据共享倡议组织数据库(Global Initiative on Sharing All Influenza Data,GISAID)收录的最早的人感染D1.1基因型禽流感病毒是2024年10月,2025年1月在奶牛中首次发现D1.1基因型禽流感病毒[7]。D1.1基因型禽流感病毒可能对哺乳动物的适应性高于2.3.4.4b进化分支其它基因型。截至2025年11月6日,D1.1基因型禽流感病毒已在美国、加拿大等地区的奶牛和人病例中检出,并成为北美迁徙野鸟中的主要毒株之一。
全球有9条主要候鸟迁徙路线,其中北美和中国均位于西太平洋迁徙路线和东亚—澳大利西亚迁徙路线,虽然我国目前暂未有D1.1基因型禽流感病毒的报道,但仍需提高警惕,防患于未然。目前对D1.1基因型禽流感病毒重要蛋白的结构和功能研究较少,流感病毒NS基因编码的NS1蛋白具有广泛的生物学特征,为了解D1.1基因型禽流感病毒NS1蛋白特征,本研究利用生物信息学方法分析D1.1基因型禽流感病毒NS1的进化变异、理化性质、蛋白结构和抗原表位,为深入了解D1.1基因型禽流感病毒研究提供科学依据。
1 材料和方法
1.1 数据来源
2025年11月6日在GISAID数据库查询D1.1基因型禽流感病毒基因序列数量,并下载人感染和奶牛感染D1.1基因型H5N1亚型禽流感病毒NS基因序列和NS1氨基酸序列各52条,2.3.4.4b进化分支A1基因型NS基因序列和NS1氨基酸序列各11条。其中,人感染D1.1基因型H5N1亚型禽流感病毒A/Washington/240/2024的NS基因序列号为EPI3630043,A/Washington/240/2024的NP基因序列号为EPI3630042。
1.2 分析方法
利用MEGA v7.0.14软件分析NS基因核苷酸相似性、NS1氨基酸相似性和构建进化树,利用ProtParam在线软件(https://web.expasy.org/protparam/)分析NS1的理化性质,利用ProtScale在线软件(https://web.expasy.org/protscale/)分析NS1的亲/疏水性。
利用PSORT Ⅱ server在线软件(https://www.genscript.com/tools/psort)分析NS1的亚细胞定位,利用cNLS Mapper软件(https://nls-mapper.iab.keio.ac.jp/cgi-bin/NLS_Mapper_form.cgi)分析核定位信号,利用TMHMM-2.0在线软件(https://services.healthtech.dtu.dk/services/TMHMM-2.0/)分析NS1的跨膜结构,利用NetPhos-3.1软件(https://services.healthtech.dtu.dk/services/NetPhos-3.1/)分析NS1磷酸化位点,利用NetNGlyc-1.0软件(https://services.healthtech.dtu.dk/services/NetNGlyc-1.0/)分析N糖基化位点,利用NetOGlyc-4.0软件(https://services.healthtech.dtu.dk/services/NetOGlyc-4.0/)分析O糖基化位点,利用GPS-lipid软件分析(http://lipid.biocuckoo.org/webserver.php)棕榈酰化、N-肉豆蔻酰化、法尼基化和香叶基香叶酰化位点。
利用HelixFold3软件(https://paddlehelix.baidu.com/?redirect=console)构建NS1的三级结构、NS1 1~73位氨基酸三级结构、流感病毒NP基因组RNA三级结构,利用PrankWeb软件(https://prankweb.cz/)对NS1的三级结构进行配体结合位点分析。
利用RPISeq 软件(http://pridb.gdcb.iastate.edu/RPISeq/)进行NS1 1~73位氨基酸与NP基因组结合分析。
利用NetMHCIIpan-4.0软件(https://services.healthtech.dtu.dk/services/NetMHCIIpan-4.0/)分析辅助性T细胞抗原表位,利用NetCTL-1.2 Server(https://services.healthtech.dtu.dk/services/NetCTL-1.2/)软件分析NS1的杀伤性T细胞抗原表位,利用SVMTriP软件(http://sysbio.unl.edu/SVMTriP)分析NS1的B细胞抗原表位。
2 结果
2.1 D1.1基因型禽流感病毒NS基因和NS1进化分析
截至2025年11月6日,GISAID数据库共收录2610条D1.1基因型禽流感病毒基因组序列,其中,人感染D1.1基因型禽流感病毒序列10条,奶牛感染D1.1基因型禽流感病毒序列42条,52条来源于人和奶牛的序列NS基因核苷酸相似性为99.16%~100%,平均遗传距离为0.0031;NS1氨基酸相似性为98.68%~100%,平均遗传距离为0.0005,如图1所示。利用MEGA软件构建NS基因和NS1进化树,人、奶牛的NS基因序列和NS1序列均高度同源,均位于D1.1进化分支,如图2和图3所示。
图1 NS基因核苷酸相似性和NS1氨基酸相似性分析
Fig.1 Analysis of nucleotide similarity of NS gene and amino acid similarity of NS1 protein
2.2 NS1的理化性质、亲/疏水性分析
以来源于人的D1.1基因型禽流感病毒A/Washington/240/2024作为研究序列,利用ProtParam软件分析NS1的理化性质,结果显示NS1含有230个氨基酸,分子式C1141H1846N324O346S11,分子量25,991.83,理论等电点5.60,含带正电荷氨基酸(Arg+Lys)31个,含带副电荷氨基酸(Asp+Glu)34个。半衰期30 h,不稳定系数61.85,脂肪指数为89.04,亲水性平均值为-0.343。NS1含有20种氨基酸,含量最多的是亮氨酸(Leu)和精氨酸(Arg),含量最少的是酪氨酸(Tyr)和半胱氨酸(Cys),见表1。
利用ProtScale软件分析NS1的亲/疏水性质,结果显示NS1最强的亲水氨基酸是第37位的Arg,亲水值为-2.77,最强的疏水氨基酸是第180位的缬氨酸(Val),疏水值为2.38。NS1总亲水值为-94.04,总疏水值为58.77,NS1是亲水性蛋白,如图4所示。
2.3 NS1的亚细胞定位和跨膜结构分析
利用PSORT Ⅱserver软件分析NS1的亚细胞定位,结果显示56.5%的NS1分布在细胞质中,30.4%的NS1分布在细胞核,13.0%的NS1分布在线粒体中。利用cNLS Mapper 软件分析核定位信号发现,NS1含有2个核定位信号,分别是第16位氨基酸到第46位氨基酸和215~225位氨基酸。利用TMHMM-2.0软件分析NS1的跨膜结构,结果显示NS1不含有跨膜结构域,属于非跨膜蛋白,如图5所示。
图5 NS1跨膜结构分析
Fig.5 Transmembrane structure analysis of NS1
2.4 蛋白翻译后修饰分析
利用NetPhos-3.1软件分析NS1磷酸化位点,结果显示,NS1含有15个磷酸化位点,其中9个S磷酸化位点,6个T磷酸化位点,见图6和表2。利用NetNGlyc-1.0软件分析N糖基化位点,结果显示,NS1含有N133糖基化位点,见图7和表2。利用NetOGlyc-4.0软件分析O糖基化位点,结果显示NS1含有S206 O糖基化位点。利用GPS-lipid软件分析棕榈酰化、N-肉豆蔻酰化、法尼基化和香叶基香叶酰化4种脂酰化位点,结果显示NS1没有这4种脂酰化位点,见表2。
表2 NS1翻译后修饰分析
Table 2 Post-translational modifications analysis of NS1
蛋白修饰位点名称 | 氨基酸位点 |
磷酸化位点 | T5、S42、S48、T49、T58、S73、T80、S87、T94、S99、S114、T151、S161、S205、S206、 |
N糖基化位点 | N133 |
O糖基化位点 | S206 |
棕榈酰化 | — |
N-肉豆蔻酰化 | — |
法尼基化 | — |
香叶基香叶酰化 | — |
注: “—”代表没有修饰位点
2.5 NS1三级结构和配体结合位点分析
利用HelixFold3软件构建NS1的三级结构,如图8所示。利用PrankWeb软件分析NS1三级结构配体结合位点,结果显示NS1的C13、WH16-17、Q40、LR43-44、IE54-55、T58位氨基酸是配体结合位点,如图9所示。
图6 NS1磷酸化位点分析
Fig.6 Phosphorylation sites analysis of NS1
图7 NS1 N糖基化位点分析
Fig.7 N-glycosylation sites analysis of NS1
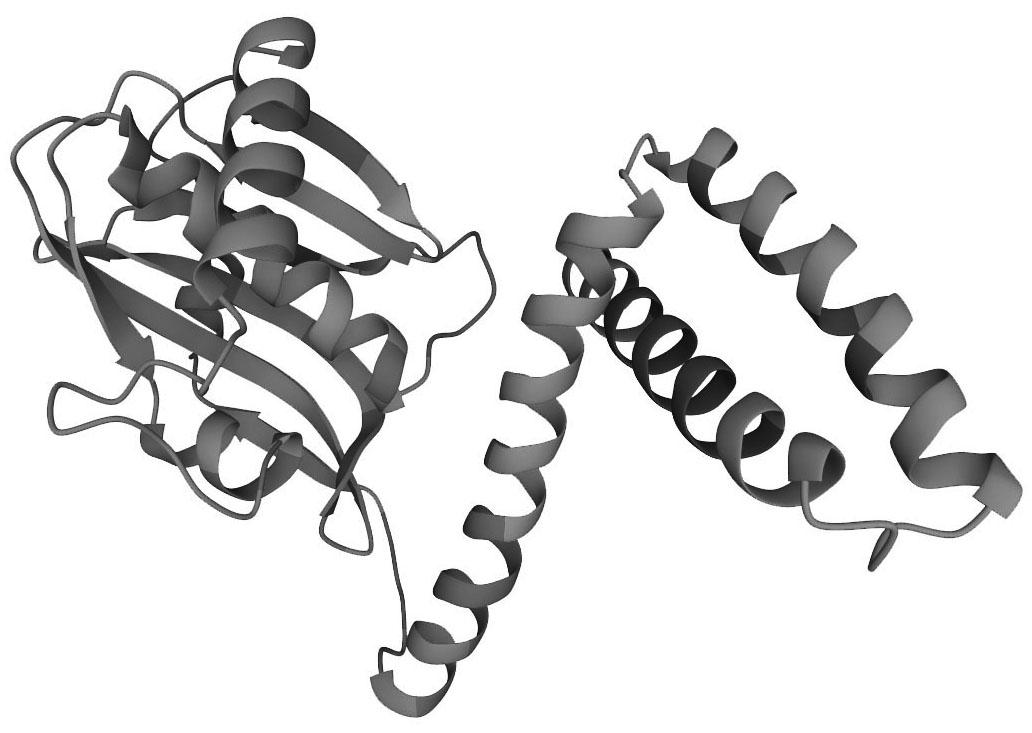
图8 NS1三级结构分析
Fig.8 The tertiary structure analysis of NS1
2.6 NS1结合基因组RNA分析
NS1的1~73位氨基酸能够结合病毒基因组,促进流感病毒复制[8-9]。利用RPISeq软件进行A/Washington/240/2024的NS1 的1~73位氨基酸与NP基因组结合预测,结果显示NS1的1~73位氨基酸能够结合NP基因组。利用HelixFold3软件构建NS1的1~73位氨基酸三级结构、流感病毒NP基因组RNA三级结构,构建NS1的1~73位氨基酸和流感病毒NP基因组RNA的相互作用模型,如图10所示。

A: NS1的1~73位氨基酸三级结构; B: 流感病毒NP基因组RNA三级结构; C: NS1的1~73位氨基酸和流感病毒NP基因组RNA三级结构相互作用
图10 NS1与NP基因相互作用分析
Fig.10 Analysis of the interaction between NS1 and NP genes
2.7 抗原表位分析
利用NetMHCIIpan-4.0软件分析辅助性T细胞抗原表位,结果显示NS1含有6个辅助性T细胞抗原表位。利用NetCTL-1.2 Server软件分析NS1的杀伤性T细胞抗原表位,结果显示NS1含有3个杀伤性T细胞抗原表位。利用SVMTriP软件分析NS1的B细胞抗原表位,NS1含有8个B细胞抗原表位,其中135-150SVIFDRLETLILIRAF抗原性最强,见表3。
3 讨论
D1.1基因型禽流感病毒是H5N1亚型2.3.4.4b进化分支的一种新的变种,更容易感染哺乳动物,有极强的致病性,已经在候鸟中广泛分布,并能够感染奶牛和人[7]。目前对D1.1基因型禽流感病毒的研究报道较少。
本研究对D1.1基因型禽流感病毒NS基因和NS1的进化分析显示奶牛和人的NS基因和NS1亲缘性很高,特别是52条来源于人和奶牛的D1.1基因型禽流感病毒NS1仅有2条序列合计存在3个氨基酸位点的差异,说明感染奶牛和人的D1.1基因型禽流感病毒NS基因和NS1比较保守,可以作为诊断依据蛋白。D1.1基因型NS1含有230个氨基酸,是亲水性蛋白质,主要分布在细胞质。此外,NS1还含有2个核定位信号,能够分布在细胞核,参与抑制宿主mRNA的出核,参与H5N1禽流感的致病过程[10]。NS1能够分布在线粒体,通过抑制线粒体抗病毒信号蛋白(Mitochondrial Anti-Viral Signaling protein,MAVS)通路抑制Ⅰ型干扰素的产生,干扰宿主对禽流感病毒的免疫清除[11]。
流感病毒蛋白翻译后修饰对蛋白功能的调节至关重要[12]。本研究确定了NS1的磷酸化、N糖基化和O糖基化修饰位点,磷酸化位点可能通过改变蛋白的电荷状态和空间构象,N糖基化位点和O糖基化位点可能参与蛋白折叠、活性等过程,影响NS1的结构和生物学功能[13],可能影响NS1与哺乳动物的相互作用,为D1.1基因型流感病毒在哺乳动物细胞内的复制与致病提供分子支持。
本研究对NS1的三级结构和配体结合位点分析发现,NS1的C13、WH16-17、Q40、LR43-44、IE54-55、T58位氨基酸形成一个口袋结构,是NS1配体结合位点,为基于NS1结构的抗病毒药物设计提供了新的靶点。NS1的1~73位氨基酸是RNA结合结构域,能够结合病毒基因组RNA促进病毒复制[8-9],本研究构建的NS1和病毒基因组RNA结合模型更直观展现了相互作用的细节,为深入研究NS1与病毒基因组RNA和宿主RNA的结合奠定基础,为研究D1.1基因型禽流感病毒更适合在哺乳动物细胞内复制提供基础数据。
NS1含有多个辅助性T细胞抗原表位、杀伤性T细胞抗原表位和B细胞抗原表位,说明NS1的免疫原性较强;其中NS1的135-150位氨基酸抗原性最强,可以作为D1.1禽流感检测和疫苗研发的参考序列;NS1含有的杀伤性T细胞抗原表位,可能引起宿主的细胞免疫,引发炎症反应[14-15]。
4 结论
综上所述,本研究通过对D1.1基因型禽流感病毒NS1理化性质、结构和功能的生物信息学分析,对进一步了解D1.1基因型禽流感病毒NS1的生物学功能具有重要意义,为深入了解H5N1禽流感病毒D1.1基因型的病毒特点提供基础,以及为抗病毒药物的研发提供参考数据。
参考文献
[1] Al-Eitan L, Khair I, Shakhatreh Z, et al. Epidemiology, biosafety, and biosecurity of Avian Influenza: Insights from the East Mediterranean region[J]. Reviews in Medical Virology, 2024, 34(4): e2559. DOI: 10. 1002/rmv. 2559.
[2] European Food Safety Authority, European Centre for Disease Prevention and Control, European Union Reference Laboratory for Avian Influenza, et al. Avian influenza overview September-December 2024[J]. Efsa Journa, 2025, 23(1): e9204. DOI: 10. 2903/j. efsa. 2025. 9204.
[3] 康宁, 谭毅. 人感染禽流感流行现状及疫苗研究进展[J]. 应用预防医学, 2021, 27(6): 569-574.
[4] European Food Safety Authority, European Centre for Disease Prevention and Control, European Union Reference Laboratory for Avian Influenza, et al. Avian influenza overview December 2021-March 2022[J]. Efsa Journa, 2022, 20(4): e07289. DOI: 10. 2903/j. efsa. 2022. 7289.
[5] Hu X, Saxena A, Magstadt D R, et al. Genomic characterization of highly pathogenic avian influenza A H5N1 virus newly emerged in dairy cattle[J]. Emerging Microbes & Infections, 2024, 13(1): 2380421. DOI: 10. 1080/22221751. 2024. 2380421.
[6] Baker AL, Arruda B, Palmer M V, et al. Dairy cows inoculated with highly pathogenic avian influenza virus H5N1[J]. Nature, 2025, 637(8047): 913-920.
[7] Kwon T, Trujillo J D, Carossino M, et al. Pathogenicity and transmissibility of bovine-derived HPAI H5N1 B3.13 virus in pigs. bioRxiv[J]. Emerging Microbes & Infections, 2025, 14(1): 2509742. DOI: 10.1080/22221751. 2025. 2509742.
[8] Robb NC, Chase G, Bier K, et al. The influenza A virus NS1 protein interacts with the nucleoprotein of viral ribonucleoprotein complexes[J]. Journal of Virology, 2011, 85(10): 5228-5231. DOI: 10. 1128/JVI.02562-10.
[9] Chien C Y, Tejero R, Huang Y, et al. A novel RNA-binding motif in influenza A virus non-structural protein 1[J]. Nature Structural Biology, 1997, 4(11): 891-895. DOI: 10. 1038/nsb1197-891.
[10] Qiu Y, Krug R M. The influenza virus NS1 protein is a poly(A)-binding protein that inhibits nuclear export of mRNAs containing poly(A)[J]. Journal of Virology, 1994, 68(4): 2425-2432. DOI: 10. 1128/JVI. 68. 4. 2425-2432. 1994.
[11] Tseng Y Y, Kuan C Y, Mibayashi M, et al. Interaction between NS1 and Cellular MAVS Contributes to NS1 Mitochondria Targeting[J]. Viruses, 2021, 13(10): 1909. DOI: 10. 3390/v13101909.
[12]单骄宇, 赵彬彬, 李斌, 等. 翻译后修饰在病毒感染过程中的作用[J]. 中国病毒病杂志, 2017, 7(1): 67-72.
[13] Hutchinson E C, Denham E M, Thomas B, et al. Mapping the phosphoproteome of influenza A and B viruses by mass spectrometry[J]. PLOS Pathogens, 2012; 8(11): e1002993. DOI: 10. 1371/journal. ppat. 1002993.
[14] 孙佳善. 禽流感病毒NS1蛋白抗原表位不同筛选方法的差异分析[D]. 哈尔滨:东北农业大学, 2013.
[15]文雪霞, 包红梅, 孙佳善, 等. 抗禽流感病毒NS1蛋白单克隆抗体的制备及其抗原表位的初步分析[J]. 中国预防兽医学报, 2013, 35(5): 414-418.

图2 人和奶牛感染D1.1基因型禽流感病毒NS基因进化树分析
Fig.2 Phylogenetic tree analysis of NS gene from D1.1 genotype avian influenza viruses isolated from humans and dairy cows

图3 人和奶牛感染D1.1基因型禽流感病毒NS1进化树分析
Fig.3 Phylogenetic tree analysis of NS1 from D1.1 genotype avian influenza viruses isolated from humans and dairy cows
表1 NS1氨基酸组成情况
Table 1 Amino acid composition of NS1
氨基酸 | 数量 (个) | 占比 (%) | 氨基酸 | 数量 (个) | 占比 (%) |
Ala (A) | 15 | 6.5 | Arg (R) | 19 | 8.3 |
Asn (N) | 8 | 3.5 | Asp (D) | 16 | 7.0 |
Cys (C) | 2 | 0.9 | Gln (Q) | 9 | 3.9 |
Glu (E) | 18 | 7.8 | Gly (G) | 14 | 6.1 |
His (H) | 2 | 0.9 | Ile (I) | 16 | 7.0 |
Leu (L) | 23 | 10.0 | Lys (K) | 12 | 5.2 |
Met (M) | 9 | 3.9 | Phe (F) | 9 | 3.9 |
Pro (P) | 11 | 4.8 | Ser (S) | 16 | 7.0 |
Thr (T) | 13 | 5.7 | Trp (W) | 4 | 1.7 |
Tyr (Y) | 1 | 0.4 | Val (V) | 13 | 5.7 |
图4 NS1亲/疏水性分析
Fig.4 Hydrophilic/hydrophobic analysis of NS1
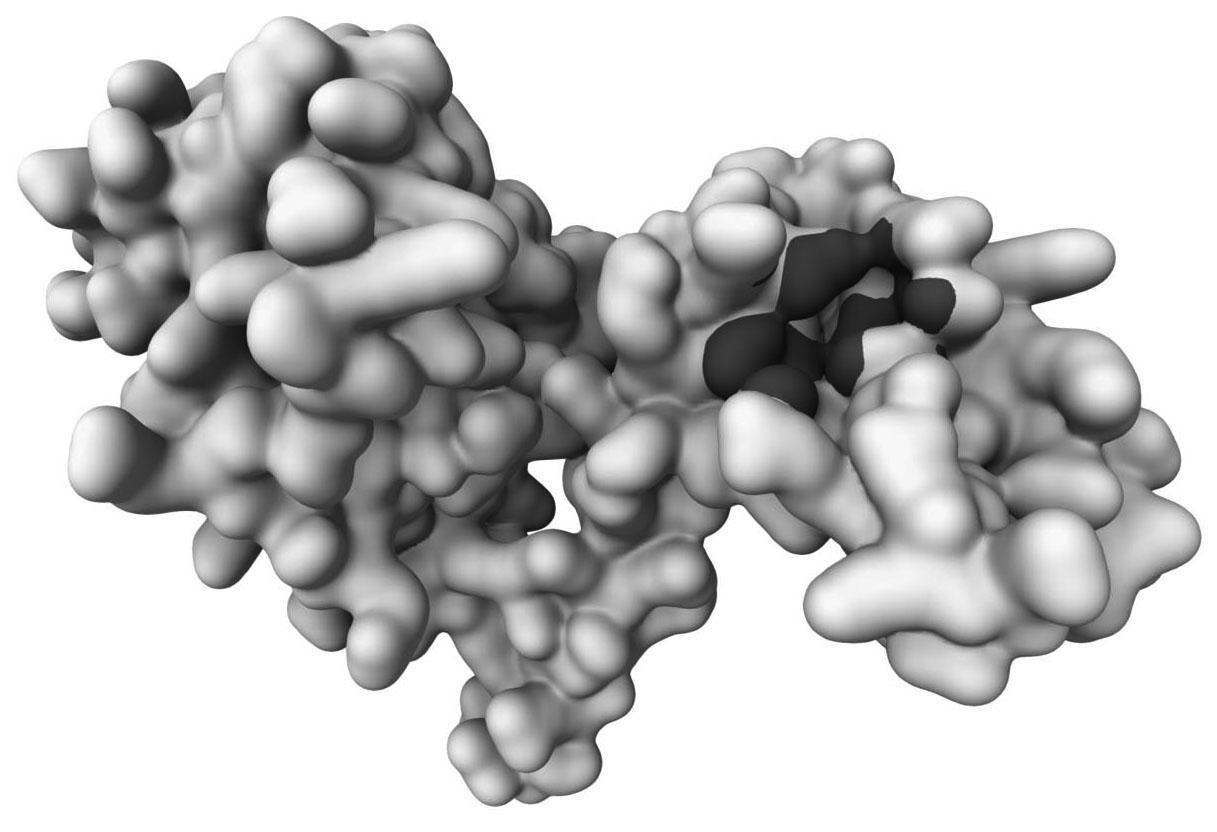
黑色代表配体结合位点
图9 NS1配体结合位点分析
Fig.9 Ligand-binding sites analysis of NS1
表3 NS1抗原表位分析
Table 3 Analysis of NS1 epitopes
名称 | NS1氨基酸位点 |
辅助性T细胞抗原表位 | 160-168ISPLPSLPG、174-182VKNAIGVLI |
杀伤性T细胞抗原表位 | 10-18QVDCFLWHV、90-98LTDMTLEE、123-131IMDKNIILK |
B细胞抗原表位 | 169-184HTNEDVKNAIGVLIGG、190-205NTVQVSETLQRFAWRS |







